你还在为找不到课题方案而焦虑吗,复现一篇论文吧!
你还在为找不到研究思路而苦恼吗,复现一篇论文吧!
你还在为找不到数据方法而悔恨吗,复现一篇论文吧
预计阅读时间4分钟
。。。。。
刚来的小伙伴不要慌了哈,小助理选的一直是同一篇论文
只不过用的是不同方法进行复现
一千种方法练一次
不如一种方法练一千次
进步的第一步就是敲下文章里面的代码
。。。。
以下就是复现过程,跟着文章走,他山之石可以攻玉,没准儿一不小心你就会了呢?
论文:
文章目录

- 1.STRING数据库
- 2.PPI网络构建
- 2.1 输入差异基因list
- 2.2 PPI图
- 2.3 保存结果
- 3.cytoscape软件筛选hub基因、功能模块
- 3.1 输入“string_interactions”文件
- 3.2 用cytohHubba插件,筛选top10 Hub基因
- 3.3 生存分析
- 3.4 用MCODE插件,筛选功能模块
。。。。。。
1.STRING数据库简介
string(search tool for the retrival of interacting genes/proteins)
基因、蛋白质相互作用关系检索工具
网址:string-db.org
简单来说:
String数据库通过文献内容管理,
来提取实验数据得出的蛋白质-蛋白质相互作用关系(PPI)。
它能够帮助用户轻松获取独特的,覆盖范围广的实验以及预测的相互作用关系信息。
string提供的相互作用关系主要基于confidence score(可靠指数),以及其他附属信息,比如提供蛋白质域和3D结构 。
- string目前的9.1版本,包括1100+个物种的5200+万蛋白质
- 联合开发的机构包括:CPR,EMBL,SIB,KU,TUD,UZH
。。
大背景:我们为什么要寻找蛋白质互作关系?
因为只有正确地发现和注释细胞中的所有功能性的相互作用关系,才能对细胞的功能进行系统层面的学习和理解。大家在收集和展现蛋白质相互作用的信息上,一直在努力地跟上相互作用关系探索的步伐。
近年来,无论是在实验观测和计算机预测技术都得到了显著的进步。但是蛋白质相互作用的信息比较容易出错,而且相当大的工作量来进行注释。
。。
String优势
1.string数据库完全是预先计算好的,无论是在高层次的网络中,还是单个相互作业关系记录的界面,所有的信息都可以被迅速获取。
2.它还支持单独选择各种证据类型,这样能够在运行的时候进行定制的搜索,同时也会有专门的查看器来对所有的关联证据进行查看。
3.该数据库是一项探索性的资源:它比基本的相互作用关系数据库包含了更大的关联数据--尽管是有不同的可能值。
4.因此,它最好被用于快速、初步地获取要查询的蛋白质的功能合作伙伴,尤其是对那种还没能很好的表征的蛋白质。
。。。。。。。。
2.PPI网络构建
2.1 输入差异基因list
进入网页,左侧选择“Multiple proteins”,输入所有差异基因list,“Organsim”选择“Homo sapiens”,然后“SERACH”
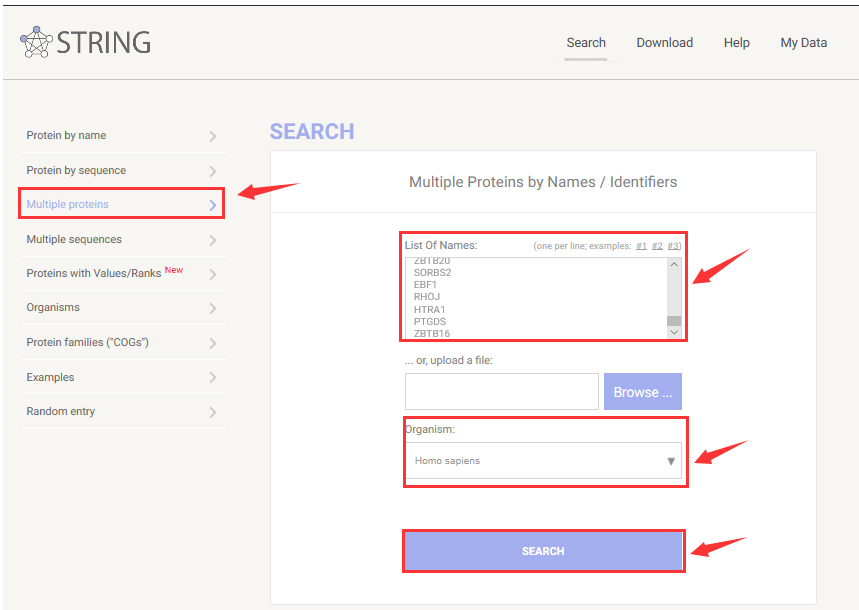
继续
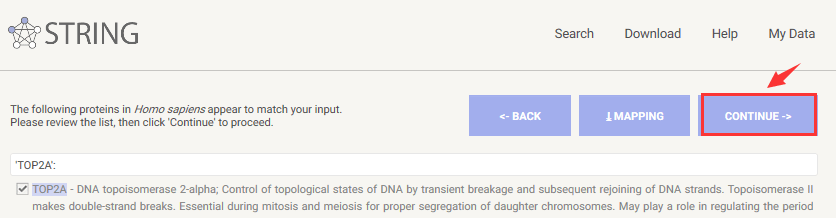
2.2 PPI图
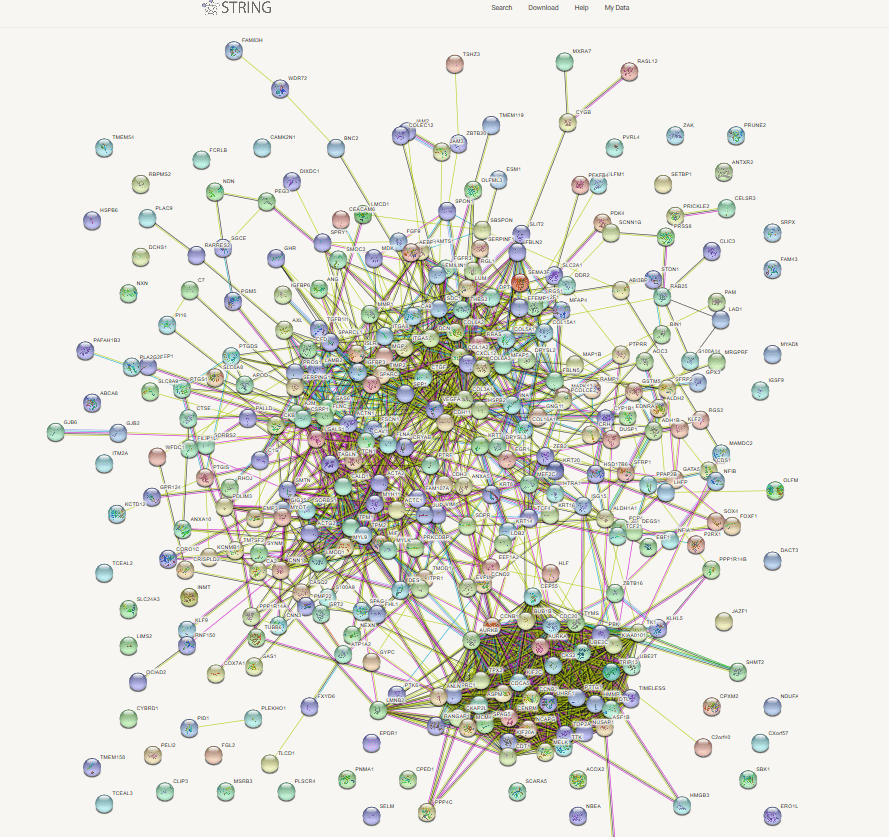
2.3 保存结果
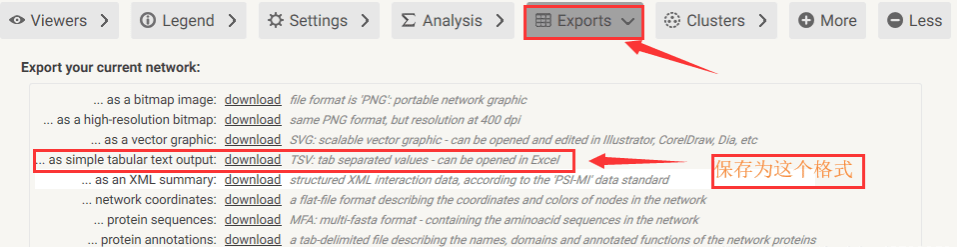
。。。。。。。。
3. cytoscape软件筛选hub基因、功能模块
在此之前,需要先下载cytohHubba插件以及MCODE插件
3.1 输入“string_interactions”文件
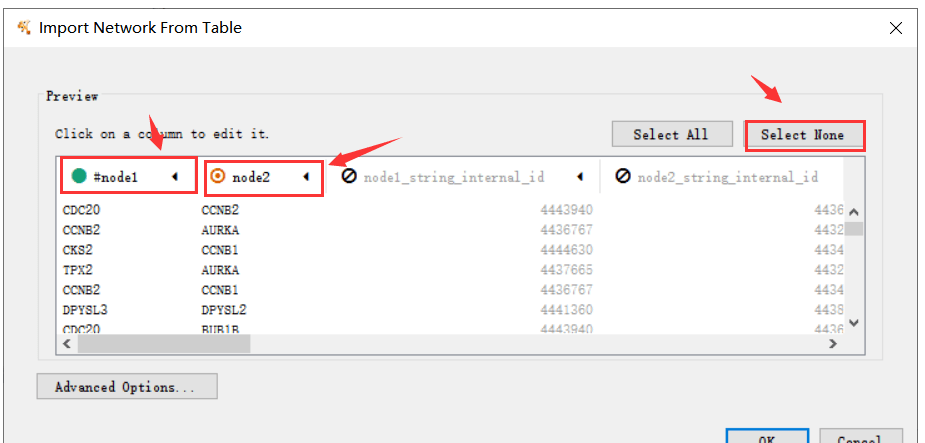
3.2 用cytohHubba插件,筛选top10 Hub基因
下面这张图主要就是根据Degree值给基因排序
- End -
- 本文固定链接: https://www.maimengkong.com/kyjc/1050.html
- 转载请注明: : 萌小白 2022年6月28日 于 卖萌控的博客 发表
- 百度已收录
