一、引言
肝脏作为人体代谢的核心器官,其转录组具有高度的复杂性和动态性。一个基因可产生多种不同转录本,且每种转录本均可能携带独特的转录后修饰,这些特性均深刻影响着肝脏的生理功能与代谢调控。然而,人类肝脏组织难以直接接受实验干预,且现有转录组注释在转录本亚型及 RNA 修饰层面覆盖不足,尤其缺乏特定代谢条件下的针对性注释,这极大限制了我们对人类肝脏转录组动态变化的深入理解。
长期以来,研究者多依赖小鼠模型推断人类肝脏的代谢调控机制,但越来越多的证据表明,人与小鼠肝脏的基因调控存在显著差异,仅通过动物研究难以准确反映人类肝脏的真实生理状态。同时,基于患者样本的研究又因个体遗传背景和环境暴露的多样性,难以捕捉转录组对代谢刺激的整体响应图景。
为突破这一困境,本研究通过构建人源化肝脏小鼠模型,通过对其施加多样化的代谢刺激,并结合Nanopore Direct RNA sequencing等技术,首次建立了覆盖代谢响应的人类肝脏转录组从头注释,系统解析了转录本表达、m⁶A 修饰及 poly (A) 尾长度的动态变化,旨在揭示人类肝脏代谢调控的全新机制。
二、研究发现
1. 从头注释人类肝脏转录组揭示与病理生理相关的代谢响应
基于Nanopore DRS技术分析人源化肝脏,构建了人类和小鼠肝脏转录组的从头注释。分别与人类参考数据库(GENCODE v33)和小鼠参考数据库(GENCODE vM24)相比,
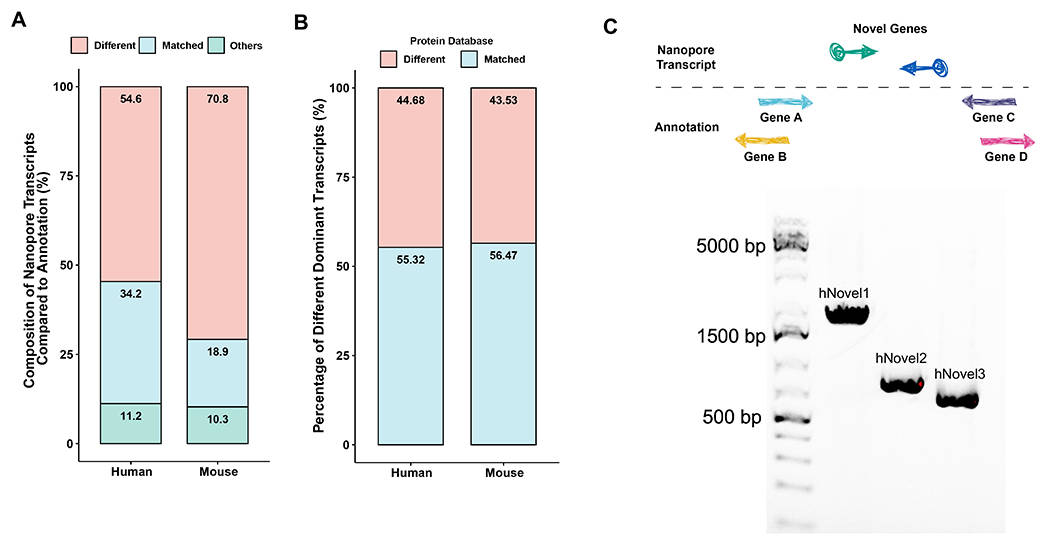
Nanopore DRS分别鉴定到54.6%和70.8%的新转录本(图1A)。这表明现有参考注释对肝脏转录本的覆盖存在显著不足,而Nanopore DRS技术能够更全面捕捉转录本的多样性。
对DRS鉴定的新转录本进行分析发现,其分别占人类和小鼠参考数据库中主要表达转录本的16.45%和18.17%。进一步比对显示,这些主要表达的新转录本中,有超过40%与参考数据库中的蛋白质编码序列不同(图1B),揭示这些新转录本的产生显著增加人类肝脏的蛋白质多样性,进而影响代谢功能。
同时,Nanopore DRS技术还在基因区间鉴定出212个新基因。为验证这些新基因的真实性,研究随机克隆其中一些新基因,并通过实验验证其序列与Nanopore DRS序列保持一致(图1C)。
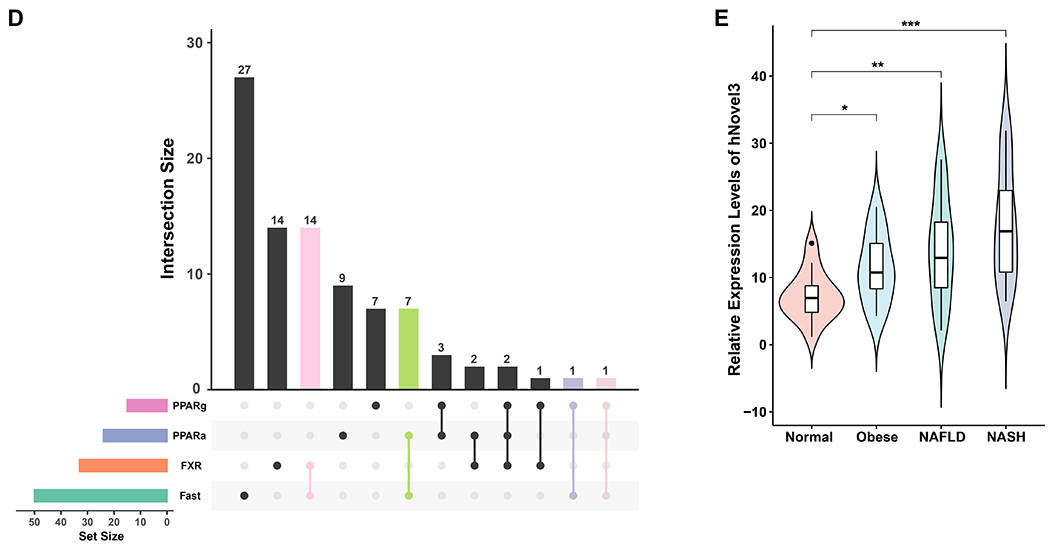
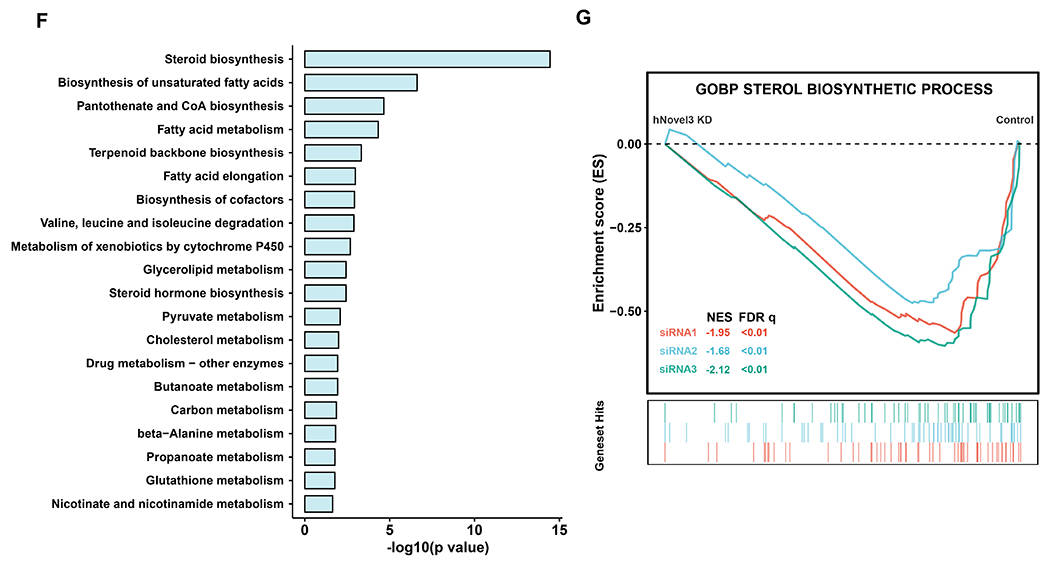
此外,研究通过分析不同代谢处理条件下对新基因的调控发现,超40%的新基因至少对一种代谢刺激有响应,部分新基因对多种刺激有响应(图1D)。例如以新基因hNovel3为例,它在FXR组中下调,而在NAFLD患者样本中表达上调(图1E)。进一步富集分析发现,其主要富集在固醇生物合成等代谢通路,暗示hNovel3可能通过调控该通路影响肝脏脂质代谢(图1F)。
最后,本研究为进一步验证hNovel3的生物学功能,在原代人类肝细胞中敲低 hNovel3 后,分析显示固醇生物合成通路显著下调,证实 hNovel3 对该通路的负调控作用,进一步支持其在肝脏代谢中的功能(图1G)。
2. 人类肝脏转录组在代表性代谢处理下的动态变化
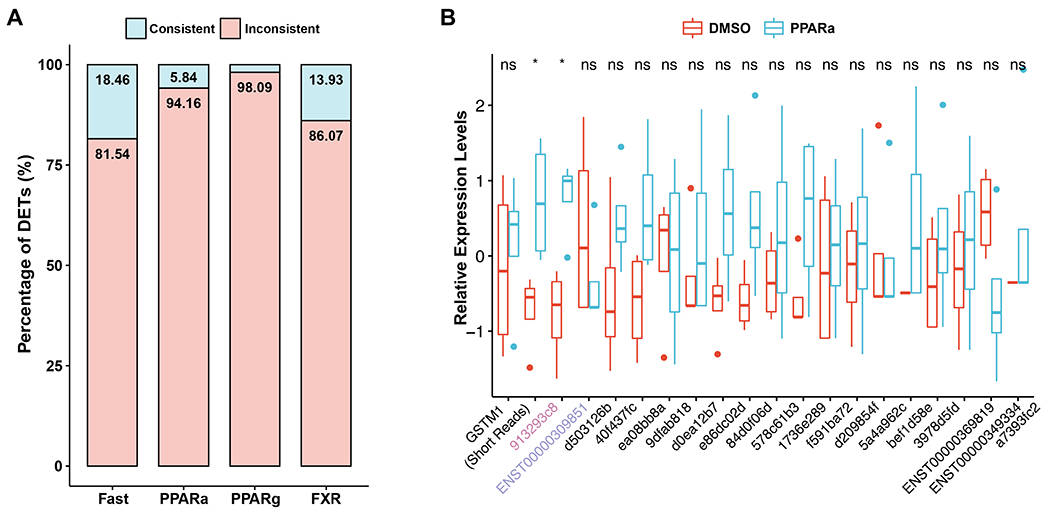
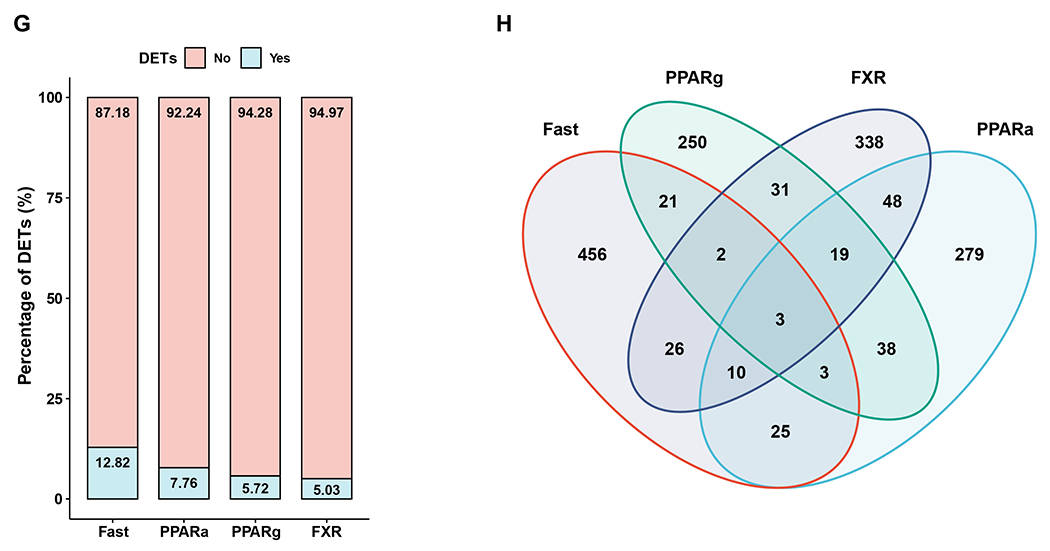
作者进一步研究在体内环境中,不同代谢刺激是如何在转录本水平上调控人类肝脏基因的表达。结果显示,在不同代谢处理下,大多数差异表达转录本与差异表达基因的变化方向不一致(图2A)。例如,PPARα 激动剂处理中,仅仅不到 6% 的差异表达转录本与差异表达基因的变化趋势一致。以谷胱甘肽转移酶Mu1(GSTM1)基因为例,其可产生20个转录本。在PPARα激动剂处理中,仅有2个转录本的表达水平发生显著变化,而其余转录本以及基因的表达水平均无明显变化(图2B)。这一结果直接体现了转录本水平调控与基因水平调控的分离,说明单个基因的不同转录本可能对代谢刺激产生差异化响应。
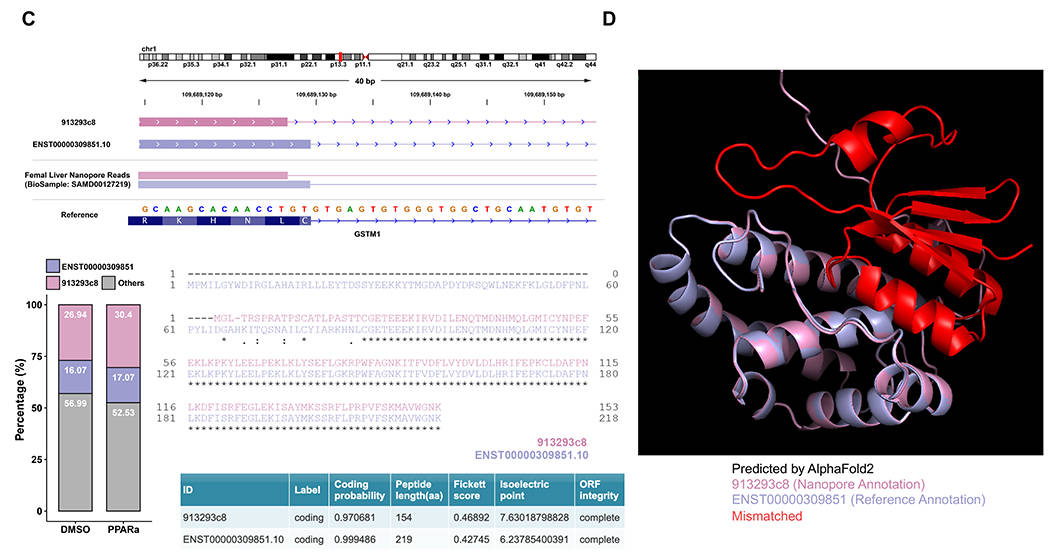
同时,GSTM1的差异转录本中,新转录本913293c8的表达量最高,且这一结果也得到Nanopore数据集(BioSample, SAMD00127219)的验证。与已知转录本ENST00000309851相比,该新转录本编码的蛋白质仅含 154 个氨基酸;蛋白序列比对显示两者存在明显差异,且编码潜力分析证实其具备蛋白质编码能力(图2C)。三维结构比对显示,GSTM1 的新转录本(913293c8)编码的蛋白比已知转录本(ENST00000309851)缺少一个大型结构域(图 2D)。这种结构差异可能导致其功能改变,提示转录本的多样性可能通过改变蛋白结构影响肝脏代谢功能。
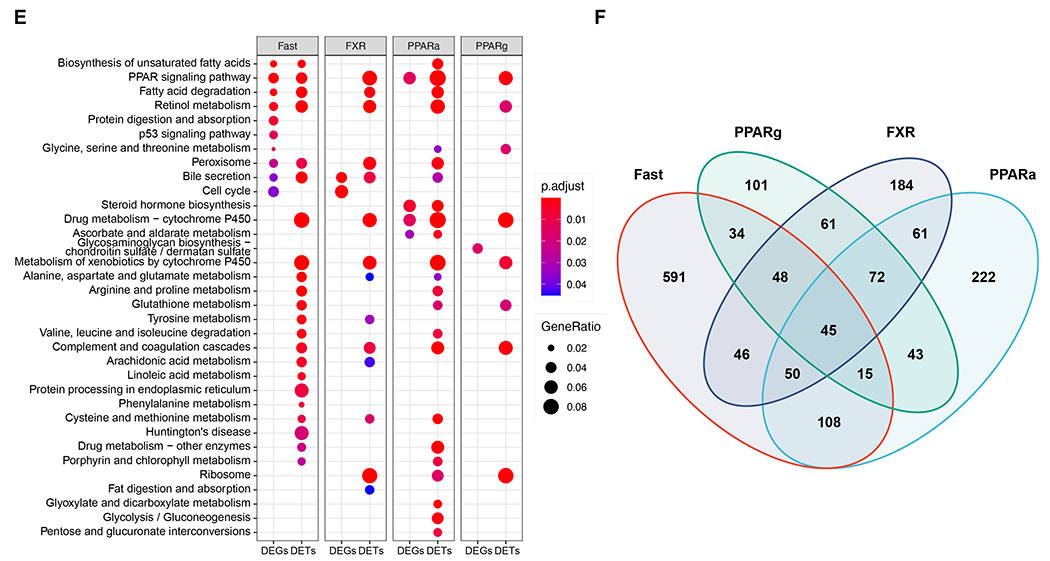
此外,差异表达转录本与差异表达基因富集的代谢通路也存在着显著差异(图2E)。这表明转录本水平的分析能捕捉到传统基因水平分析遗漏的关键代谢调控通路。分析进一步显示,相当一部分转录本受到至少两种代谢刺激的调控(图2F)。同时也检测到数百种转录本受到不同代谢刺激的特异性调控,并在不同的代谢途径中富集。
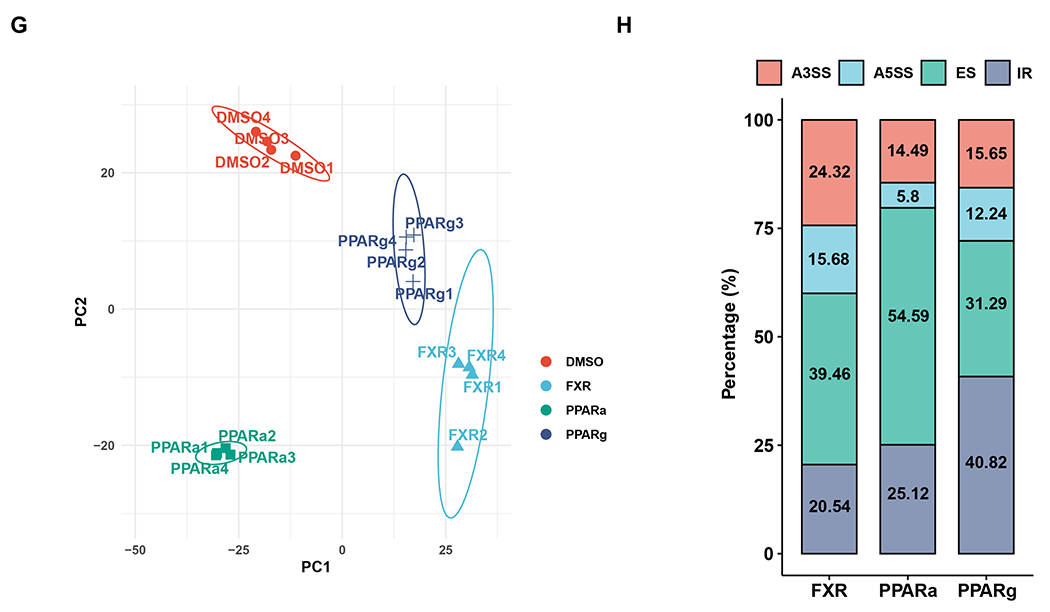
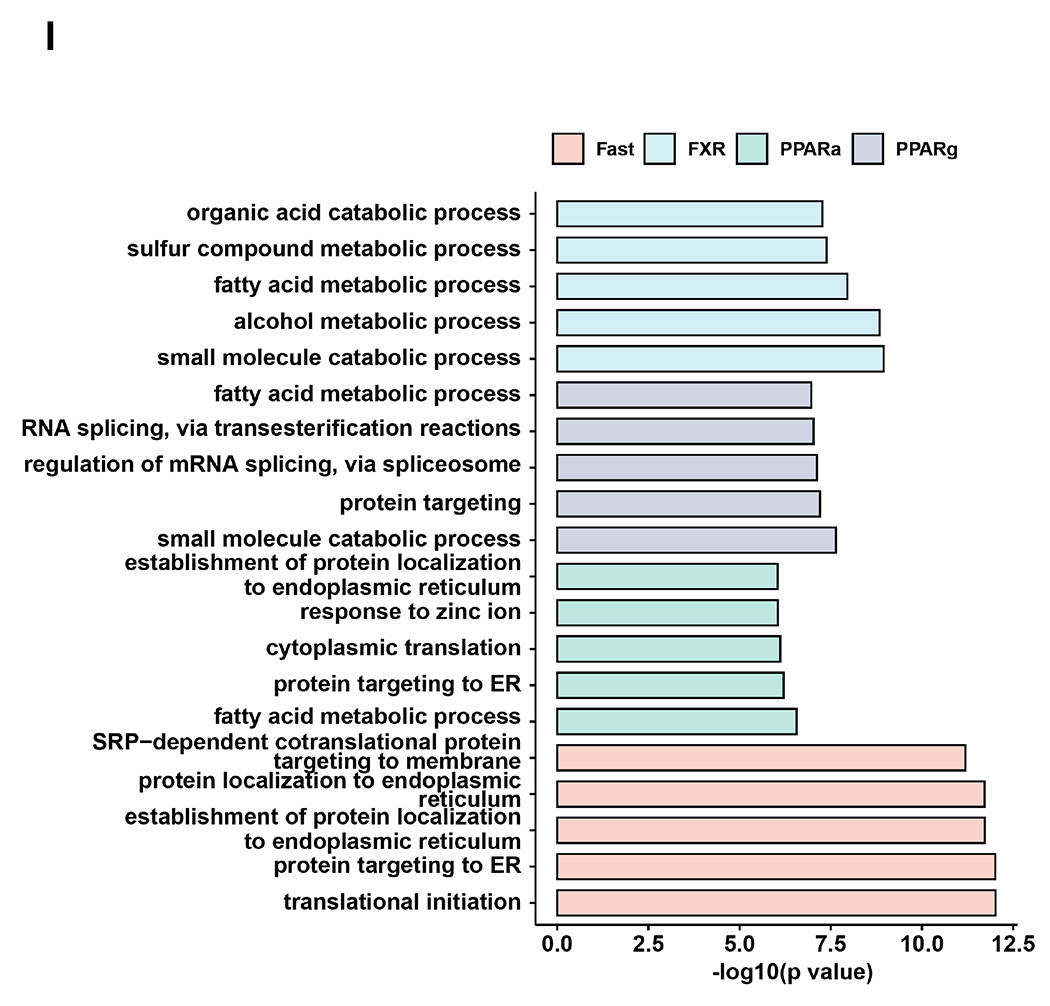
同样,Nanopore DRS能够可靠地识别剪接事件。PCA分析显示,不同代谢刺激的人源化肝脏样品的转录本和剪接事件明显分离(图2G)。不同代谢处理调控的可变剪接事件类型也存在差异,例如PPARα处理组中,外显子跳跃(ES)是主要的剪接事件;而PPARγ处理组中,内含子保留(IR)更为常见(图2H)。通路富集分析显示,显著变化的可变剪接事件参与重要代谢通路,且在不同处理中富集模式不同(图2I)。
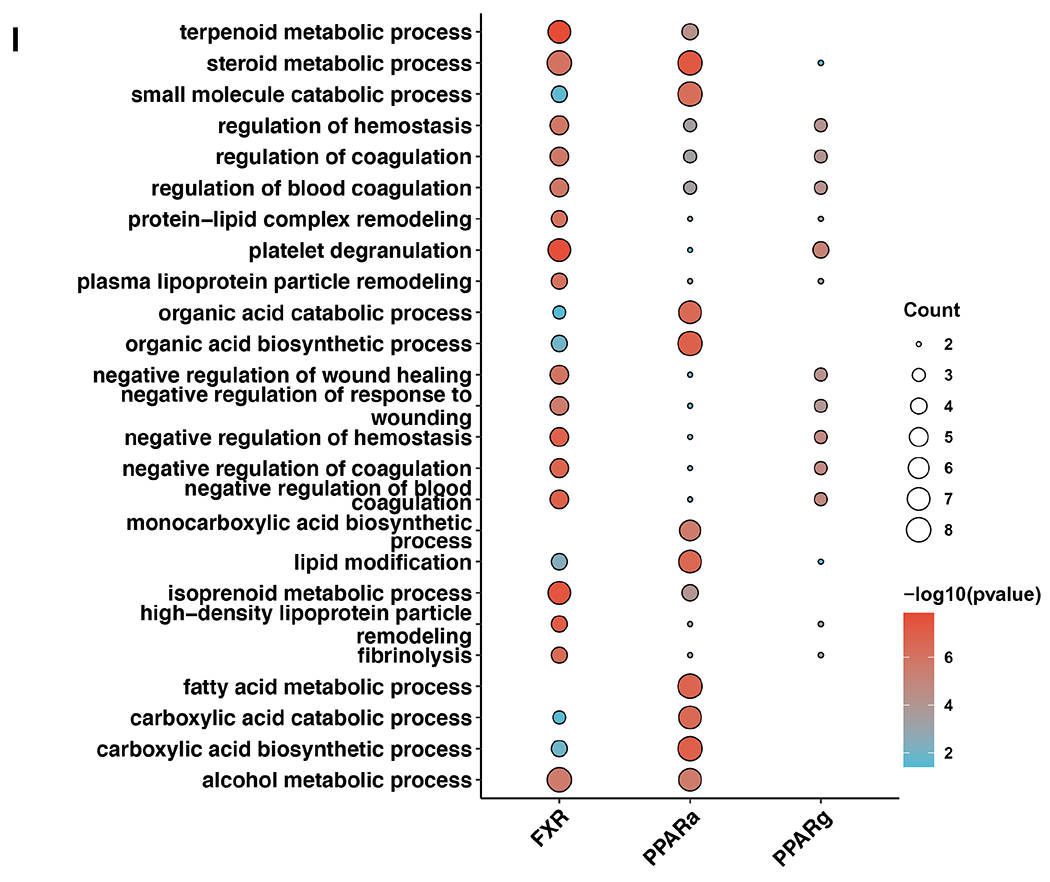
3. 人源化肝脏中RNA转录本的m6A修饰和poly(A)尾长度动态
Nanopore DRS数据分析显示,人源化肝脏中RNA的修饰位点主要富集于CDS和3’UTR区,且在CDS末端和3’UTR起始处形成明显峰值。PCA分析进一步证实,3’UTR区域的m6A修饰模式在不同处理间存在显著差异(图3A),而CDS区域的修饰模式则无明显区分。虽然大多数m6A位点在AL和Fast处理组中共享,但Fast组比AL组更偏向3’UTR区(图3B)。
通过差异修饰位点分析发现,超三分之一的位点包含GGACA motif,其中40%以上的差异修饰位点所在的转录本或基因存在表达水平变化(图3C),表明m6A修饰可以调控其表达水平。通路富集分析显示,差异m6A修饰的转录本主要来自脂肪酸代谢和酒精代谢等关键代谢通路的基因上(图3D)。
此外,Nanopore DRS技术可精确测定转录本的poly(A)尾长度。分析发现,同一基因的不同转录本在poly(A)尾长度分布上存在差异。进一步研究显示,不同的处理以背景依赖性的方式调控同一基因不同转录本poly(A)尾长的多样性(图3E)。例如,APOA2基因的两个主要转录本在所有处理组中表达水平均无显著变化,但它们的poly(A)尾长在PPARα和FXR处理组中差异变化显著(图3F)。同时,进一步比较差异表达转录本的poly(A)尾长发现,大多数差异表达转录本的poly(A)尾长没有差异(图3G)。另外,大多数差异表达转录本的poly(A)尾长呈现出特异性调节的特征(图3H)。通路富集分析表明,具有poly(A)尾长差异的转录本主要富集在关键的代谢途径上(图3I)。
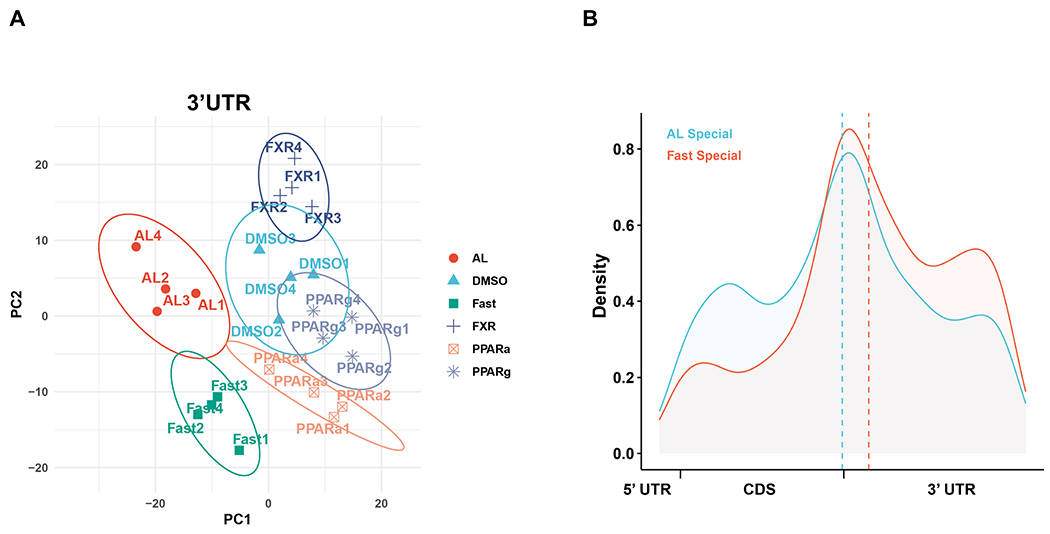
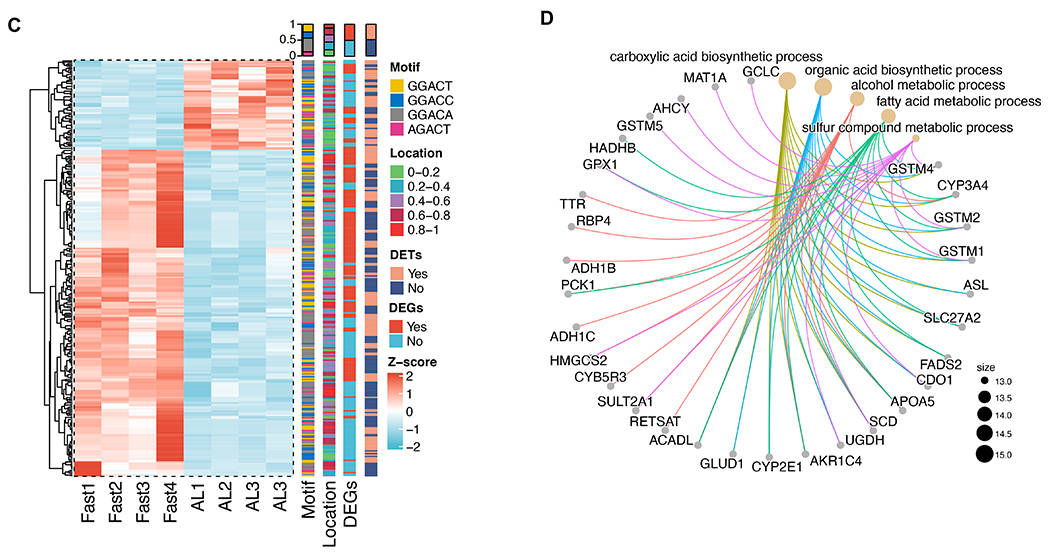
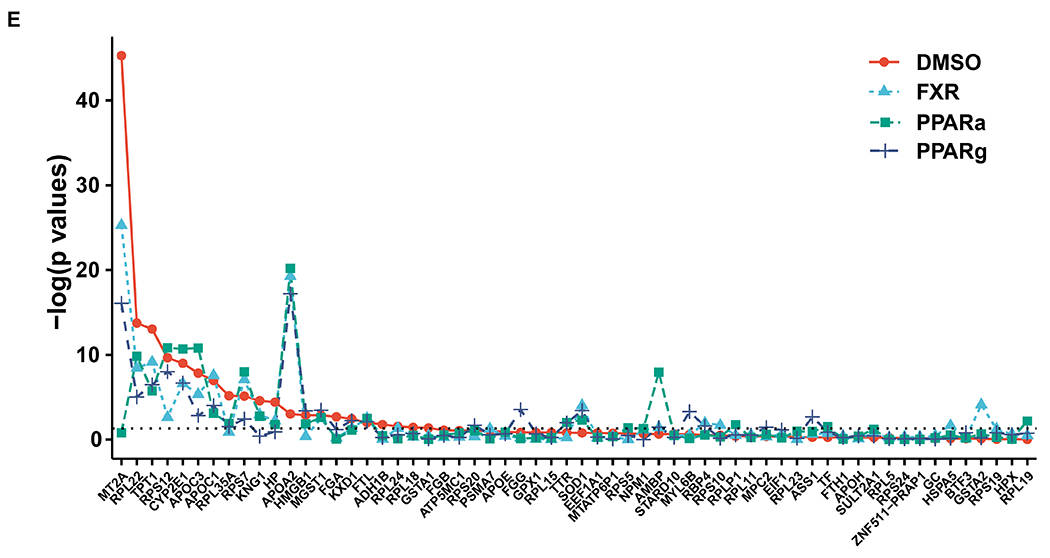
4. 不同遗传背景个体间转录组结构与动态的差异
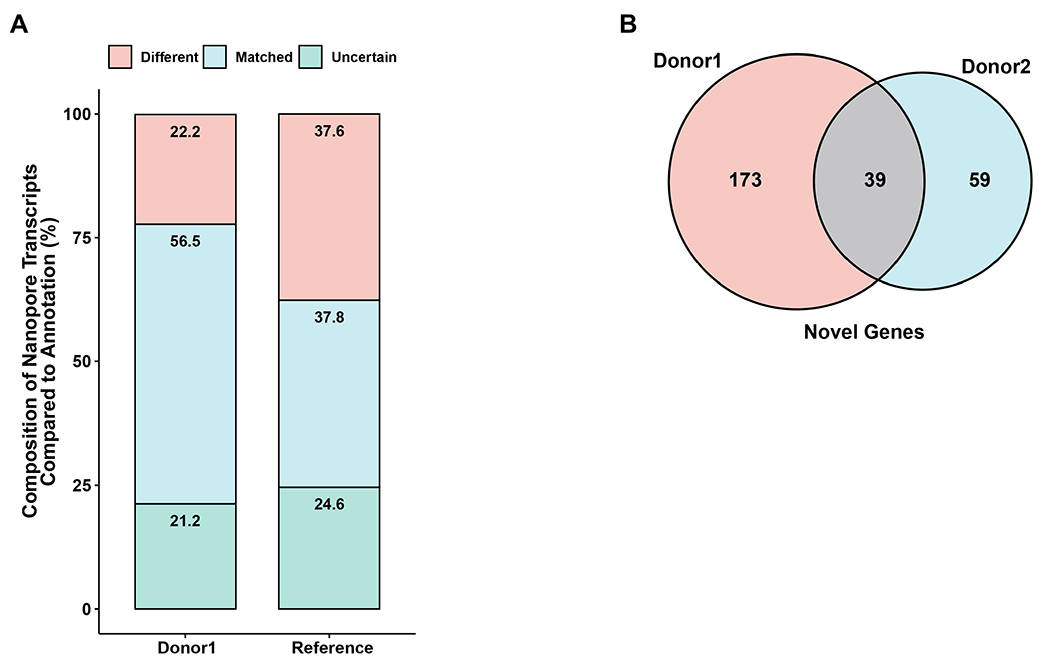
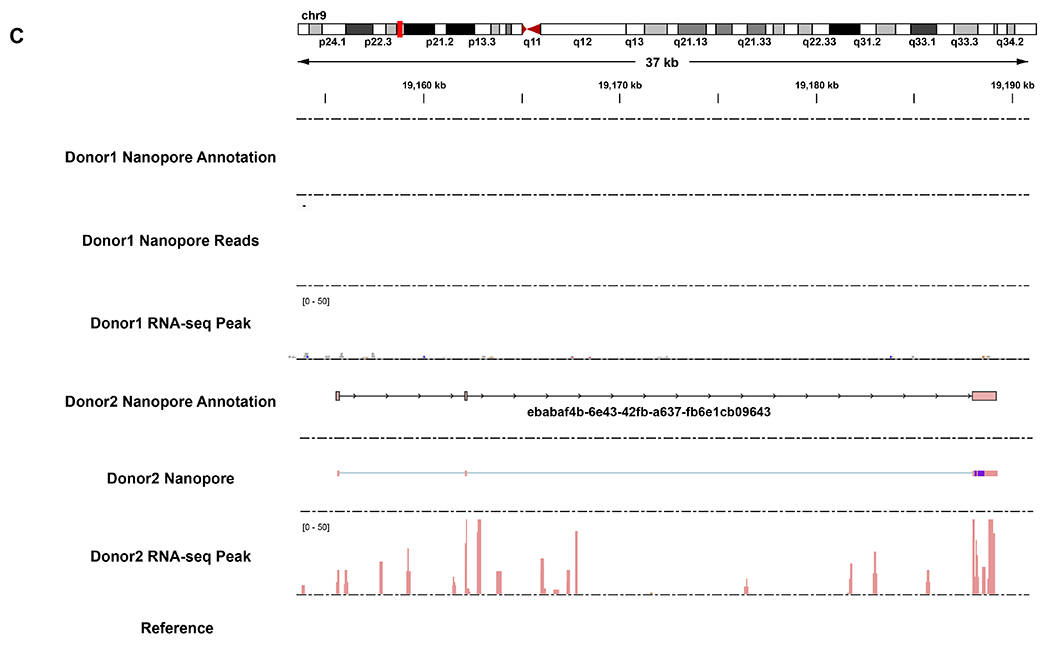
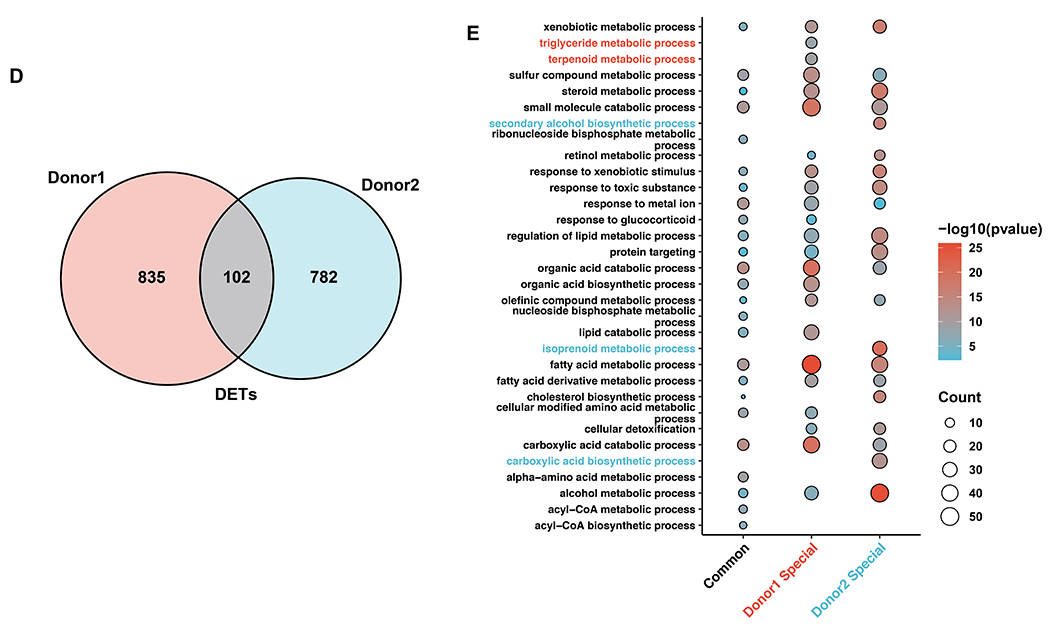
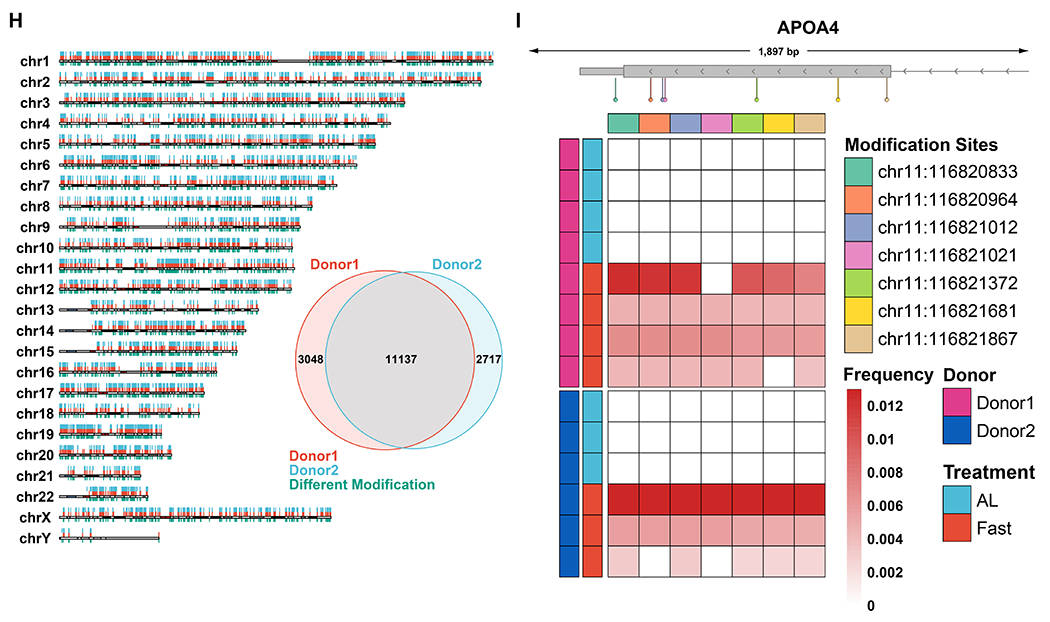
为了评估不同个体之间转录组的差异,本研究比较了供体2与供体1的数据,结果发现供体2中约55%的转录本与供体1匹配,且有约20%的转录本在两个供体中存在明确差异(图4A)。同时,在供体1中鉴定的新基因中,约40%的新基因在供体2中也表达(图4B)。例如,以新基因ebabaf4为例,其在供体2中被检测到全长序列,且短读长RNA-seq的峰值也证实该基因的存在,但在供体1中未检测到任何信号(图4C)。
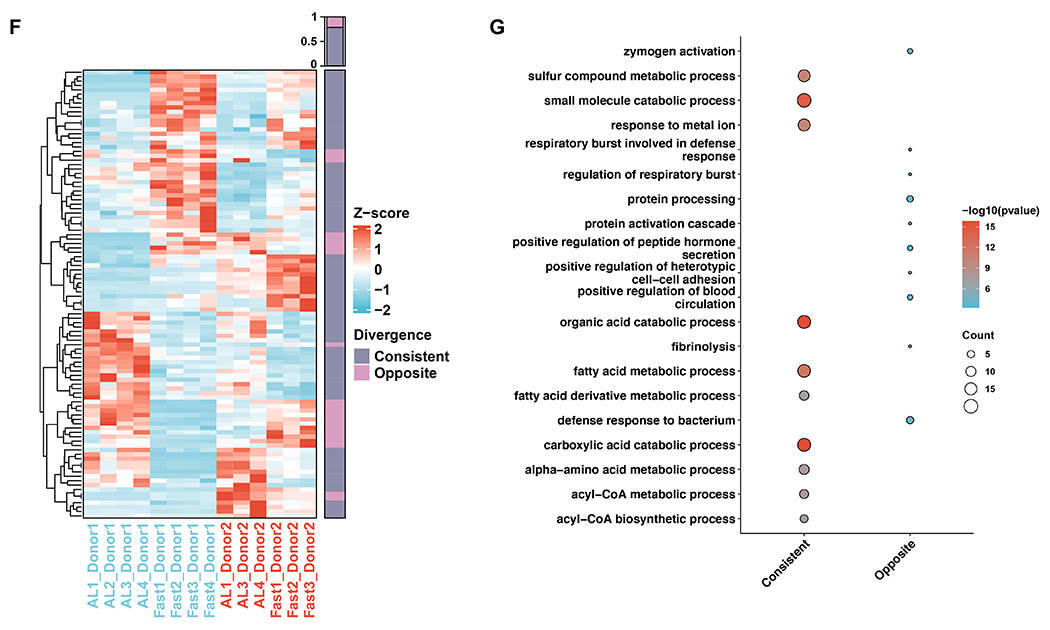
同时,分析发现两个供体中仅约10%的差异表达转录本变化趋势一致,其余90%为供体特异性调控(图4D)。基于供体特异性差异转录本的通路富集分析显示,供体1和供体2的富集通路模式存在明显差异(图4E)。比较AL和Fast处理组的2个供体的主要差异表达转录本发现,大多数转录本的表达趋势一致,但其中约20%的转录本呈现相反调控(图4F)。通路富集分析显示,两个供体中调控方向一致的转录本主要参与核心代谢通路;而调控方向相反的转录本则更多富集于炎症防御反应(图4G)。
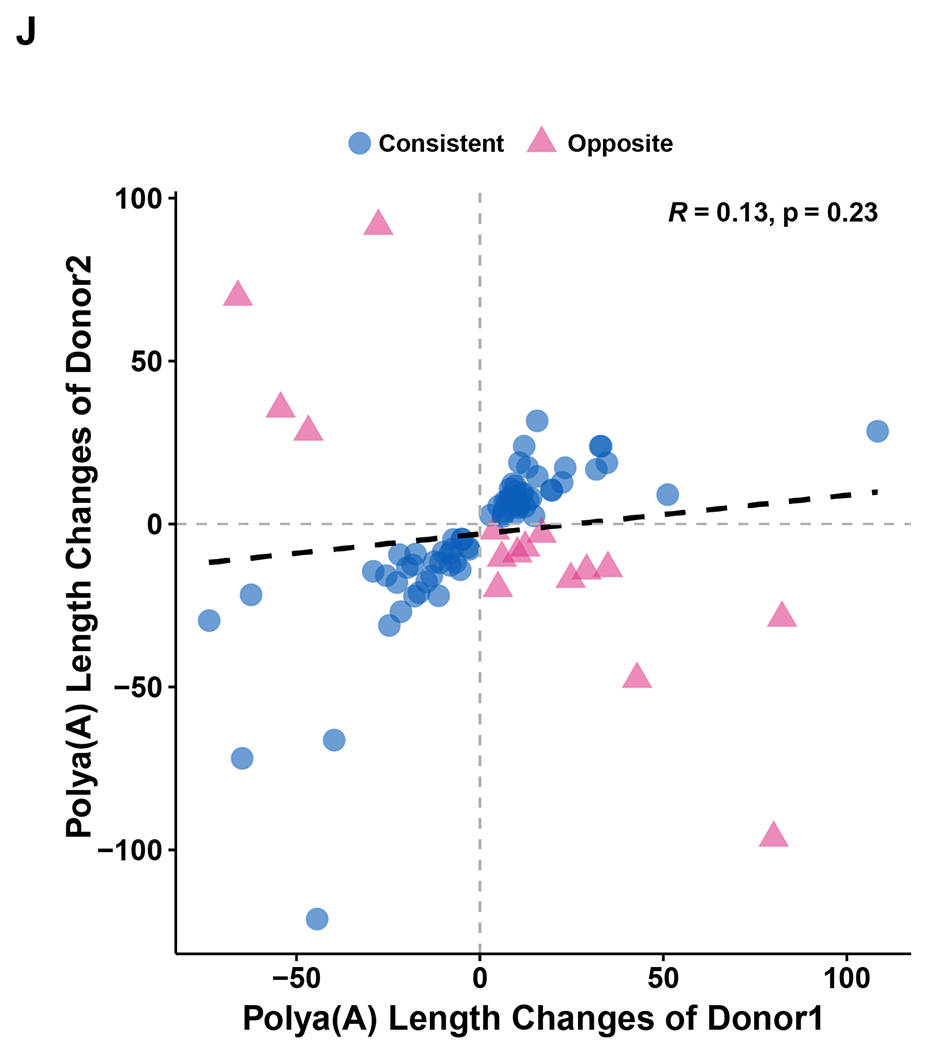
对比两个供体的m6A修饰位点发现,约80%的修饰位点在两者中共同存在,但仍有20%为供体特异性(图4H)。以 APOA4 基因为例,在AL处理条件下,其均无 m⁶A 修饰;而在Fast处理条件下,两个供体在该区域均诱导出 7 个 m⁶A 修饰位点(图 4I)。此外,两个供体之间poly(A)尾长度的变化模式也表现出相似性(图4J)。
三、总结
本研究以人源化肝脏小鼠为模型,结合Nanopore DRS 技术,构建人类肝脏转录组从头注释,发现大量新转录本,揭示转录组动态调控、RNA 修饰及 poly (A) 尾变化,还存在物种和个体差异,为肝脏代谢研究提供重要资源。主要结论归纳为以下四点:
1. 从头转录组注释:通过 DRS 构建的人类肝脏转录组注释中,54.6% 的转录本为新亚型,超 30% 新转录本仅在特定处理下表达;鉴定出 212 个基因间区新基因,部分与非酒精性脂肪肝相关。
2. 转录组动态调控:代谢处理中,绝大多数差异表达转录本无对应基因水平变化;可变剪接事件具有处理特异性,且富集通路与基因水平分析不同。
3. RNA 修饰与 poly (A) 尾动态:m⁶A 修饰富集于 CDS 和 3'UTR,代谢处理使其向 3'UTR 偏移,差异修饰位点多位于脂肪酸、酒精代谢相关基因;人类转录本 poly (A) 尾平均长约 100nt,lncRNA 的 poly (A) 尾显著长于 mRNA,且长度与表达水平负相关,其调控具有处理特异性。
4. 物种与个体差异:人与小鼠肝脏在转录本调控、m⁶A 修饰、poly (A) 尾长度等方面存在显著差异;不同遗传背景个体的转录本结构、代谢响应及 RNA 修饰存在约 20% 差异,基线转录组差异影响代谢刺激响应。
综上,Nanopore DRS技术为人类肝脏转录组研究提供了新资源,助力解析代谢相关疾病机制。其揭示的转录本、RNA 修饰动态等,可推动疾病标志物发现与精准疗法开发,也为其他人体器官基因调控研究提供范式。
参考文献
Jiang C, Li P, Ma Y, Yoneda N, Kawai K, Uehara S, Ohnishi Y, Suemizu H, Cao H. Comprehensive gene profiling of the metabolic landscape of humanized livers in mice. J Hepatol. 2024 Apr;80(4):622-633. doi: 10.1016/j.jhep.2023.11.020
转自:康测科技- 本文固定链接: https://maimengkong.com/kyjc/1979.html
- 转载请注明: : 萌小白 2025年7月23日 于 卖萌控的博客 发表
- 百度已收录
