一、研究背景
单细胞RNA测序(scRNA-seq)技术在解析细胞类型、状态和动态变化方面发挥着重要作用,但传统的短读长测序技术在全面表征RNA异构体方面存在不足。为克服限制,研究者们开发了单细胞靶向异构体长读长测序(scTaILoR-seq)技术,这是一种基于杂交捕获的方法,能够针对超过一千个感兴趣的基因进行靶向测序,显著提高了每个细胞中靶向转录本的中位数,为深入研究卵巢癌等疾病的转录组变异提供了有力工具。
二、研究内容及相关结论
(一)scTaILoR-seq技术的优化与性能评估
研究者们首先对scTaILoR-seq技术进行了优化,以提高长读长测序的效率和准确性。通过对比不同的文库制备方法,包括靶向、靶向+AM(artifact mitigation,伪影减少)和靶向+R2C2(rolling circle amplification,滚环扩增)策略,研究者们发现靶向+AM策略在提高完整读取比例和降低TSO-TSO(template switch oligo,模板转换寡核苷酸)伪影方面表现最佳(图1A)。具体而言,靶向+AM策略使完整读取比例增加了11.8%,同时显著减少了TSO-TSO伪影,与靶向策略相比,靶向+AM策略在保持较高读取通量的同时,提高了完整读取的比例(图1B)。
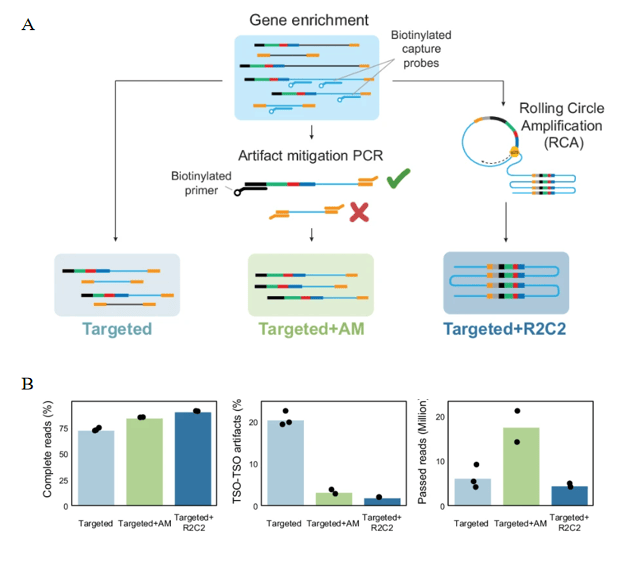 图1
图1
(二)单细胞水平上的转录组分析
研究者们进一步评估了scTaILoR-seq技术在单细胞水平上的转录组分析能力。通过对三种卵巢癌细胞系的混合样本进行分析,scTaILoR-seq技术能够有效区分不同的卵巢癌细胞系(图2A)。此外,scTaILoR-seq技术还能够检测到细胞类型特异性的可变剪接事件。例如,在PARP2基因中,研究者们发现了2号外显子上5’可变剪接位点的使用,其频率在三种细胞系中有所不同(图2B)。在RTKN基因中,研究者们发现了特定于SK-OV-3细胞系的可变5’-UTR和第一个外显子的使用事件(图2C)。这些结果表明,scTaILoR-seq技术能够在单细胞水平上揭示转录本的异质性和细胞类型特异性的转录本表达模式。
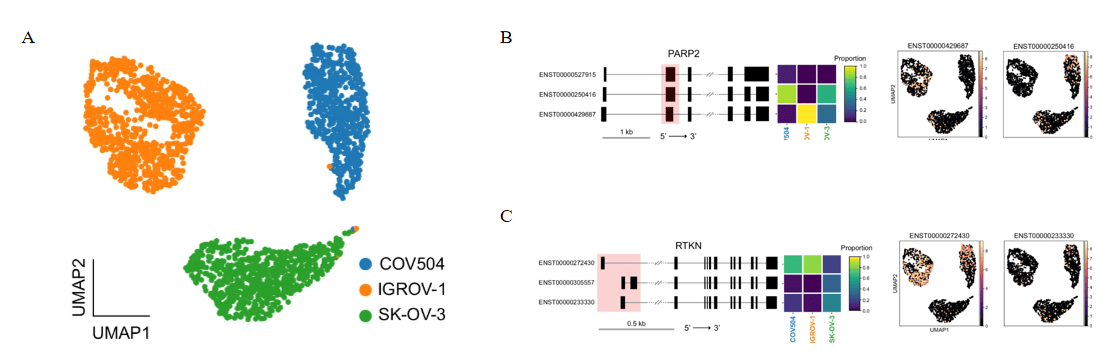 图2
图2
(三)卵巢癌肿瘤微环境的转录组景观
研究者们还利用scTaILoR-seq技术对卵巢癌患者的解离肿瘤细胞(DTCs)进行了分析,以探索卵巢癌肿瘤微环境(TME)中的转录组变异。通过对2243个基因进行靶向测序,研究者们检测到了8695个细胞,并识别了多种主要细胞类型,包括B细胞、T/NK细胞、髓系细胞、成纤维细胞和上皮细胞(图3A)。在这些细胞中,研究者们发现了细胞类型特异性的转录本表达模式,例如EPCAM在上皮细胞中的表达、COL3A1和COL1A2在成纤维细胞中的表达、CD3E和CD2在T细胞中的表达以及C1QB在髓系细胞中的表达(图3B)。此外,scTaILoR-seq技术还能够检测到细胞类型特异性的可变剪接事件。例如,研究者们发现了IL-32基因的两种不同异构体在CD8+ T细胞和PDGFRα-/β+成纤维细胞中的差异表达(图3C)。IL-32β异构体在所有细胞类型中占主导地位,而IL-32θ异构体在PDGFRα-/β+成纤维细胞中的表达显著降低。这些结果表明,scTaILoR-seq技术能够在卵巢癌肿瘤微环境中揭示细胞类型特异性的转录本表达和可变剪接事件。
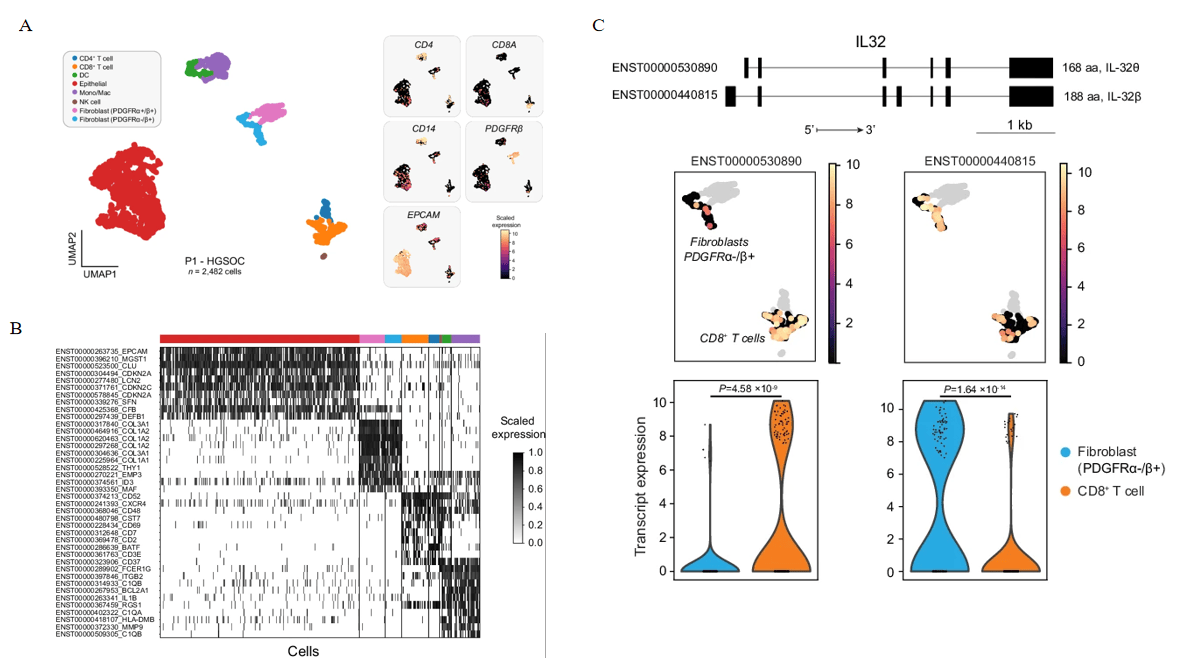 图3
图3
(四)T细胞受体(TCR)重建与肿瘤微环境中的T细胞克隆性
scTaILoR-seq技术还能够重建T细胞受体(TCR)α/β链配对,为研究肿瘤微环境中的T细胞克隆性提供了新的视角。通过对两名患者的DTCs进行分析,研究者们发现scTaILoR-seq技术能够成功重建TCRα/β链配对,配对率达到了49.7%(图4A)。在扩增的T细胞群体中,研究者们识别了15个高阶克隆型,其中最大的克隆型群体包含7个细胞,具有相同的CDR3序列(图4B)。这些结果表明,scTaILoR-seq技术能够在卵巢癌肿瘤微环境中揭示T细胞的克隆性,为研究T细胞与肿瘤抗原的相互作用提供了新的工具。
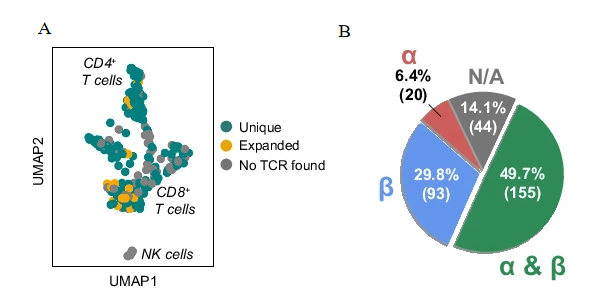 图4
图4
(五)单核苷酸变异(SNVs)的检测与转录本结构变异
scTaILoR-seq技术还能够检测到表达的单核苷酸变异(SNVs),并揭示这些变异与转录本结构变异之间的关联。通过对TP53基因的分析,研究者们发现了一个位于外显子7的SNV(chr17:7674241 G > C),该变异在肿瘤上皮细胞的一个亚群中特异性表达(图5A)。此外,研究者们还利用SpliceAI模型预测了与SNVs相关的隐匿性剪接事件,并通过评估参考碱基(REF)和替代碱基(ALT)匹配读取的覆盖度差异来识别转录本结构变异(图5B)。在82个预测的SNVs中,44个显示了覆盖率差异,表明REF和ALT等位基因之间的转录本结构存在差异(图5C)。这些结果表明,scTaILoR-seq技术能够在单细胞水平上揭示SNVs与转录本结构变异之间的关联,为理解癌症中的转录复杂性提供了新的视角。
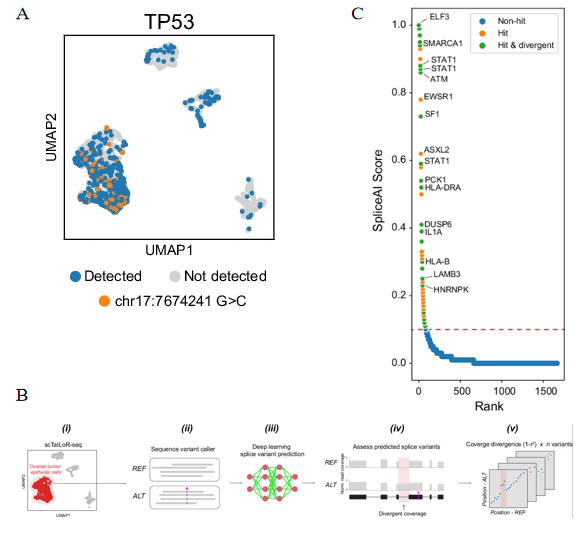 图5
图5
三、总结与展望
scTaILoR-seq技术通过靶向富集和伪影减少策略,显著提高了转录本检测的灵敏度,并能够在单细胞水平上揭示转录组变异、等位基因不平衡以及T细胞受体的多样性。这些发现不仅为卵巢癌的转录组研究提供了新的见解,也为其他复杂疾病的研究提供了新的技术手段。未来,随着单细胞全长RNA测序技术的进一步优化和应用,有望在更多疾病模型中实现更全面和深入的转录组分析,为疾病诊断、治疗和预后提供新的生物标志物和治疗靶点。
参考文献:
Byrne A, Le D, et al. Nature Communications (2024). DOI: 10.1038/s41467-024-51252-6.
- 本文固定链接: https://maimengkong.com/kyjc/1966.html
- 转载请注明: : 萌小白 2025年7月15日 于 卖萌控的博客 发表
- 百度已收录
