在Seurat的输出结果中,有一个展示表达量变化最大10个基因的图表令人印象深刻,如下图。关于该图表的具体内容参考之前 《单细胞转录组学习笔记之Seurat 3.0(一)》 一文。那么,常规散点图能不能画成这样效果?
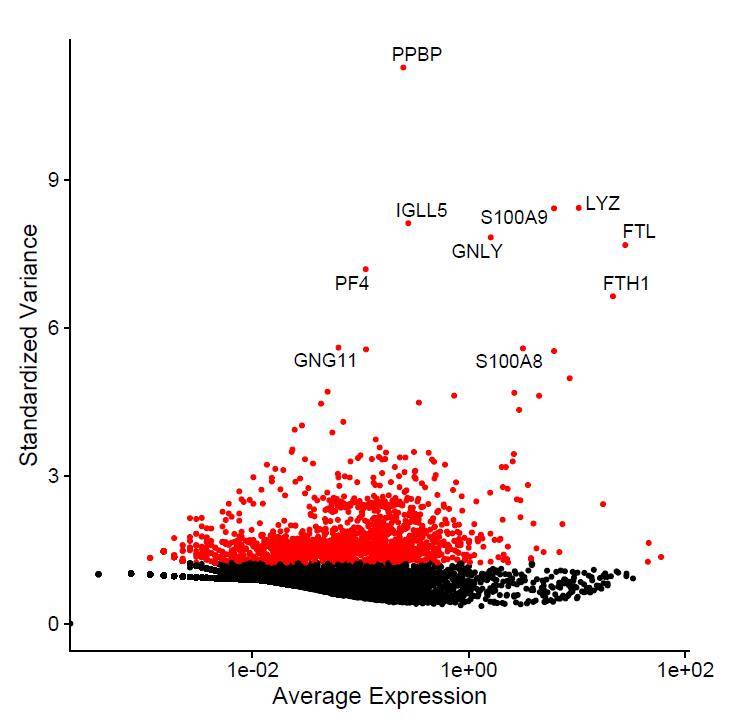
如果仍然使用针对单细胞数据高度定制的Seurat,显然是非常麻烦的。而使用ggplot2绘制类似这样的多标签图表,又容易出现“数据标签重叠”、超出绘图区域的标签“显示不全“和邻近数据点”指示不明“三大难题。
于是ggrepel这个R包应用而生,它让文字标签周围产生“力场”,相互排斥,相互靠近的点会添加“指引线”避免指示不明。
整个R包主要有两个用法几乎一样的函数geom_text_repel和geom_label_repel,可以把它们视作ggplot2中geom_text的加强版,很显然,Seurat集成了这个R包。下面就仍以上图的数据为例,一起看下具体如何使用ggrepel吧!
数据准备
这里的范例数据表格是直接从 《单细胞转录组学习笔记之Seurat 3.0(一)》 一文中Seurat对象中导出的。为了方便大家练习,本文范例数据已上传到OmicShare论坛,登陆后下载即可。
下载链接:
https://www.omicshare.com/forum/thread-6312-1-1.html
#读入数据:
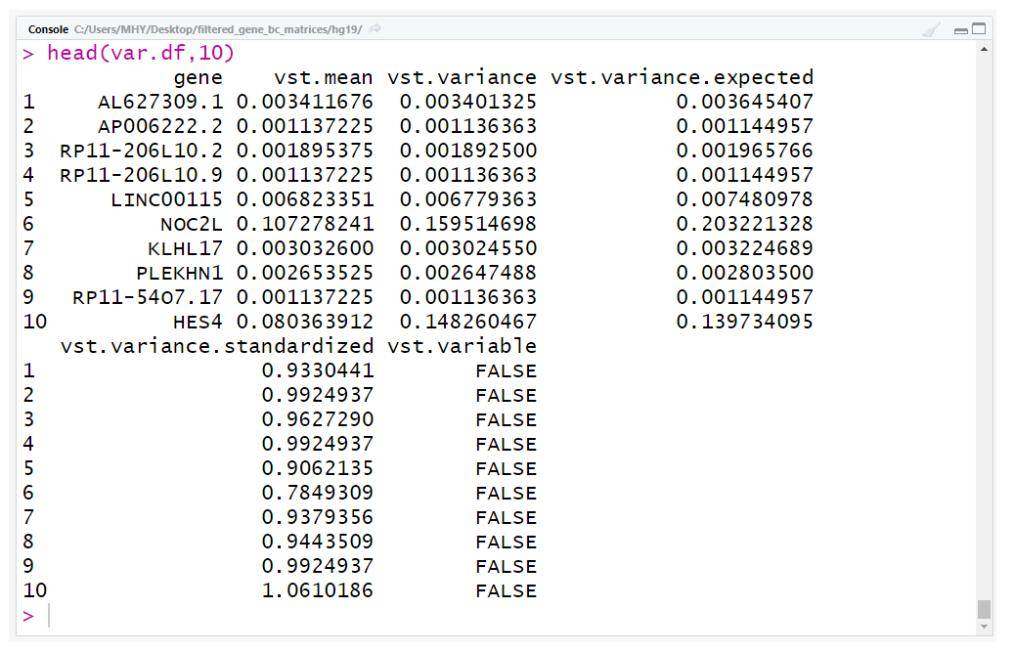
var.df <- read.table( "var.genes.xls",header = T,sep = "t")
head( var.df, 10)
#载入dplyr包;
library(dplyr)
#按照variance.standardized这一列数值降序排列;
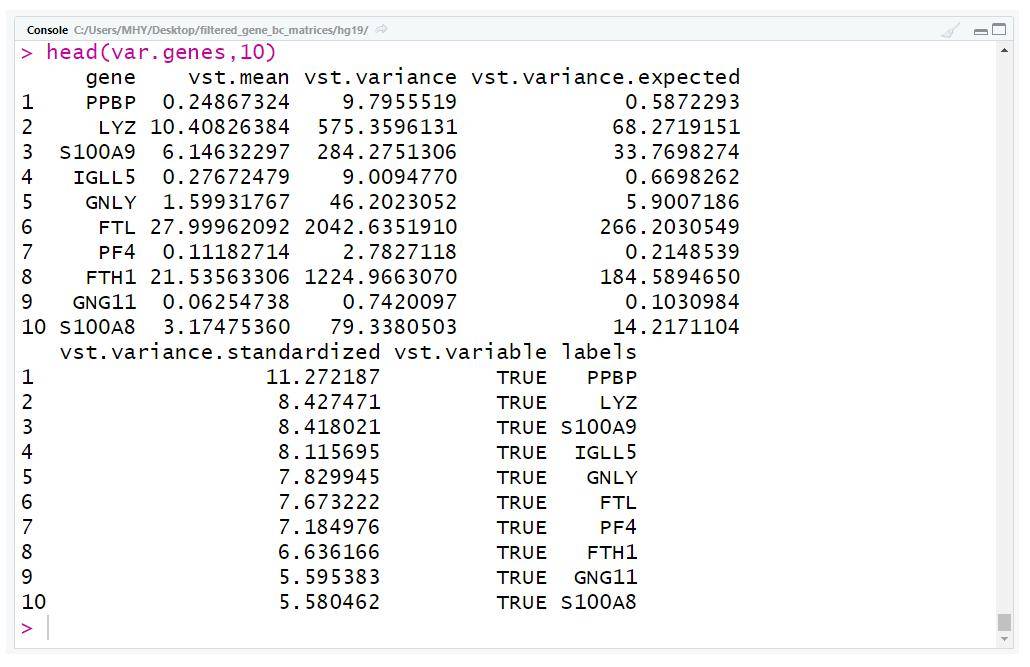
var.genes<-arrange( var.df,desc(vst.variance.standardized))
head( var.genes, 10)
为了检验这个包的性能,我这里选择绘制15个标签,当然选择展示20个也没问题。
#提取TopN基因集:
n <- 15
topn <- head( var.genes$gene,n)
topn
#追加标签列;
var.genes$labels <- ""
var.genes$labels[ 1:n] <- topn
head( var.genes, 10)
关于“散点”叠放细节调整,将数据改为升序排列,目的是先画非高变基因,关注Topn的基因最后再画,实现后者的散点置于“顶层”效果。
var.genes<-arrange( var.genes,vst.variance.standardized)
添加箭头
#载入所需的画图主力R包;
library(ggplot2)
library(ggrepel)
#常规的geom_text函数的效果;
p1 <- ggplot(data = var.genes,aes(log10(vst.mean),vst.variance.standardized,
color=vst.variable,label=labels)) +
geom_point
p1+geom_text(color= "grey20",size= 3)
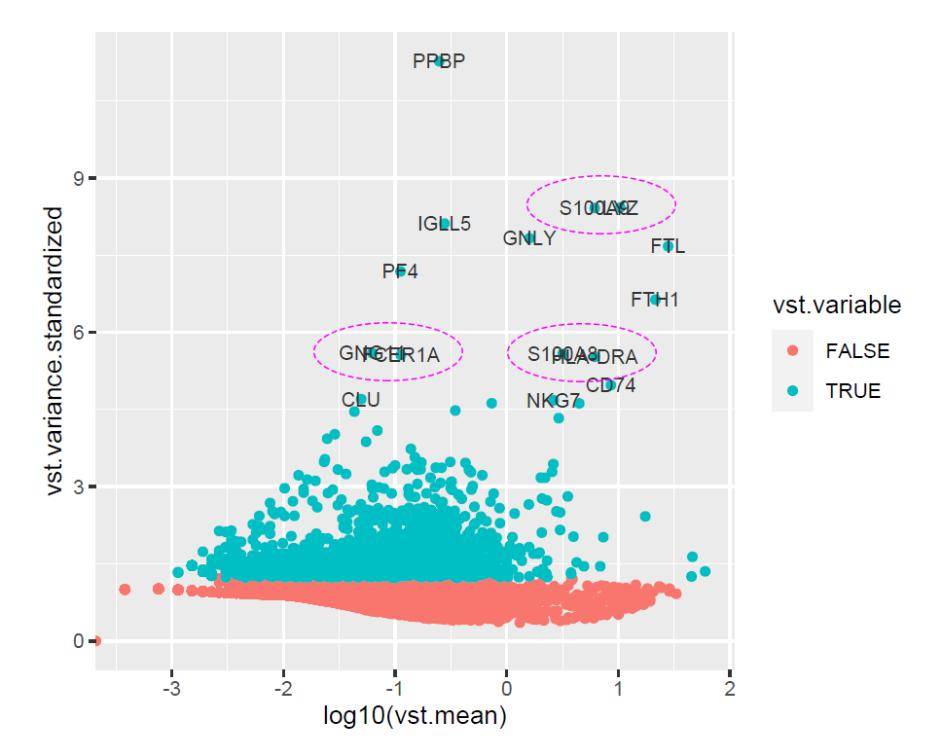
从上图我们可以看出,如果直接使用ggplot2的geom_text函数会出现文本标签相互重叠的情况(虚线椭圆所示),也会出现标签对邻近的点指示不明的情况,如上图的CLU、NKG7基因。那么如何避免上述问题呢?我们先使用一下geom_text函数的加强版:geom_text_repel。
set.seed( 100)
p2 <- p1+geom_text_repel(color= "grey20",size= 3,point.padding = NA)
p2
geom_text_repel和 geom_label_repel的核心参数:
nudge_x/y:
数据点与相应数据标签的距离,例如1表示标签在点右/上的1个单位处,而-2.2表示标签在点左/下2.2个单位处;
direction:
标签分布方向,x表水平分布,y 表示垂直分布,both 表示随机分布;
segment.size:
指定线段的粗细;
point.padding:
表示点周围的空余区域,决定连接线端点到到数据点中心的距离,单位为line。
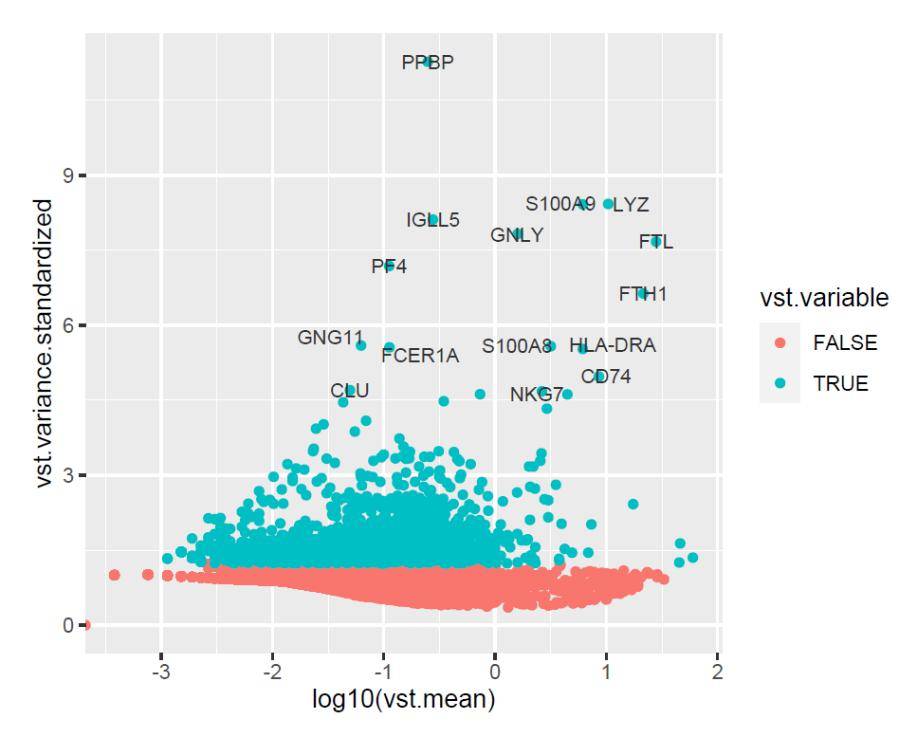
标签的“排斥”效果很不错,但是还不够“高大上”。接着,在垂直方向上给标签多一些偏移,并绘制箭头指引线。
p3 <- p1+geom_text_repel(force= 20,color= "grey20",size= 3,point.padding = 0.5,hjust = 0.5,
arrow = arrow(length = unit( 0.01, "npc"), type = "open", ends = "last"),
segment.color= "grey20",segment.size= 0.2,segment.alpha= 0.8,nudge_y= 1)
p3
关于为线段添加“箭头”,这里主要用到grid::arrow函数,注意,它的参数只适于调整“整支箭的箭头”部分,如下:
angle: 箭头的尖角的角度;
length: 箭头尖角的长度;
ends: "last", "first", "both", 指定线段的那端画箭头;
type: "open"和"closed" 指定箭头是否为封闭的三角形。
然后应用自定义主题,调整纵轴范围、散点颜色、图表主题等。
p3 + scale_y_continuous(breaks = c( 0, 3, 6, 9, 12),limits = c( 0, 12)) +
xlab( "Average expression") +
ylab( "Standardized variance") +
scale_color_manual(values=c( "#a2a9af", "#a8df65")) +
theme_classic+
theme(plot.margin = unit(c( 1, 1, 0.5, 0.5), "cm"),
legend.position = "none",
axis.title = element_text(size = rel( 1.2)),
axis.text = element_text(size=rel( 1)),
axis.line = element_line(size = 0.5, colour = "grey30"))
效果如下:
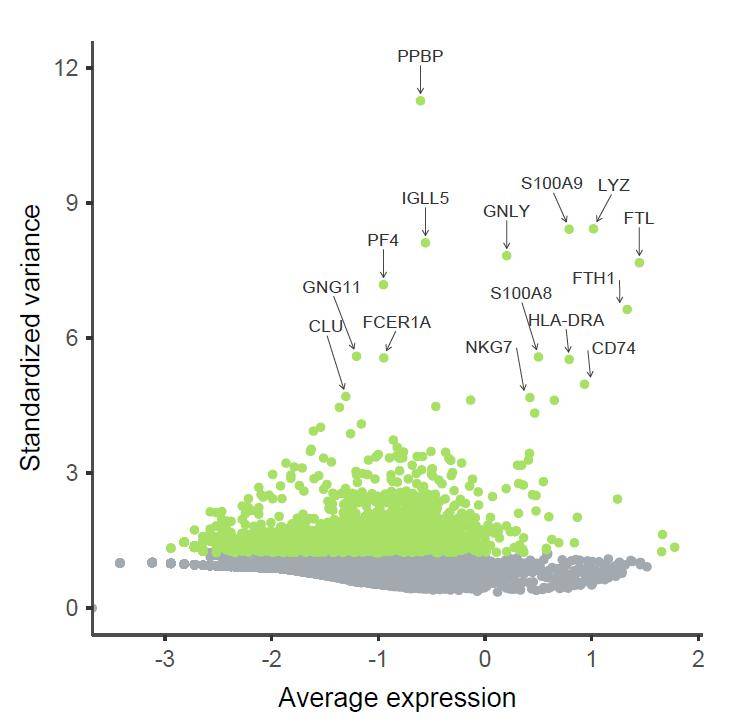
分组标记
上面的图表添加上箭头指引线,瞬间觉得最常见的散点图也变得“高大上”起来了呢。但对于“审美强迫症”来说,这些标签的排列还是有点乱,如何把他们排整齐呢?
下面就增加点难度,以图表的中轴线为界,把两侧的标签整齐排列。具体实现方法就是根据横坐标将数据集分成两部分,分别指定标签的偏移距离和排列方式。比如这里以“0”为分界,使用subset函数将数据框分成两部分,分两次添加文字标签
p4 <- ggplot(data = var.genes,aes(log10(vst.mean),vst.variance.standardized,
color=vst.variable,label=labels)) +
geom_point +
geom_text_repel(
data = subset( var.genes, log10(vst.mean) >= 0),
nudge_x = 3- log10(subset( var.genes, log10(vst.mean) >= 0)$vst.mean),
color = "grey20",
point.padding = NA,
size = 3,
segment.size = 0.2,
segment.color = "grey50",
direction = "y",
hjust = 1
) +
geom_text_repel(
data = subset( var.genes, log10(vst.mean) < 0),
nudge_x = -3- log10(subset( var.genes, log10(vst.mean) < 0)$vst.mean),
color = "grey20",
point.padding = NA,
size = 3,
segment.size = 0.2,
segment.color = "grey50",
direction = "y",
hjust = 0
) +
scale_x_continuous(
breaks = c( -3, -2, -1, 0, 1, 2, 3),
limits = c( -3.5, 3.5))
p4
这里较难理解一点的是nudge_x(“标签”到“点”的水平距离)的设置,要想标签垂直对齐排列,需要为标签指定同样的横坐标,如本文的-3和3,只要用-3和3分别减去“点”的横坐标值就是对应的nudge_x值啦!当然这里的hjust参数也很重要,“0”表示文字左对齐,“0.5”表示文字居中,“1”表示文字右对齐。接着是图表的主题调整,方法同上。
p4 + scale_y_continuous(breaks = c( 0, 3, 6, 9, 12),limits = c( 0, 12)) +
xlab( "Average expression") +
ylab( "Standardized variance") +
scale_color_manual(values=c( "#a2a9af", "#a8df65")) +
theme_classic+
theme(plot.margin = unit(c( 1, 1, 0.5, 0.5), "cm"),
legend.position = "none",
axis.title = element_text(size = rel( 1.2)),
axis.text = element_text(size=rel( 1)),
axis.line = element_line(size = 0.5, colour = "grey30"))
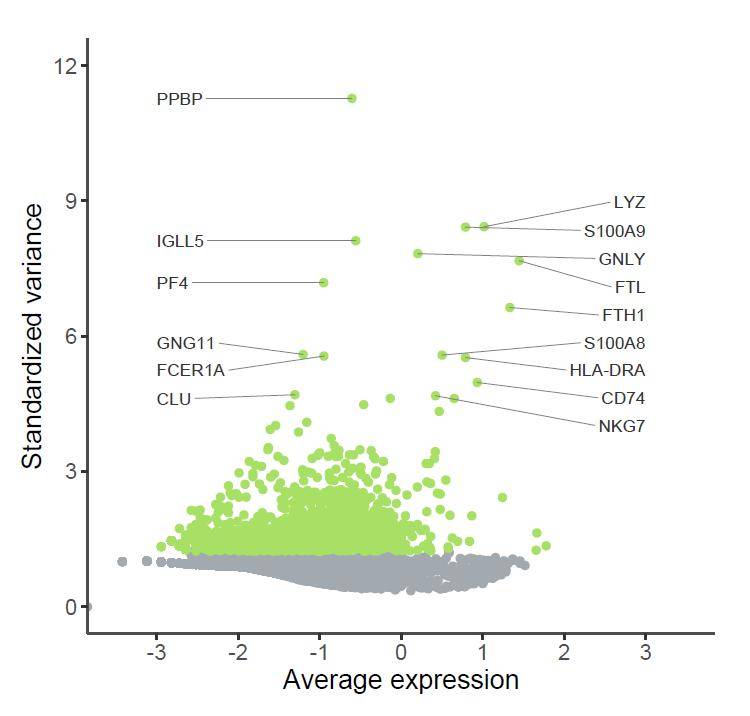
如果觉得纯文字标签逼格还不够,我们还可以用圆角矩形标签的方式展示,这时候我们就用到了geom_label_repel函数,参数与上文几乎一样,只需做简单调整。主要是通过fill参数调整矩形标签的颜色,其他不变。
p5 <- ggplot(data = var.genes,aes(log10(vst.mean),vst.variance.standardized,
color=vst.variable,label=labels)) +
geom_point +
geom_label_repel(
data = subset( var.genes, log10(vst.mean) >= 0),
nudge_x = 3- log10(subset( var.genes, log10(vst.mean) >= 0)$vst.mean),
color = "white",
alpha = 0.9,
point.padding = 0.5,
size = 3,
fill = "#96C93D",
segment.size = 0.5,
segment.color = "grey50",
direction = "y",
hjust = 1
) +
geom_label_repel(
data = subset( var.genes, log10(vst.mean) < 0),
nudge_x = -3- log10(subset( var.genes, log10(vst.mean) < 0)$vst.mean),
color = "white",
alpha = 0.9,
point.padding = 0.5,
size = 3,
fill = "#9881F5",
segment.size = 0.5,
segment.color = "grey50",
direction = "y",
hjust = 0
) +
scale_x_continuous(
breaks = c( -3, -2, -1, 0, 1, 2, 3),
limits = c( -3.5, 3.5))
p5
图表的主题调整同上。
p5 + scale_y_continuous(breaks = c( 0, 3, 6, 9, 12),limits = c( 0, 12)) +
xlab( "Average expression") +
ylab( "Standardized variance") +
scale_color_manual(values=c( "#a2a9af", "#ff85cb")) +
theme_classic+
theme(plot.margin = unit(c( 1, 1, 0.5, 0.5), "cm"),
legend.position = "none",
axis.title = element_text(size = rel( 1.2)),
axis.text = element_text(size=rel( 1)),
axis.line = element_line(size = 0.5, colour = "grey30"))
绘制效果如下:
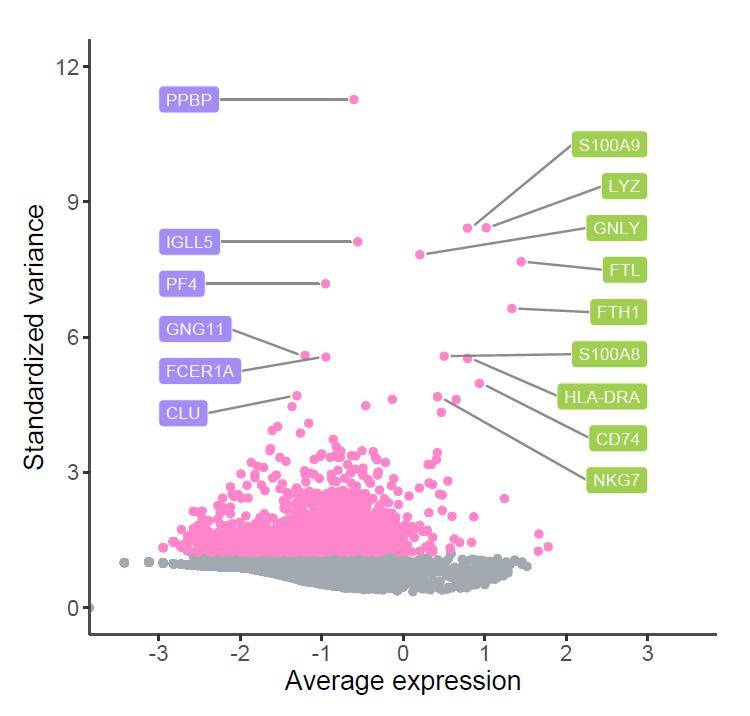
是不是好看多了?至此,添加个性化的标签无需Ai(Illustrator)也能批量自动完成啦!今天的内容就先到这里啦~
- 本文固定链接: https://maimengkong.com/kyjc/958.html
- 转载请注明: : 萌小白 2022年6月3日 于 卖萌控的博客 发表
- 百度已收录
