什么是火山图?
火山图(Volcano plot)——顾名思义,因其绘制后图形的效果酷似火山而命名。用于显著差异基因表达的展示,包含显著和差异两个重要信息。显著性一般用T-Test的P值衡量,差异性常用Fold Change值作为筛选标准。
如何看懂火山图?
每个人在使用火山图时的需要可能不一样,绘制出来的效果就不一样。小编汇总了部分火山图的展示效果,给大家一些参考~
看到这些火山图,我们的疑问是:如何看懂这张火山图所包含的信息呢?
简单来说火山图的本质就是一张散点图,被用来展示显著差异表达的基因,故而我们需要两个数据:一个是 p 值,即显著性;另一个是 FC 值(Fold Change),即差异表达倍数。
同时,我们在解读一张火山图时应该如何看懂?我们可以通过观察他的横纵坐标log2(Fold Change)和-log10(p-value);火山图选用的作图数据是经过log2处理的Fold Change(FC)和经过-log10处理的p-value(T检验显著性P指的负对数)。标准的火山图将所有的数据点分为了 6 个区域,其中A 区代表着显著性差异且下调的数据;C 区为显著差异且上调的数据;其他区为不显著差异的数据。不显著差异的数据具体又分为显著无差异(B)、差异不显著(D、F)、不显著且不差异(E)等不同区域。
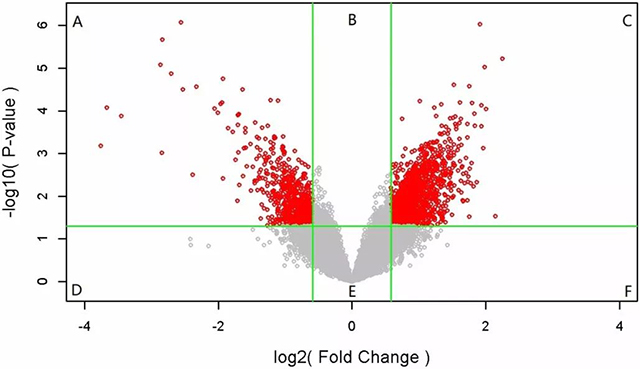
所以简而言之一句话,火山图最左上角(A)和右上角(C)的物质是在FC检验和T检验中,显著差异性最大的物质。所以我们火山图最重要的就是左上角和右上角的物质了。
如何绘制火山图?
我们在知道了什么是火山图?如何理解看懂火山图之后?我们进入的第三个问题就是,如何绘制火山图呢?
说起绘图,有些同学脑中可能会浮现出Photoshop、Adobe image 、AutoCAD等,但是生物信息学分析的绘图是基于数据做的可视化。需要的并不是常规的绘图软件,而是可用于数据分析和可视化的变成软件和语言,这里我们给大家介绍的是最普遍、最常见、也是非常容易上手的R语言来做这个火山图。
1.数据处理
我们首先要对数据进行处理,其中包括对数据进行T检验、计算p-value和-log10(P-value)、进行Fold Change(FC)检验、计算Fold Change(FC)和log2(Fold Change);进行多元统计,得到VIP值。然后根据Fold Change(FC)和VIP的值判断代谢物的上下调,这一步的简单方式可以在excel中计算后粘贴到TXT文件中哦~
示例如下:
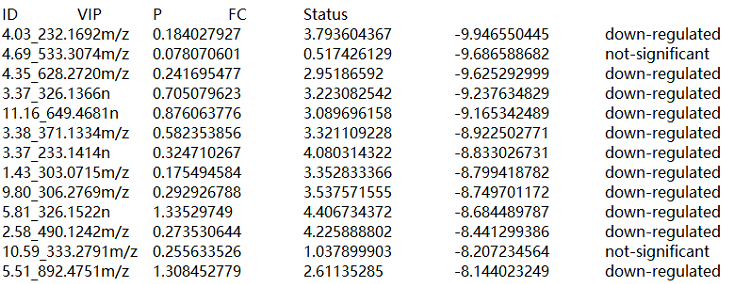
注:(图片中的P是指-log10(P-value),FC是指log2(Fold Change)哦)
2.设置路径和数据加载
设置路径:setwd('F:/test/vol') #这是文件vol.txt的路径,注意要用/哦~
数据加载:
data<- read.table("vol.txt",head = T,sep="\t",row.names=1,encoding="utf-8",check.names=F,quote="",as.is=TRUE) #加载我们预先处理好的数据vol.txt文件
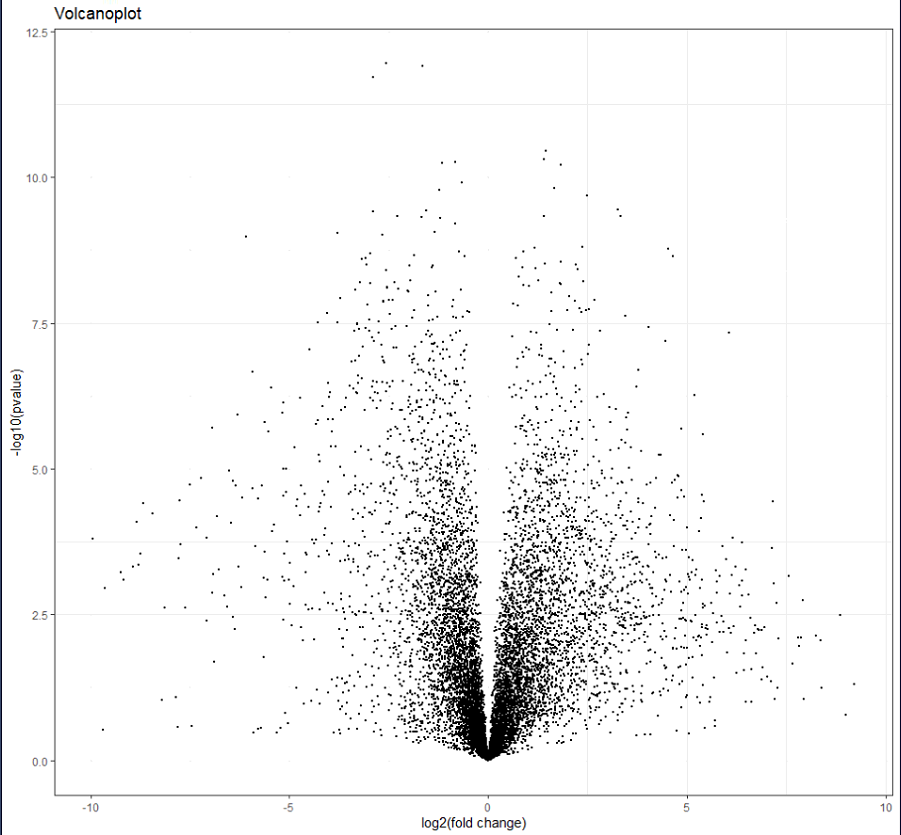
3.绘制火山图·基础版
我们在此次火山图绘制的时候使用的是R语言的ggplot2包,这是一款很适合火山图使用且有非常强大功能的包。
library("ggplot2") #导入ggplot2包
volcano <- ggplot(data, aes(x= FC, y= P)) #设置x轴和y轴数据
volcano+geom_point(size=0.5)+ #设置点的大小
labs(title="Volcanoplot",x="log2 (fold change)",y="-log10(pvalue)") #设置横纵坐标及标题
示例如下:
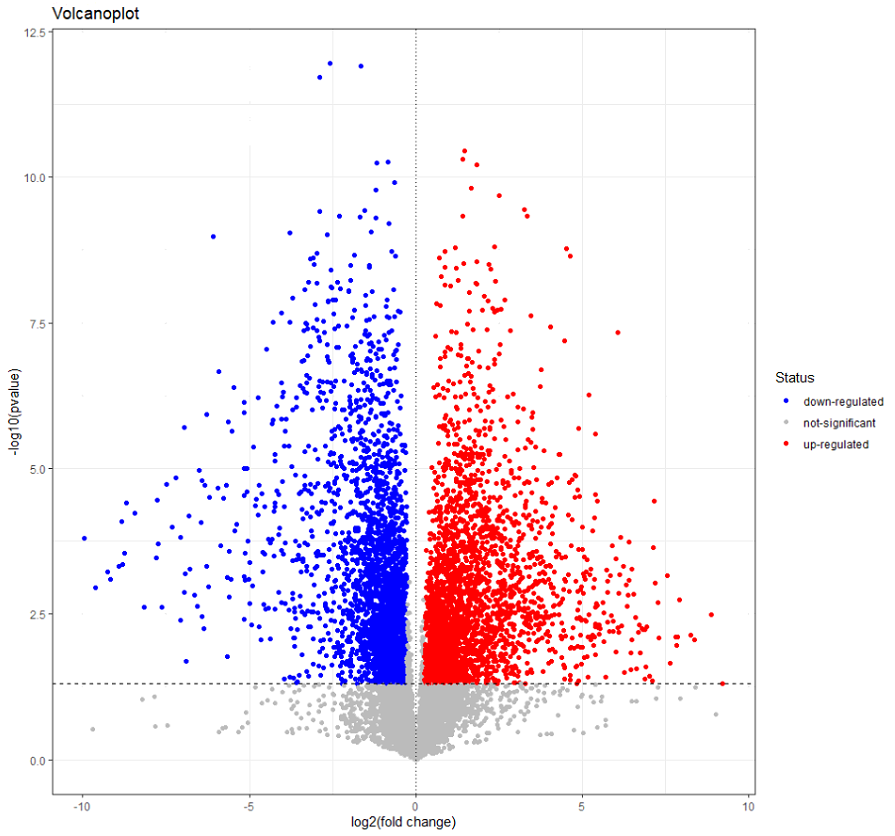
4.美化的“亿点细节”
我们现在得到的火山图明显不够“漂亮”,为了图片更漂亮(不是),为了能更好的展示实验数据,分析代谢物的差异性,我们需要对这张图做“亿点”小小的,细节上的美化~
library("ggplot2")
volcano <- ggplot(data, aes(x= FC, y= P)) #设置x轴和y轴数据
volcano+geom_point(aes(color=Status))+ #加入上下调图例
scale_color_manual(values=c("blue","grey","red"))+ #区分上下调点的颜色
labs(title="Volcanoplot",x="log2 (fold change)",y="-log10 (pvalue)")+ #设置横纵坐标及标题
geom_hline(yintercept=1.3,linetype=2)+ #添加辅助线,选择辅助线的类型
geom_vline(xintercept=0,linetype=3)+ #添加辅助线,选择辅助线的类型
theme(legend.position='right') #图例的位置在右侧
示例如下:
- 本文固定链接: https://www.maimengkong.com/kyjc/882.html
- 转载请注明: : 萌小白 2022年4月30日 于 卖萌控的博客 发表
- 百度已收录
