这期番茄君为大家带来如何利用DAVID在线数据库做GO与KEGG分析。
1.点击文末“阅读全文”,获取DAVID在线数据库网址链接。打开后如下(部分):
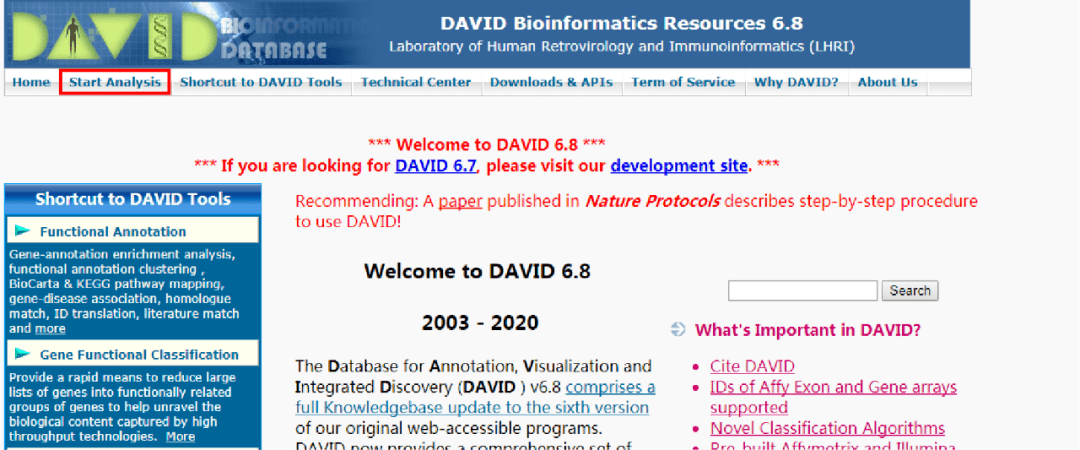
2.点击上图中红色框里的Start Analysis。如下:
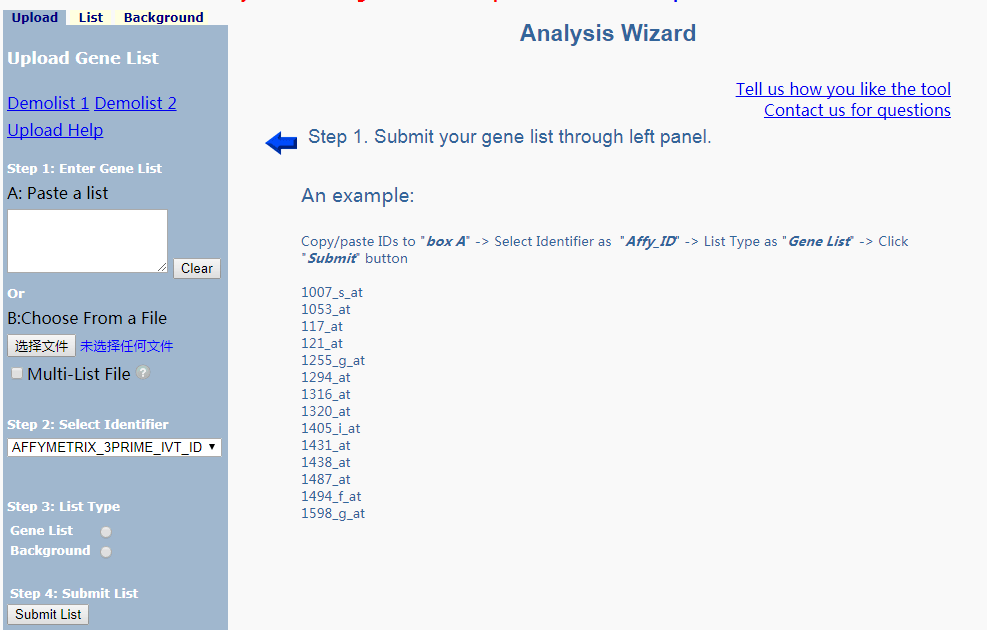
3.上传基因。Step1: Enter Gene List有两个选项可以上传基因:第一种方法是在A: Paste a list的白色框里输入基因名字;第二种方法是利用B:Choose From a File上传文件。我们输入了57个和奶牛产奶性状相关的基因(注意,所有基因排成一列后再输入,如果是上传word文档,也是将所有基因排成一列)。
4.点击Step2: Select Identifier下拉选项框,由于我们直接输入的就是基因的名字,所以这里选择OFFICIAL_GENE_SYMBOL。
5.点击Step3:List Type的选项Gene List。
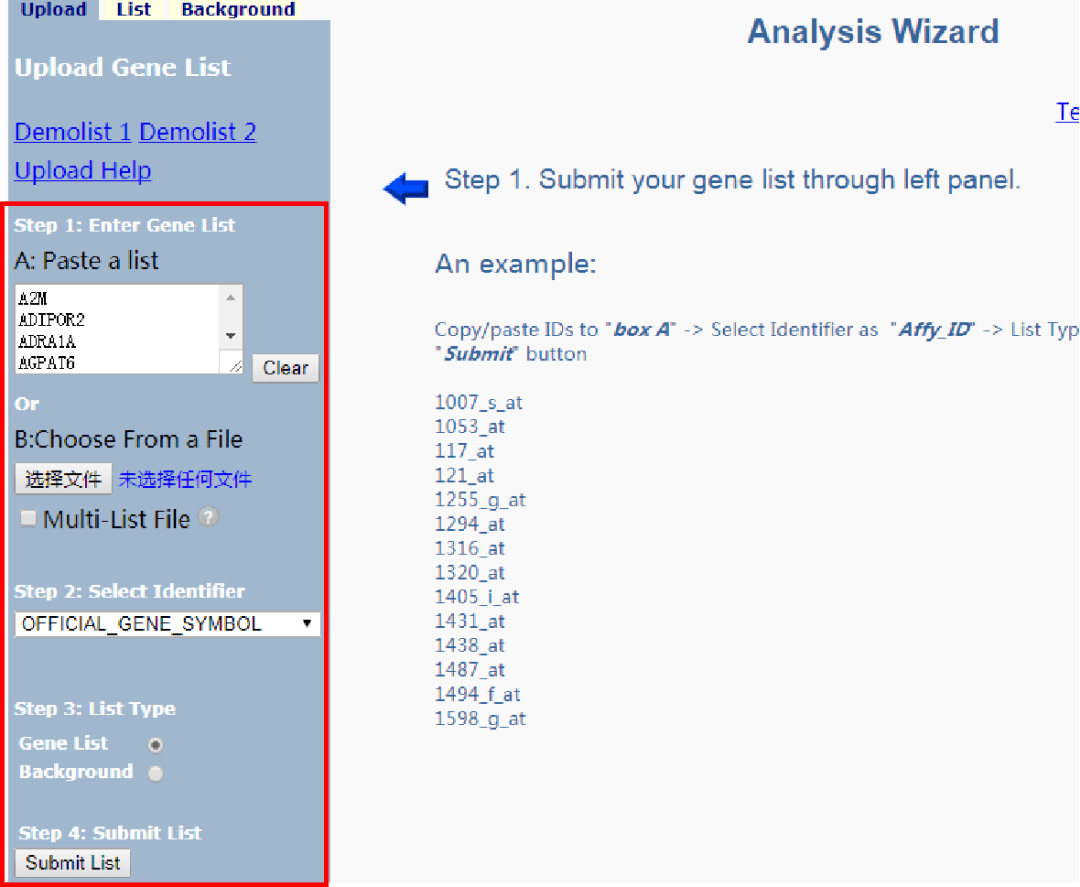
6.点击Step4:Submit List的Submit List。出现如下提醒,原因是我们没有选择物种,先点击确定,自动转到了List栏。
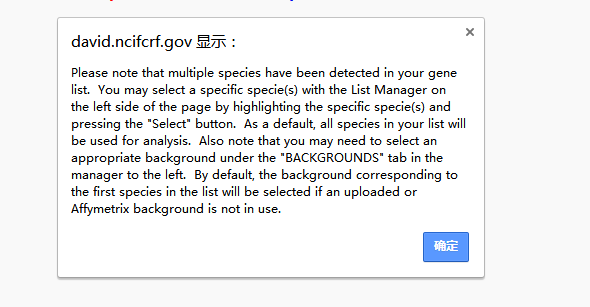
7.选择物种。因为我们输入的这些基因与奶牛产奶性状相关,所以选择奶牛Bos taurus,点击selected species。
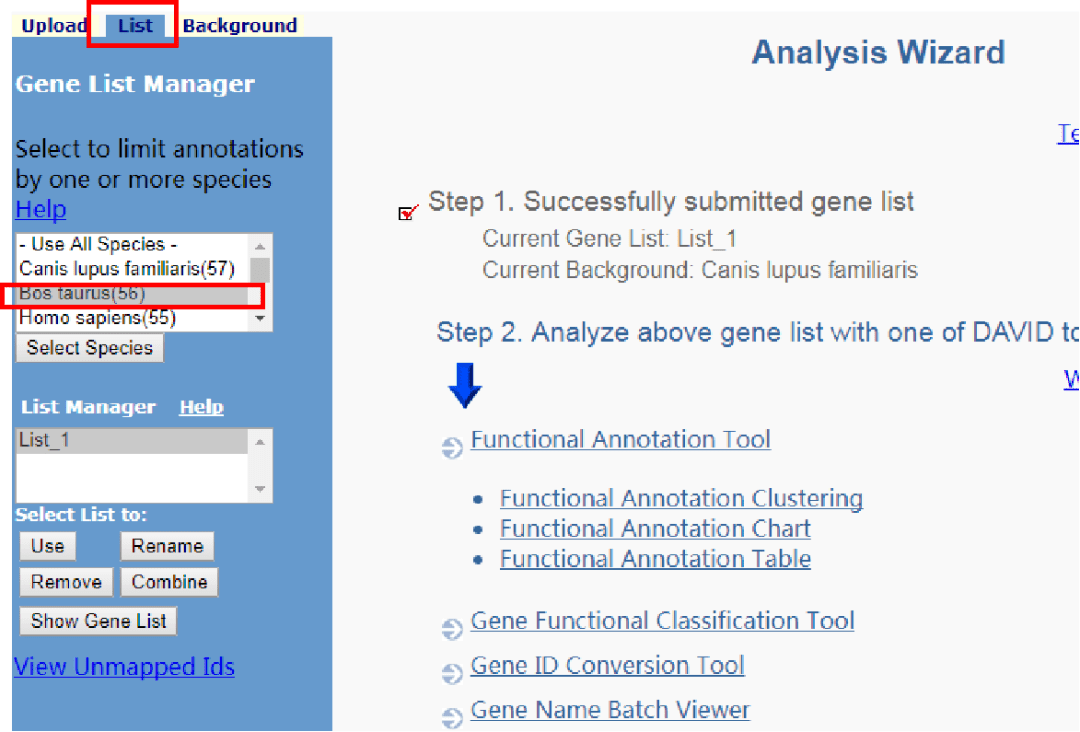
8.点击Backgroud,同样选择Bos taurus,点击use。
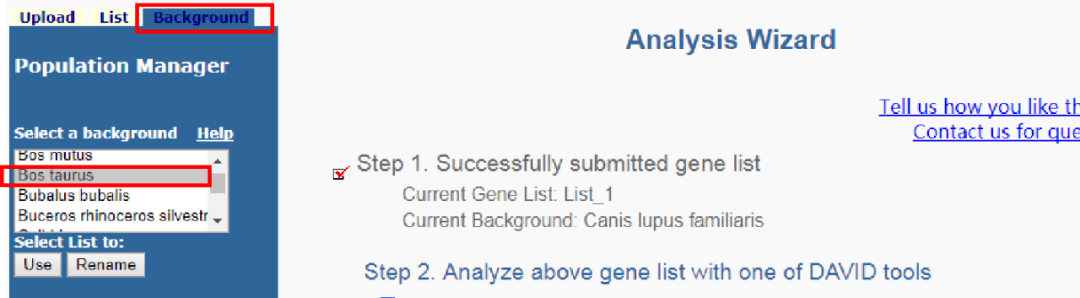
这样分析过程就完成了,接下来我们来看一下结果解析。
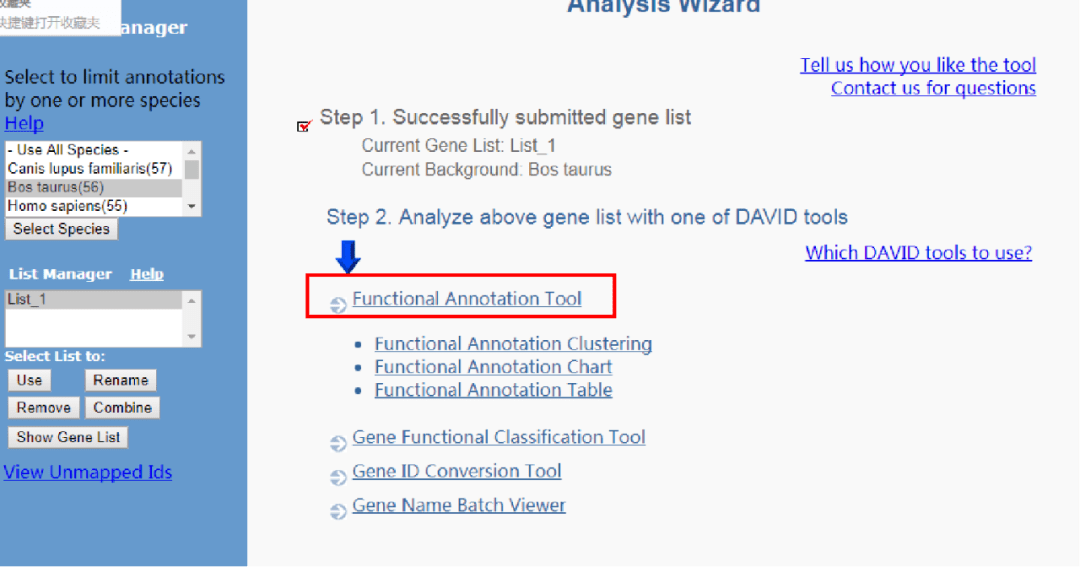
1.点击Functional Annotation Tool,跳转到Annotation Summary Results界面。
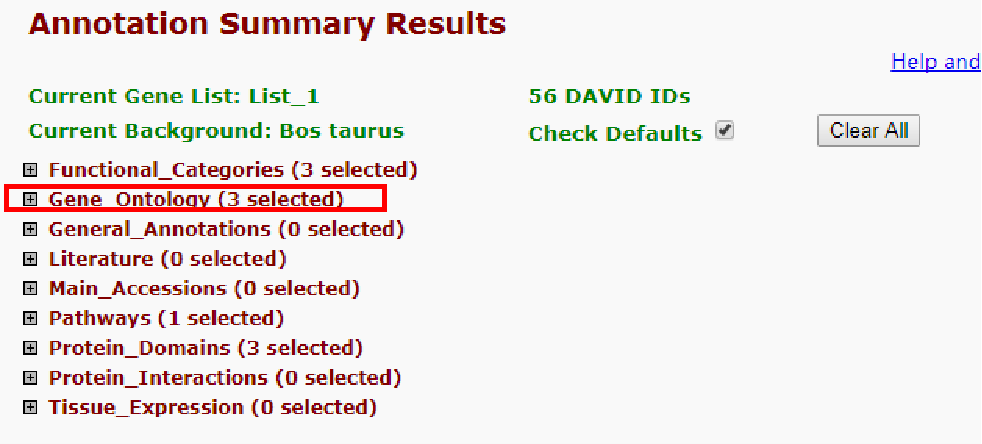
2.点击上图中Gene_Ontology前面的+号,即出现GO分析的三种类型BP, CC和 MF。
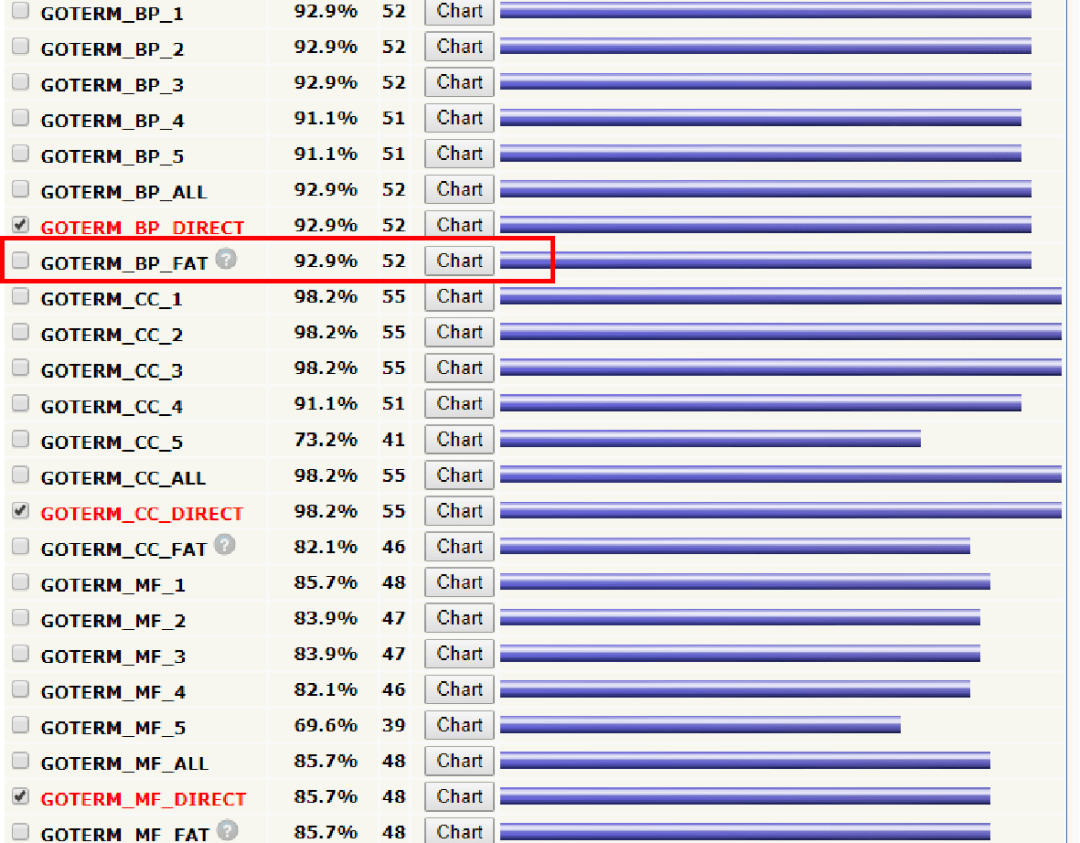
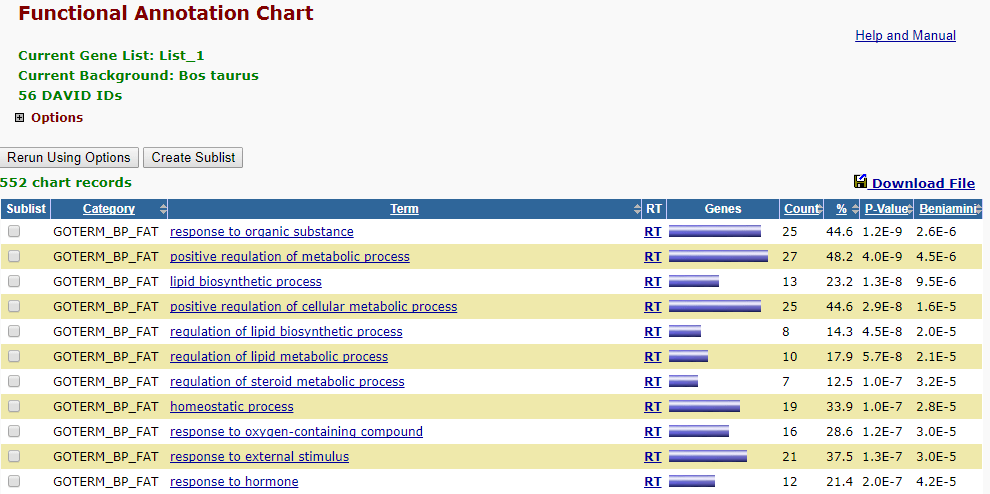
3.点击GOTERM_BP_FAT后面的Chart,即出现了这些基因聚集到的BP类型。
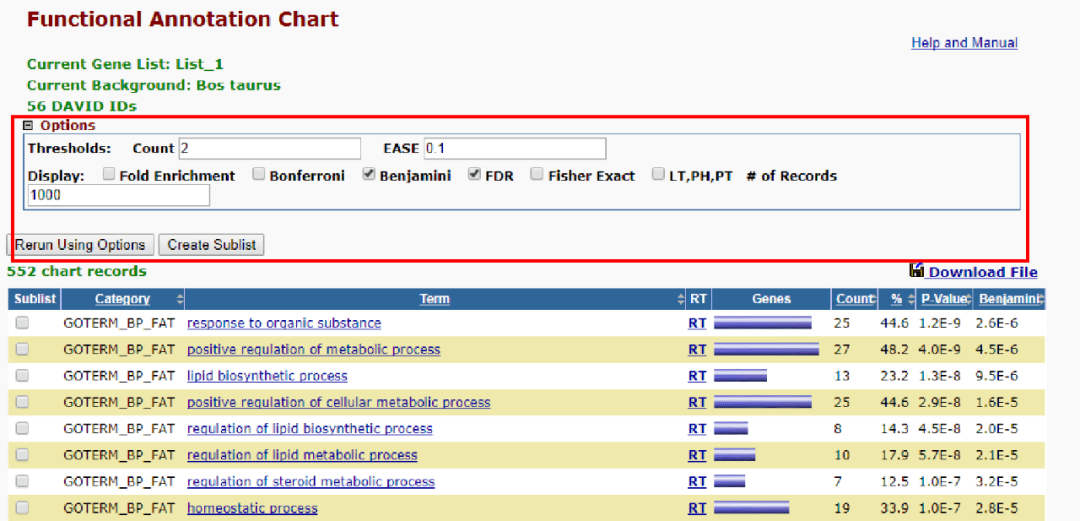
还可以选择不同的参数,点击Options前面的+号,有些文章以FDR为参考,选中FDR,点击Rerun Using Options即可。
同理,GO分析的CC和MF类型以及KEGG_PATHWAY分析的结果解析和上面是一样的。
DAVID数据库功能强大,远不止可以做GO和KEGG分析,还可以做基因ID之间的转换、ID转换为基因名称等等。当然做GO与KEGG分析的数据库还有很多很多,例如KOBAS以及其他在线数据库等等,各有优缺点,按需所取。- 本文固定链接: https://www.maimengkong.com/kyjc/1009.html
- 转载请注明: : 萌小白 2022年6月22日 于 卖萌控的博客 发表
- 百度已收录
