一、根底知识
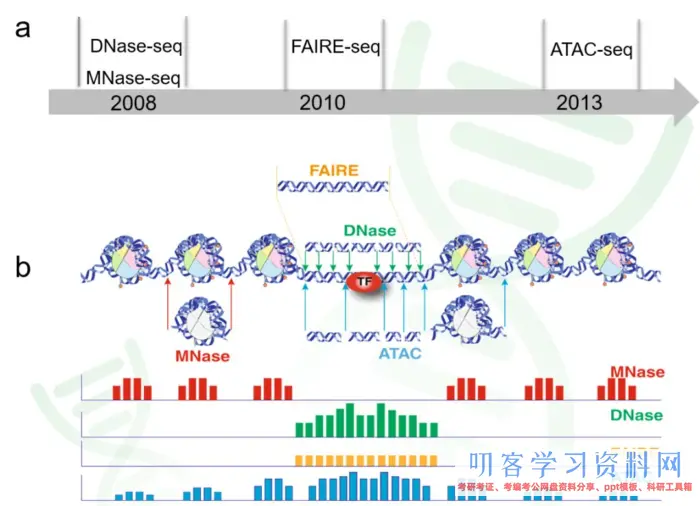
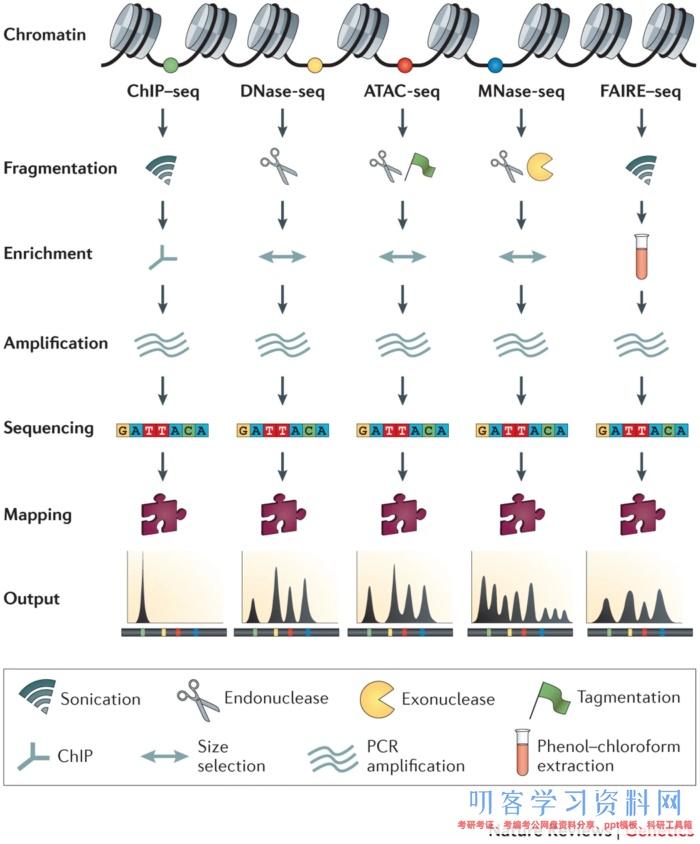
1. 解密表观遗传学的三个方向与测序办法
-
1. 探究染色质的敞开性(chromatin accessibility)
ATAC-seq: Assay of Transposase Accessible Chromatin sequencing
DNase-seq: DNase I hypersensitive sites sequencing
FAIRE-seq: Formaldehyde-Assisted Isolation of Regulatory Elements sequencing -
2. 探究转录因子的绑定与组蛋白润饰(TF binding and histone modifications)
ChIP-seq: Chromatin Immuno-Precipitation sequencing -
3. 探究核小体占位(nucleosome positioning and occupancy)
MNase-seq: Micrococcal Nuclease sequencing
2. 名词解说
-
epigenetics:表观遗传学。表观遗传学润饰在不改动DNA序列的状况下操控着基因的表达,包括染色质重塑、组蛋白润饰、DNA甲基化和microRNA通路等 -
peaks: 峰。常用来标明染色质的敞开程度,因为是测序的reads落在了染色质的敞开区,堆叠后被可视化的一种丰度的表现。
但peaks究竟代表什么?莫非peaks地点区域要么是启动子要么是增强子?再或许便是抽象界说为调控元件?(我现在的观念是,只能把他了解为酶切的位点,假如非要界说是否是特定分调控元件,只能进行后续剖析。)
-
THSs: 转座酶超敏感位点(transposase hypersensitive sites)。 -
CREs: 顺式调控元件(cis-regulatory elements)。即DNA分子中具有转录调理功用的特异DNA序列。按功用特性,真核基因顺式作用元件分为启动子、增强子及沉默子。 -
ACRs: 染色质敞开区域(accessible chromatin regions)。即正常或核小体被酶切裸露出来的DNA片段地点的区域。这篇【NP | 2019】根据ACRs距离最近基因的距离将ACRs分为三种类型:genic (gACRs; overlapping a gene), proximal (pACRs; within 2kb of a gene) or distal (dACRs; >2 kb from a gene),别离是跨越基因的,近端的,远端的染色质敞开区。 -
transposon: 转座子。一段能够从原位上独自复制或断裂下来,环化后刺进另一位点,并对其后的基因起调控作用的DNA序列。 -
promoter: 启动子。启动子是坐落结构基因5\'端上游的DNA序列,能活化RNA聚合酶,使之与模板DNA精确的结兼并具有转录开端的特异性。每个启动子包括至少一个转录开端点以及一个以上的功用组件(典型的如TATA盒子) -
proximal promoters: 近端启动子。是DNA上坐落基因开端之前的一个区域,在那里蛋白质和其他分子结合在一起预备读取该基因。 -
enhancer: 增强子。增强子是远离转录开端点、决定基因的时刻、空间特异性表达、增强启动子转录活性的DNA序列,其发挥作用的办法一般与方向、距离无关,可坐落转录开端点的上游或下游。从功用上讲,没有增强子存在,启动子一般不能表现活性;没有启动子时,增强子也无法发挥作用。根据南京大学陈迪俊老师的研究标明增强子比启动子能结合更多的转录因子(Nature Communications) -
TFs: 转录因子(transcription factors)是保证意图基因以特定的强度在特定的时刻与空间表达的蛋白质分子。与RNA聚合酶Ⅱ形成转录开端复合体,共同参加转录开端的过程。 -
TSS: 转录开端位点(transcription start site)。在一个典型的基因内部,摆放顺序为转录开端位点(TSS,一个碱基)-开端密码子编码序列 (ATG)-停止密码子编码序列(TGA)-转录停止位点 (TTS),即TSS-ATG-TGA-TTS -
histone:组蛋白。一般含有H1,H2A,H2B,H3,H4等5种成分,其间H1与H3极度富含赖氨酸(lysine),H1不保存,其他组蛋白的基因非常保存。除H1外,其他4种组蛋白均别离以二聚体(共八聚体)相结合,形成核小体核心。DNA便环绕在核小体的核心上。而H1则与核小体间的DNA结合 -
nucleosome: 核小体。是由DNA和组蛋白形成的染色质基本结构单位。每个核小体由146bp的DNA环绕组蛋白八聚体1.75圈形成。核小体核心颗粒之间经过50bp左右的衔接DNA相连,暴露在核小体外表的DNA能被特定的核酸酶接近并切开 -
H3K4me1: 组蛋白H3上的第4位赖氨酸产生单甲基化(mono-methylation of H3 at lysine 4) (NG | H3K4me1与增强子的关系) -
H3K9ac:组蛋白H3上的第9位赖氨酸产生乙酰化(H3 acetylation marks at lysine 9)关于H3K4me1与H3K9ac的不同,仍旧能够参阅这篇文献【NP | 2019】或【NC | 2019】 -
组蛋白甲基化:甲基化可产生在组蛋白的赖氨酸和精氨酸残基上,而且赖氨酸残基能够产生单、双、三甲基化,而精氨酸残基能够单、双甲基化,这些不同程度的甲基化极大地增加了组蛋白润饰和调理基因表达的复杂性 -
组蛋白乙酰化:四种类型的组蛋白相互作用,将细胞核里的DNA紧紧地包装起来。这样的紧密包装能够有用阻挠酶读取DNA上的遗传信息。可是,乙酰基连到组蛋白上能削弱它们对DNA的占有。因而部分乙酰化能暴露出相应的基因,让它们更容易激活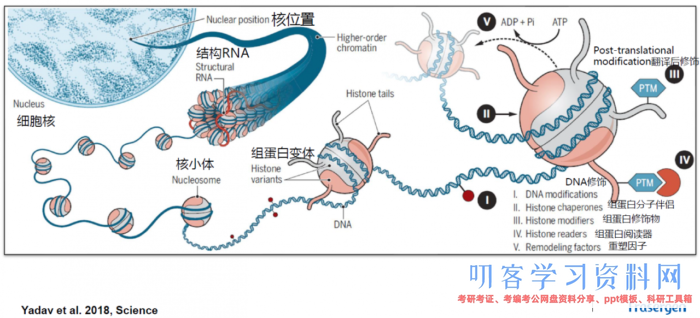 真核生物细胞核内染色质结构
真核生物细胞核内染色质结构
二、根底剖析
1. 剖析前的预备
1.1 下载ATAC测序数据(或许用你自己的数据也行)
- 所需软件:Aspera Connect
- 软件装置与运用:参阅这系列文章
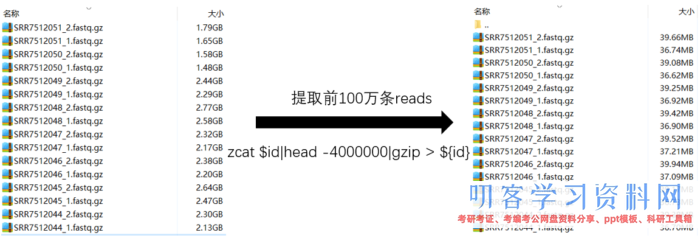
- 数据下载:项目ID【PRJNA480717】【文章原文】,SRR序列号如下图,下载办法和上文相同。因为数据量大,测验浪费时刻,所以我在每个文件中取了100w条reads,因为百度网盘太慢了,你能够去扣扣群559758504里边随意下载一个用于测验。
# 提取100w条reads代码
ls *gz|while read id;do zcat $id|head -4000000|gzip > ./100/${id};done
1.2 下载tair10基因组与gff3注释文件
wget https://www.arabidopsis.org/download_files/Genes/TAIR10_genome_release/TAIR10_chromosome_files/TAIR10_chr_all.fas wget https://www.arabidopsis.org/download_files/Genes/TAIR10_genome_release/TAIR10_gff3/TAIR10_GFF3_genes_transposons.gff # 改名 mv TAIR10_chr_all.fas TAIR10_genome.fa mv TAIR10_GFF3_genes_transposons.gff TAIR10.gff3
1.3 装置miniconda
- 因为后续剖析大多数软件都是来源于conda,因而你有必要知道怎样运用。参阅【conda的装置与运用】
1.4 所需软件装置(剖析中所用到的软件都汇集于此,下文不再介绍怎么下载)
请先激活conda环境在装置!
conda install -c bioconda -y fastqc conda install -c bioconda -y trimmomatic conda install -c bioconda -y bwa conda install -c bioconda -y samtools conda install -c bioconda -y picard conda install -c bioconda -y macs2 conda install -c bioconda -y bedtools conda install -c bioconda -y deeptools conda install -c bioconda -y homer
后续剖析是运用SRR7512044作为测验数据,可是思路和办法都能够学习。
2. 测序质量检测
- 所需软件:FastQC【官方网站】
fastqc -t 8 -o ./ *.fastq.gz
-t: 线程
-o: 存放途径,不必指定前缀,默以为.fastq.gz前面的字段
*.fastq.gz: fastqc能够同时承受多个fastq.gz文件,因而选用正则表达式【*】标明全部
-
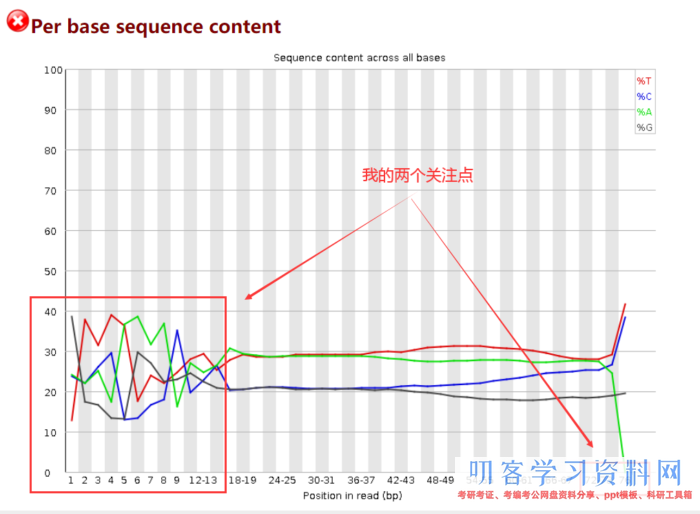
成果解读
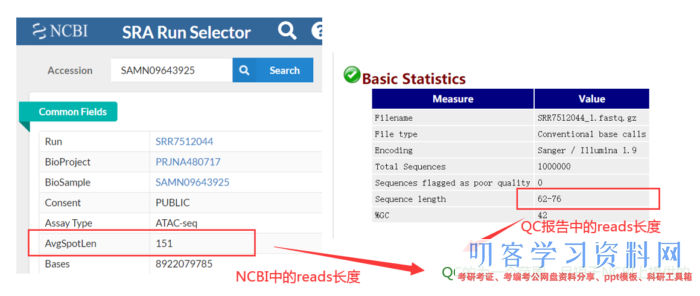
因为开端测序时不精确,因而前15bp左右的ATGC含量有动摇,需求在后边截掉。而最长reads为76bp,则暗示着我最终至少要保存多长的序列。
一般我的核算思路是:保存的序列长度 =(最长的的reads长度-前面动摇的碱基长度)× 40%
2. 测序质量检操控
-
所需软件:Trimmomatic【官方网站】【中文介绍】
其实这批数据现已做过质控了,怎么判断的呢?QC的为一个范围且短于供给给NCBI上的数据。因而咱们没必要质控,但为了流程的完好性,仍是假装质控一下。质控之后,还要进行质检,如此循环,直到合格。
# 界说一些变量,后边修改起来便利
filename=SRR7512044
trimmomatic PE -threads 10
./${filename}_1.fastq.gz ./${filename}_2.fastq.gz
./${filename}_1.clean.fq.gz ./${filename}_1.drop.fq.gz
./${filename}_2.clean.fq.gz ./${filename}_2.drop.fq.gz
HEADCROP:15 LEADING:3 TRAILING:3 SLIDINGWINDOW:4:15 MINLEN:24
PE: 双端模式。需求两个输入文件,正向测序序列和反向测序序列:sample_R1.fastq.gz和sample_R2.fastq.gz;
以及四个输出文件:sample_1.clean.fq.gz和sample_1.drop.fq.gz;sample_2.clean.fq.gz和sample_2.drop.fq.gz
-threads: 线程数
HEADCROP: 从 reads 的最初切掉指定数量的碱基,即从fastqc中看的
LEADING: 从 reads 的最初切除质量值低于阈值的碱基
TRAILING: 从 reads 的结尾开端切除质量值低于阈值的碱基
SLIDINGWINDOW: 从 reads 的 5\' 端开端,进行滑窗质量过滤,切掉碱基质量平均值低于阈值的滑窗
MINLEN: 假如经过剪切后 reads 的长度低于阈值则丢掉这条 reads,即上面的核算办法:(76-15)×40%≈24
3. 建立索引与序列比对
- 所需软件:bwa和samtools
- 建立基因组索引。需求的时刻挺长的,主张放后台。建好之后以后就能够越过这一步了,且在经典的比对软件STAR、hisat2、bowtie2和bwa-mem中我更喜欢运用STAR和bwa-mem。本次运用bwa-mem操作演示
bwa index -p /途径/bwaIndexTair /途径/TAIR10_genome.fa
-p: 索引前缀,后缀自动弥补
TAIR10_genome.fa: 基因组文件
-
序列比对
因为sam文件较大,因而咱们直接越过sam,运用samtools转化为排序后的bam文件,留意【|】不要漏掉,且排序是根据reads比对到基因组的方位排的
# 界说一些变量,后边用
filename=SRR7512044
index=/途径/bwaIndexTair
bwa mem -M -t 8
-R \"@RGtID:${filename}tSM:${filename}tLB:WXStPL:Illumina\"
$index
${filename}_1.clean.fq.gz
${filename}_2.clean.fq.gz
| samtools sort -O bam -@ 10 -o ./${filename}.raw.bam
#无论干啥,生成bam文件后就要建立bam索引,并检查比对状况
samtools index -@ 10 -b ./${filename}.raw.bam
samtools flagstat ./${filename}.raw.bam > ./${filename}.raw.stat
cat ${filename}.raw.stat
-M: 将 shorter split hits 标记为次优,以兼容 Picard’s markDuplicates 软件
-R: STR 完好的read group的头部,能够用 \'t\' 作为分隔符, 在输出的SAM文件中被解说为制表符TAB. read group 的ID,会被添加到输出文件的每一个read的头部,
-O: 标明输出的bam文件
-o: 输出的bam文件名
-t与@: 线程数
- 关于stat中内容的解说如下
14608455 0 in total (QC-passed reads QC-failed reads) reads总数 37967 0 secondary 出现比对到参阅基因组多个方位的reads数 0 0 supplementary 或许存在嵌合的reads数 0 0 duplicates 重复的reads数 14590894 0 mapped (99.88% : N/A) 比对到参阅基因组上的reads数 14570488 0 paired in sequencing 归于PE read的reads总数。 7285244 0 read1 PE read中Read 1 的reads 总数。 7285244 0 read2 PE read中Read 2 的reads 总数。 14507068 0 properly paired (99.56% : N/A) 完美比对的reads总数。PE两头reads比对到同一条序列,且根据比对成果揣度的刺进片段巨细符合设置的阈值。 14551500 0 with itself and mate mapped PE两头reads都比对上参阅序列的reads总数。 1427 0 singletons (0.01% : N/A) PE两头reads,其间一端比上,另一端没比上的reads总数。 26260 0 with mate mapped to a different chr PE read中,两头别离比对到两条不同的序列的reads总数。 17346 0 with mate mapped to a different chr (mapQ>=5) PE read中,两头别离比对到两条不同
4. 去除PCR重复并再次进行质控
-
所需软件:picard与samtools
不主张运用sambamba,尽管快,可是缺点多,尤其是在你不知情的状况下,软件卡死或许在最终输出文件时提示输出文件夹过多等。
picard MarkDuplicates
REMOVE_DUPLICATES=true
I=${filename}.raw.bam
O=${filename}.rmdup.bam
M=${filename}.rmdup.log
# 实时监测咱们的数据产生了什么改变
samtools index ${filename}.rmdup.bam
samtools flagstat ${filename}.rmdup.bam > ./${filename}.rmdup.stat
REMOVE_DUPLICATES=true: 标明将检测出的PCR duplication直接去除;
I: 或许INPUT指定输入的bam文件;
O: 或许OUTPUT指定输出去除PCR duplication的bam文件;
M: 或许METRICS_FILE指定输出的matrix log文件。
- 下面是再次进行质控
samtools view -h -f 2 -q 30 ${filename}.rmdup.bam
| grep -v -e \"mitochondria\" -e \"*\" -e \"chloroplast\"
| samtools sort -O bam -@ 10 -o - > ${filename}.last.bam
# 实时监测咱们的数据产生了什么改变
samtools index ${filename}.last.bam
samtools flagstat ${filename}.last.bam > ./${filename}.last.stat
-h: 输出的sam文件带header,默许不带
-f: 输出含有一切flag的reads
-q 比对的最低质量值,一般20/30都能够
其他参数介绍看 samtools 运用简述,需求阐明的是因为比对到线粒体和叶绿体的reads不是咱们关注的重点,因而运用grep除掉掉,当然了,做动物的话需求除掉什么视状况而定了。
-
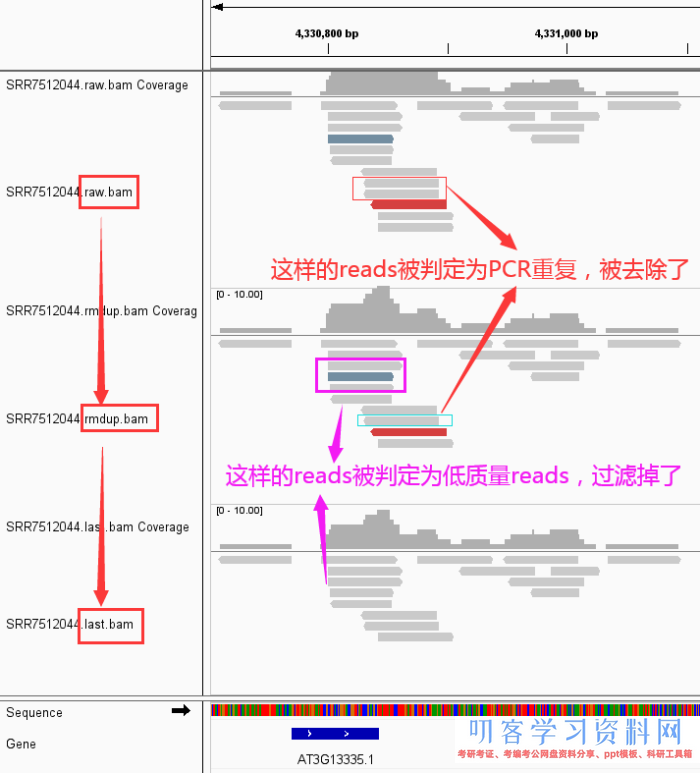
好了,下面来看看咱们的数据从 raw data -> rmdup -> last 究竟产生了那些改变

-
然后,从IGV看看产生了什么改变
关于运用IGV检查bam文件中reads出现不同颜色的阐明
5. 核算过滤后的成果中Insertsize长度散布
-
所需软件:picard
Insertsize =(read2的比对位点 - read1的比对位点) read2的长度。也能够随意点击上图IGV中任一一条reads检查刺进片段的巨细
picard CollectInsertSizeMetrics
I=./${filename}.last.bam
O=./${filename}.last.insertsize
H=./${filename}.last.hist.pdf
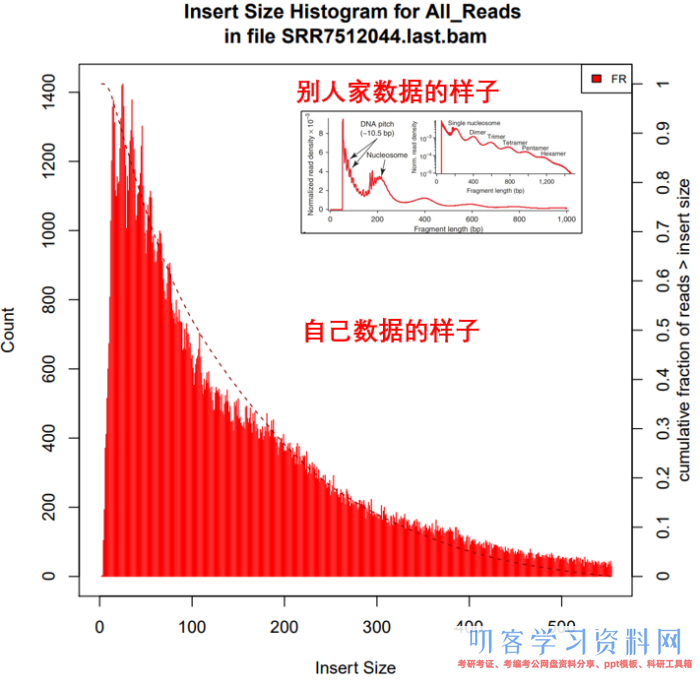
5. peak calling与FRiP_score/SPOT_score的核算
-
所需软件:macs2和bedtools
因为macs2需求用到基因组的有用巨细,而软件本身就供给了4个物种的有用基因组巨细。
effective_genome_size=119481543
bedtools bamtobed -i ${filename}.last.bam > ${filename}.last.bed
macs2 callpeak
-t ${filename}.last.bed
-g ${effective_genome_size}
--nomodel --shift -100 --extsize 200
-n ${filename}.last
-q 0.01 --outdir ./peaks
-
其实,这儿边评论最多的是
--nomodel --shift -100 --extsize 200这些参数怎么挑选,下面的图很形象的展现了参数的作用。当然,我也是查阅了许多资料与文献,一般默许在ATAC-seq,DNase-seq,FAIRE-seq的时分将shift设置为extsize的一半,且参数固定为:--nomodel --shift 100 --extsize 200。而在MNase-seq的时分,参数固定为:--nomodel --shift 37 --extsize 73。在ChiP-seq的时分不必移峰,所以只运用-nomodel,作为组蛋白润饰的时分,因为peak并不典型,所以运用–nomodel –shift 73参数。别的怎么运用macs2 predictd预测双峰模型,这儿就不展开介绍了,能够私聊我,我把脚本发你。
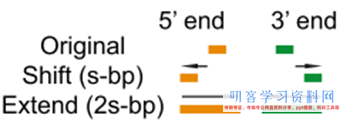
- 下面是我见过的图中解说移峰现象最清楚的了,能够参阅
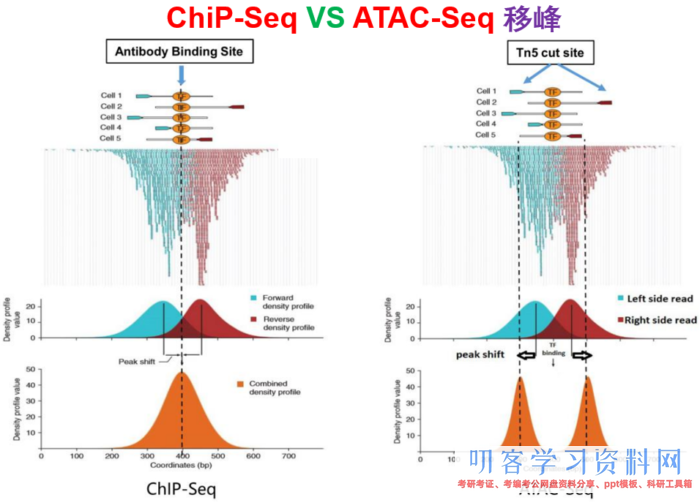
-
最终,便是输出文件的解读
- ${filename}_peaks.xls
前几行是callpeak时的指令。后边是: 1.染色体号 2.peak开端位点 3.peak结束位点 4.peak区域长度 5.peak的峰值位点(summit position) 6.peak 峰值的高度(pileup height at peak summit, -log10(pvalue) for the peak summit) 7.peak的富集倍数(相对于random Poisson distribution with local lambda)
-
- ${filename}_peaks.narrowPeak
narrowPeak文件是BED6 4格局,能够上传到IGV检查 1.染色体号 2.peak开端位点 3.peak结束位点 4.peak name(样本名 _peak_1的后缀之类的) 5.-10*log10qvalue 6.正负链 7.fold change 8.-log10pvalue 9.-log10qvalue 10.相对于峰的峰顶方位(relative summit position to peak start)
-
留意:excel里的开端坐标需求减1才与narrowPeak的坐标共同,见下图。
- ${filename}_summits.bed
BED格局的文件,包括peak的summits(峰顶)方位 1.染色体号 2.峰的峰顶开端位点(留意是峰顶,不是峰,他只是一个点) 3.峰的峰顶开端位点(因而二者就差一个碱基的方位) 4.峰顶(样本名 _peak_1的后缀之类的) 5.-log10pvalue
- 留意:excel里的开端坐标需求减1才与narrowPeak的坐标共同,见下图。
-
别的,需求告诉你的是,bed文件中第2列中的峰顶开端位点是怎么核算的?
bed文件中第2列(峰的峰顶开端位点) = xls第二列 xls第十列 -1。或许↓↓↓↓
bed文件中第2列(峰的峰顶开端位点) = narrowPeak第二列 narrowPeak第十列 -
总览比较:
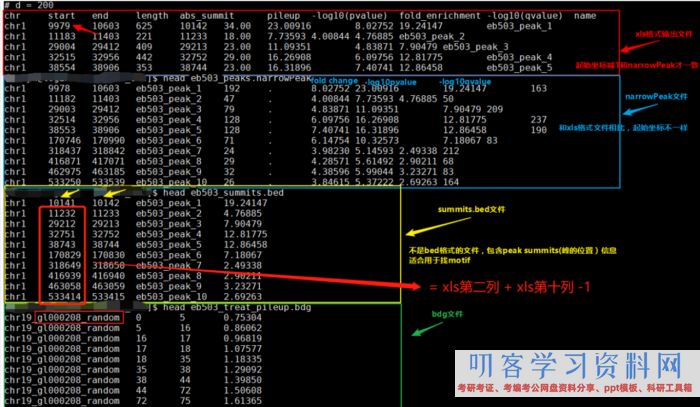 bed,narrowPeak,bdg,xls四种类型输出文件的比较
bed,narrowPeak,bdg,xls四种类型输出文件的比较 -
FRiP_score的核算【官方介绍】
简略来说,FRiP_score说的便是核算落在Peaks里边的reads数量与一切比对到基因组上reads数量的比值。能够作为断定样本测序质量的一个指标。
Reads=$(bedtools intersect -a ${filename}.last.bed -b ${filename}_peaks.narrowPeak |wc -l|awk \'{print $1}\')
totalReads=$(wc -l ${filename}.last.bed|awk \'{print $1}\')
echo ${Reads} ${totalReads} ${filename} \'FRiP_score:\' $(echo \"scale=2;100*${Reads}/${totalReads}\"|bc)\'%\'
- 官网上说ATAC-Seq的FRiP score should be >30%(https://www.encodeproject.org/atac-seq/)我猜是假如大多数reads都不在peaks区域,阐明测序质量或许试验作用不是太好,因为酶切切开的便是peaks区域,太少不是就偏离试验意图了嘛
-
SPOT_score的核算【官方介绍】【软件装置】
简略来说,SPOT_score便是在FRiP_score的根底上增加了测序深度的条件约束,比方FRiP是核算全局的reads,而SPOT只核算测序深度为30×的reads,大于30×的reads不再参加核算。现在我还在探究中怎么核算。
二、高档剖析
所谓的高档并不高大上,做了有或许也不能让你的文章提升层次,望周知!
1. IGV可视化
- 前面的bam文件,narrowPeak文件,bed文件都有了,现在还差一个bw文件,因而咱们先跑下面的代码,然后在IGV里边共同看一下
-
所需软件:deeptools和IGV
主要是为生成bw文件用于IGV展现
bamCoverage --bam ${filename}.last.bam -o ${filename}.last.bw
--binSize 10
--normalizeUsing RPGC
--effectiveGenomeSize ${effective_genome_size}
--ignoreForNormalization chrX
--extendReads
注:假如发现没有这个包pysam.libchtslib,直接装置pip install numpydoc
-o: 生成bw文件
binSize: 自行设定掩盖度核算的窗口巨细(bin)
normalizeUsing: 数据准化办法,有三种RPGC,RPKM,CPM,BPM
ignoreForNormalization: 设置你想疏忽的染色体称号
extendReads: 让reads扩展至fragment的巨细
- IGV一览
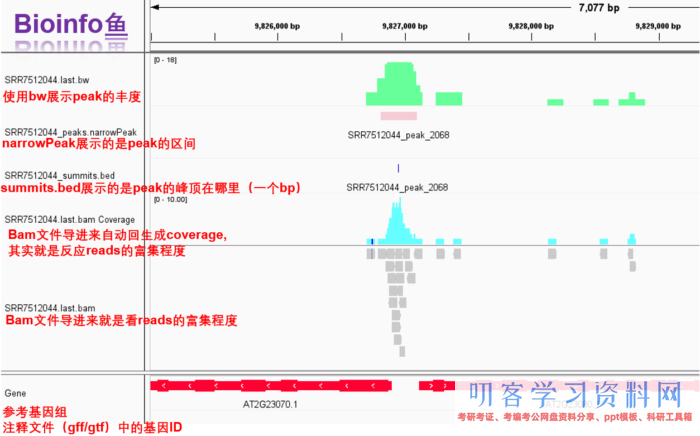
2. TSS/TES 可视化
-
所需软件:deeptools
主要是怎么生成Refseq文件,尽管官网上供给了一些,但总归是不能掩盖一切物种,因而,咱们能够从gff/gtf文件中自己创造。 -
先提取基因的方位信息(不是cds或许exon),根据不同的gff进行修改,但格局始终是
chrom/chromStart/chromEnd/name/score/strandawk -vFS=\'[\\t=;]\' -vOFS=\"\\t\" \\ \'{if ($3==\"gene\") print $1,$4,$5,$10,$6,$7}\' \\ TAIR10.gff|sed \'s/Chr//g\' > refseq.bed #因为tair官网上供给的基因组文件中染色人命名没有Chr,为保持共同,这儿去掉
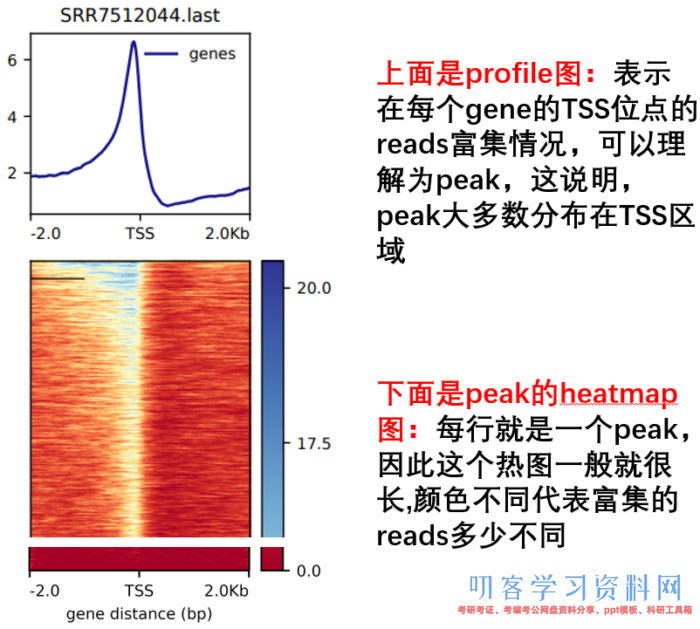
- 然后进行TSS可视化
computeMatrix reference-point --referencePoint TSS -p 15
-b 2000 -a 2000
-R ./refseq.bed
-S *.bw
-o TSS.gz
--skipZeros
--outFileSortedRegions Heatmap1sortedRegions.bed
plotHeatmap -m TSS.gz -out TSS.Heatmap.pdf --plotFileFormat pdf
plotProfile -m TSS.gz -out TSS.Profile.pdf --plotFileFormat pdf --perGroup
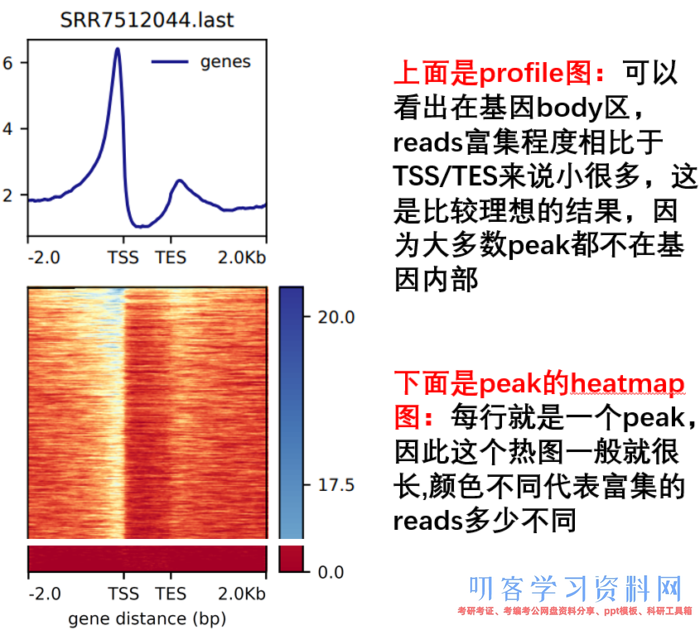
- 然后进行TES 可视化
computeMatrix scale-regions -p 15
-b 2000 -a 2000
-R ./refseq.bed
-S *.bw
--skipZeros -o body.gz
plotHeatmap -m body.gz -out body.Heatmap.pdf --plotFileFormat pdf
plotProfile -m body.gz -out body.Profile.pdf --plotFileFormat pdf --perGroup
- 别的,参阅下面这个图,看每个参数操控的哪里,怎么同时绘制多个等。这是我好久之前总结的,清晰度堪忧
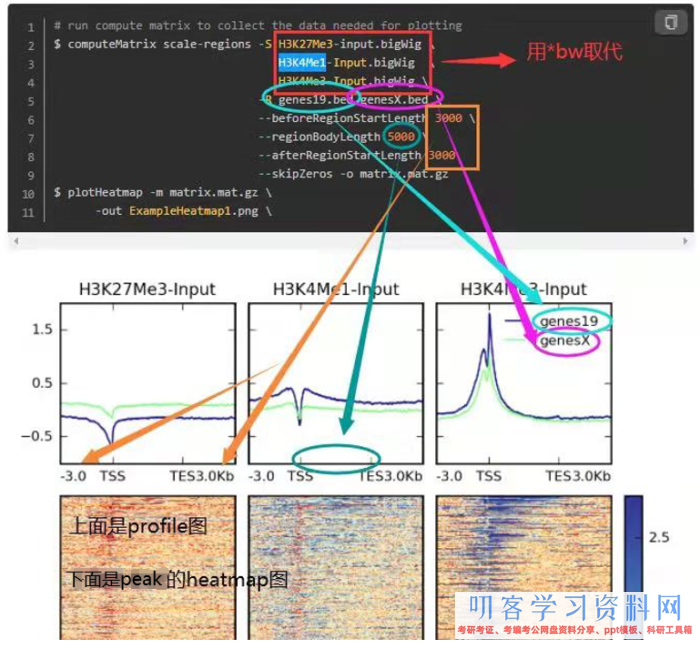
3. 相关性可视化(仍旧是运用deeptools)
- 因为我测验样本少,所以就运用官网的图了哈,但代码是能跑通的,放心运用。
- 先运用 [bw] 文件生成 [npz] 文件。一切样本做相关性,用 [*.last.bw],多个单列出来也行
multiBigwigSummary bins -b *.last.bw -o number.of.bins.npz
- 画相关性热图
plotCorrelation -in number.of.bins.npz --corMethod pearson --skipZeros --plotTitle \"Pearson Correlation of Average Scores Per Transcript\" --plotFileFormat pdf --whatToPlot heatmap --colorMap RdYlBu --plotNumbers -o heatmap_PearsonCorr_bigwigScores.pdf --outFileCorMatrix PearsonCorr_bigwigScores.tab
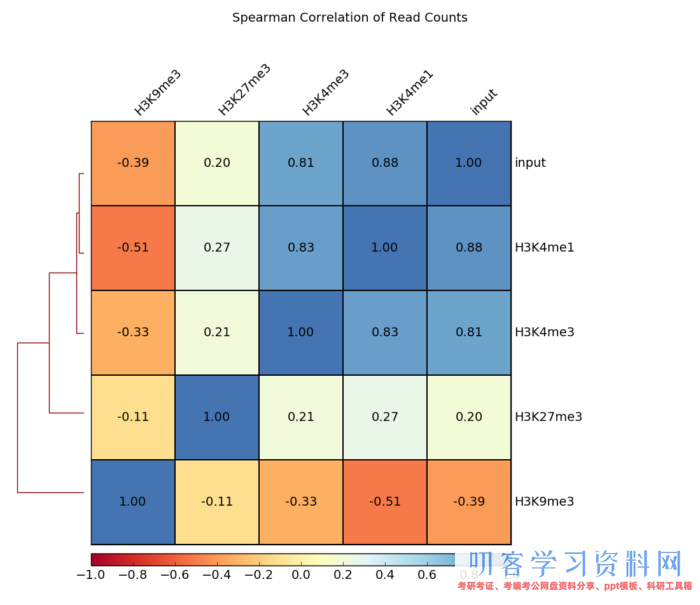
- 画相关性点图
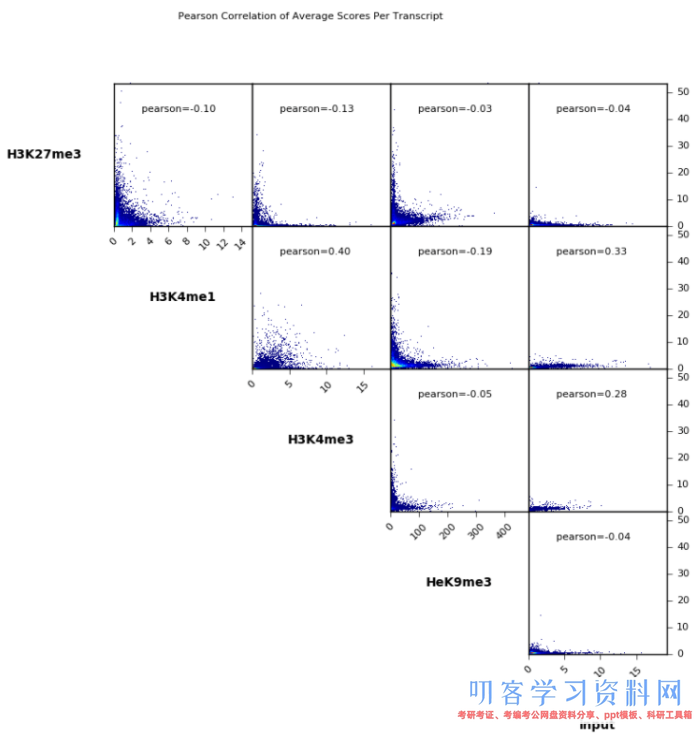
plotCorrelation -in number.of.bins.npz --corMethod pearson --skipZeros --plotTitle \"Pearson Correlation of Average Scores Per Transcript\" --plotFileFormat pdf --whatToPlot scatterplot -o scatterplot_PearsonCorr_bigwigScores.pdf --outFileCorMatrix PearsonCorr_bigwigScores.tab
5. ChIPseeker对peaks进行注释和可视化
-
对peak的注释分为两个部分——结构注释和功用注释
结构注释会将peak所落在基因组上的区域结构注释出来,比方说启动子区域,UTR区域,内含子区域等等。同时,也会将与peak最接近的基因注释出来,非常好用。
功用注释其实并不是对peak进行注释的,而是对peak最接近的基因注释的,因而这部分就和传统的GO/KEGG等剖析千篇一律啦。
5.1 结构注释
5.1.1 预备工作:导入包,没有的装置吧。别的需求运用gff/gtf构建一个TxDb。因为官网上TxDb也不是每一个物种都有(现在共43个),因而咱们自己手动构建。
#if (!requireNamespace(\"BiocManager\", quietly = TRUE))
# install.packages(\"BiocManager\")
#BiocManager::install(\"clusterProfiler\")
library(clusterProfiler)
library(ChIPseeker)
library(GenomicFeatures)
txdb <- makeTxDbFromGFF(\"public/bioinfoYu/genome/tair10/TAIR10.gff\",
format=\"gff\") #也能够运用gtf
keytypes(txdb) #感兴趣的话,能够用以下办法探究txdb都包括了什么内容
keys(txdb)
5.1.2 单个文件的结构注释(不推荐在这一步加OrgDB的内容,没用)
#读入单个summits文件
peaks <- readPeakFile(\"SRR7512044_summits.bed\")
#结构注释
peakAnno <- annotatePeak(peaks,
TxDb=txdb,
tssRegion=c(-1000, 1000))
上面的tssRegion取值有说法的,因为断定\"Promoter\", \"Exon\", \"Intron\", \"Intergenic\"等结构的根据便是根据范围来的。比方界说1000以内的,则在1kbp之内规定为\"Promoter (<=1kb)\",而1k-2k内界说为\"Downstream (1-2kb)\"。但你界说2000以内呢,则在1kbp之内规定为\"Promoter (<=1kb)\",而1k-2k内界说为\"Promoter (1-2kb)\"。尽管,ChIPseeker会标记出来区域,但仍是三思后再取范围。
#注释完,进行可视化,多种图可供挑选 plotAnnoBar(peakAnno) plotDistToTSS(peakAnno) vennpie(peakAnno) plotAnnoPie(peakAnno) #install.packages(\"ggupset\") library(ggupset) upsetplot(peakAnno) #install.packages(\"ggimage\") library(ggimage) upsetplot(peakAnno, vennpie=TRUE) #最终将咱们的注释成果转为数据框,便于检查 df <- as.data.frame(peakAnno) #将注释到的基因提取出来(第14列),用于后续功用剖析 gene <- df[,14]
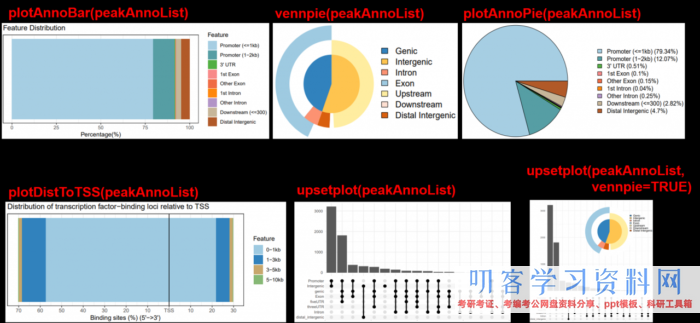
5.1.3 多个文件的结构注释
#一次也能够读入多个summits文件,运用list存储,然后运用lapply注释
files = list(peak1 = \"peaks.bed\", peak2 = (\"peak.bed\"))
peakAnnoList <- lapply(files,
annotatePeak,
TxDb=txdb,
tssRegion=c(-2000, 2000))
plotAnnoBar(peakAnnoList)
plotDistToTSS(peakAnnoList)
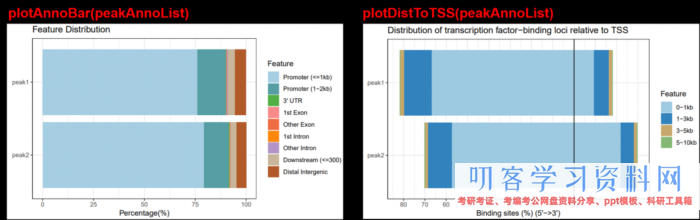
5.2 功用注释
5.2.1 可选支线
因为这儿需求物种的OrgDb注释库。假如你满足走运,研究的物种正好在官网仅供给的20个物种中存在,那就拿来用吧。除了以上这些都好说,还有是id转化,后边再说。
5.2.2 OrgDb下载
本次测验是用拟南芥的样本,我推荐在官网中直接将所需求的的OrgDb库下载到服务器上,这样就不必在R里边重复在网上获取浪费时刻了。比方说拟南芥,就能够下载,这样就把【org.At.tair.db】下载到了服务器或许本地了。
wget http://www.bioconductor.org/packages/release/data/annotation/src/contrib/org.At.tair.db_3.13.0.tar.gz tar -zvxf org.At.tair.db_3.13.0.tar.gz
5.2.3 导入OrgDb
#从本地下载OrgDb库(假如你直接下载到了R的lib里边,就直接library也可) install.packages(\"/public/bioinfoYu/genome/tair10/org.At.tair.db\", repos=NULL) #别的一种办法 #if (!requireNamespace(\"BiocManager\", quietly = TRUE))install.packages(\"BiocManager\") #BiocManager::install(\"org.At.tair.db\") #载入库 library(org.At.tair.db) #感兴趣的话,看看org.At.tair.db包括的都是些啥 #检查包括的列名 keytypes(org.At.tair.db) #检查包括的基因ID keys(org.At.tair.db) #随意挑选一个基因和一些列名,看一看到第是什么东东,不过因为导入了其他包,下面指令报错,我至今还没有解决 select(org.At.tair.db,keys=\"AT1G01010\",columns = c(\"ENTREZID\",\"GO\",\"SYMBOL\"))
5.2.4 ID转化(非有必要)
上面5.1.2现已得到peak附近的基因集【gene】了,假如需求ID转化,便是用clusterProfiler中的bitr函数
#首要,去除版本号,如AT1G50040.1后边的.1,当然咱们的测验集没有,因而不必做
#gene_rm <- gsub(\".[0-9] $\",\"\",gene)
#下面进行ID转化
keytypes(org.At.tair.db)
gene_last <- bitr(geneID = gene,
fromType = \"TAIR\",
toType = c(\"ENTREZID\", \"SYMBOL\", \"GENENAME\"),
OrgDb = org.At.tair.db)
# geneID 需求转化的ID
# fromType 当前ID类型
# toType 转化成什么ID,运用keytypes()检查有哪些类型
# OrgDb 注释数据库
5.2.5 GO功用注释
# 因为keyType不承受数据框类型的数据,因而咱们转化为字符型
genelist <- gene.symbol[,2] #留意我这儿把ENTREZID独自拿出来了,所以下面自然是keyType = \"ENTREZID\",
go_result <- enrichGO(genelist,
keyType = \"ENTREZID\",
OrgDb = org.At.tair.db,
ont = \"BP\", #BP,MF,CC,ALL可选
pAdjustMethod = \"BH\",
qvalueCutoff = 0.05,
readable = FALSE)
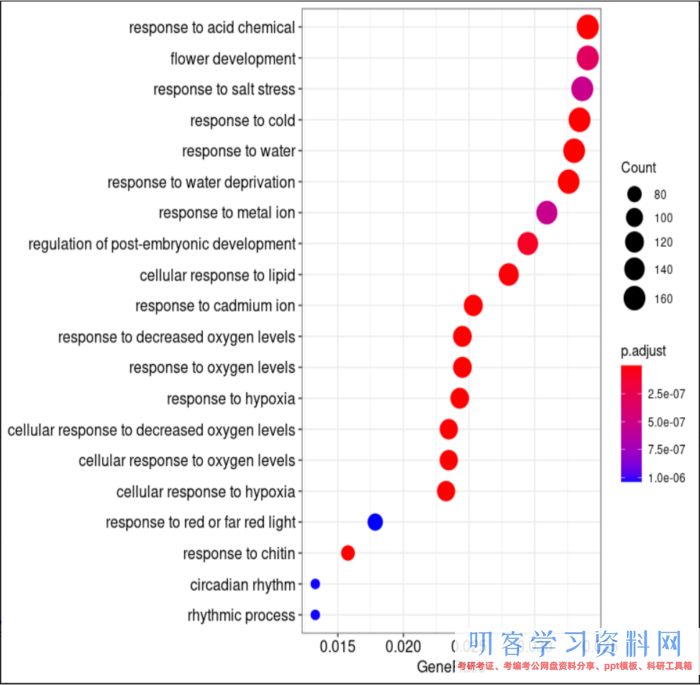
#可视化,showCategory=20调整显现的条目多少
dotplot(go_result, showCategory=20)
#将成果转为数据框,能够挑选导出
goresult <- as.data.frame(go_result)
5.2.6 KEGG通路注释
#geneClusters有必要是数据框类型,ID有必要是ENTREZID类型
class(gene.symbol)
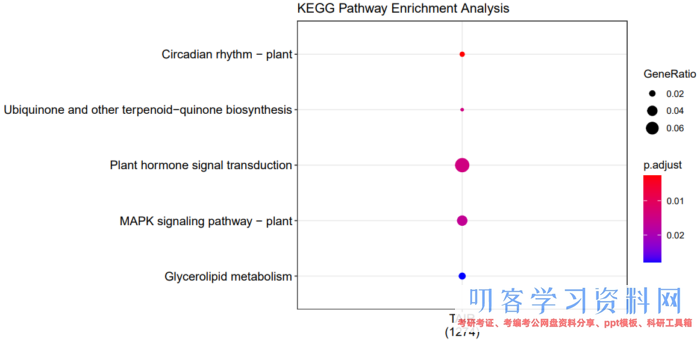
compKEGG <- compareCluster(geneClusters=gene.symbol,
fun = \"enrichKEGG\",
organism = \"ath\", #在 http://www.genome.jp/kegg/catalog/org_list.html 上检查物种简写
pvalueCutoff = 0.05,
pAdjustMethod = \"BH\")
dotplot(compKEGG, showCategory = 20, title = \"KEGG Pathway Enrichment Analysis\")
6. homer寻找motif
- 所需软件:homer【官方网站】
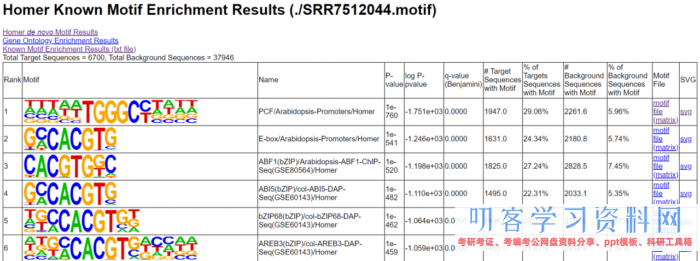
# 提取peaks的方位信息文件
awk \'{print $4\"t\"$1\"t\"$2\"t\"$3\"t \"}\'
SRR7512044_peaks.narrowPeak > SRR7512044.homer_peaks.tmp
# 寻找motif
findMotifsGenome.pl SRR7512044.homer_peaks.tmp
./TAIR10_genome.fa #指定参阅基因组
./SRR7512044.motif #输出文件夹的名字
-size 200 -len 8,10,12
7. DiffBind找差异peak
- 所需软件:DiffBind
首要根据我自己的了解将找差异peak的原理说一下:既然是找差异的peak,那首要就要保证不相同本间peak地点的区间基本共同才能够。因而第一步便是找到多个样本相同的peak区间。第二步,根据这些相同区域的peak开端找差异,这个地方的差异和传统的RNA-Seq找差异基因的原理一模一样,都是对落在峰区间的reads进行计数,数量不同的话差异就由此而来。但软件都帮咱们做了,就不必考虑那么多了
-
我废话真多,其实最费事的便是预备输入数据,下面来看怎样做
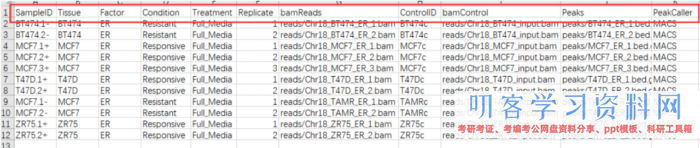
一般状况相下需求包括以下几列:\"SampleID\", \"Tissue\", \"Factor\", \"Condition\" ,\"Treatment\",\"Replicate\", \"bamReads\" ,\"ControlID\" ,\"bamControl\" ,\"Peaks\"和\"PeakCaller\"
-
但咱们做ATAC-Seq的哪里有什么
control,controlID,bamControl,因而删掉,而\"Tissue\", \"Factor\", \"Condition\" ,\"Treatment\",\"Replicate\",标明的都是分组的意思,因而我保存了两个\"Factor\",\"Replicate\",最终csv文件的姿态如下: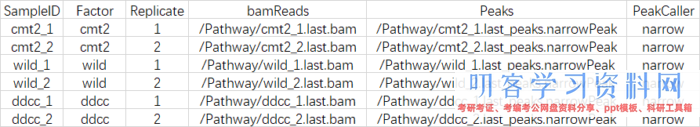
SampleID:不能有重复
Factor:进行分组,后边要做差异剖析的根据
Replicate:重复的编号
bamReads:最终生成的bam文件。全途径 bam文件名
Peaks:macs2生成的narrowPeak文件。全途径 narrowPeak文件名
PeakCaller:阐明peak类型,运用narrowPeak文件则为narrow。运用xls文件,则用macs。运用bed文件,则用bed
- 开端正题
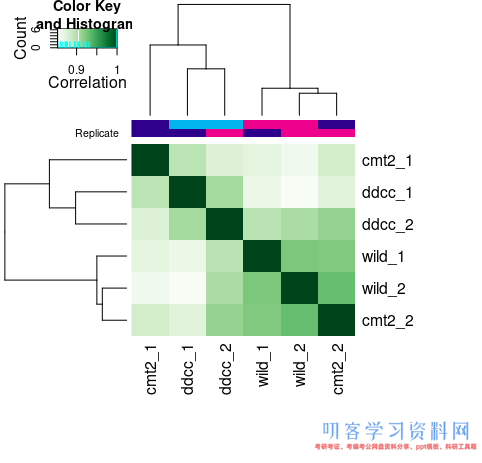
# 装置包 if (!requireNamespace(\"BiocManager\", quietly = TRUE))install.packages(\"BiocManager\") BiocManager::install(\"DiffBind\") library(DiffBind) #导入数据,dba函数会将会将bam文件一同导入,因而csv中的途径一定要精确 tamoxifen <- dba(sampleSheet=\"atDiff.csv\") #检查样本间的相关性 plot(tamoxifen)
#核算每个样本中的reads数 tamoxifen <- dba.count(tamoxifen) #简略检查核算成果 info <- dba.show(tamoxifen) libsizes <- cbind(LibReads=info$Reads, FRiP=info$FRiP, PeakReads=round(info$Reads * info$FRiP)) rownames(libsizes) <- info$ID libsizes
#核算成果
LibReads FRiP PeakReads
cmt2_1 6720462 0.29 1948934
cmt2_2 7747491 0.33 2556672
wild_1 7078309 0.36 2548191
wild_2 8030752 0.34 2730456
ddcc_1 6370232 0.30 1911069
ddcc_2 6956429 0.35 2434750
<pre\">#默许根据测序深度对数据进行标椎化 tamoxifen <- dba.normalize(tamoxifen) norm <- dba.normalize(tamoxifen, bRetrieve=TRUE) norm #检查进行标准化的文库巨细(library-size),其实便是各样本文库巨细总和的平均值,你能够核算验证一下 normlibs <- cbind(FullLibSize=norm$lib.sizes, NormFacs=norm$norm.factors, NormLibSize=round(norm$lib.sizes/norm$norm.factors)) rownames(normlibs) <- info$ID normlibs
#标椎化成果
FullLibSize NormFacs NormLibSize
cmt2_1 6720462 0.9398443 7150612
cmt2_2 7747491 1.0834724 7150612
wild_1 7078309 0.9898885 7150612
wild_2 8030752 1.1230859 7150612
ddcc_1 6370232 0.8908652 7150612
ddcc_2 6956429 0.9728438 7150612
#分组,格局是表头在最前面,要分的组顺次写在后边,只能两两比较,因而后边只能写两组,但能够多履行几回,都会追加到tamoxifen 中 #分组1,后边运用contrast=1独自检查 tamoxifen <- dba.contrast(tamoxifen, contrast=c(\"Factor\",\"ddcc\",\"wild\")) #分组2,后边运用contrast=2独自检查 tamoxifen <- dba.contrast(tamoxifen, contrast=c(\"Factor\",\"cmt2\",\"wild\")) #当然还可有分组3,4,5等,均能够运用contrast=number独自检查 tamoxifen #按照分组别离进行差异剖析,默许运用DESeq2进行核算,能够挑选method = DBA_EDGER(edgR),或许两个都要method = DBA_ALL_METHODS tamoxifen <- dba.analyze(tamoxifen) dba.show(tamoxifen, bContrasts=TRUE) #检查差异剖析的成果与导出为csv文件 #检查第1组差异剖析的成果 tamoxifen.DB_1 <- dba.report(tamoxifen,contrast=1) tamoxifen.DB_1 write.csv(tamoxifen.DB_1, file=\"diffBind_result.csv\") #检查第2组差异剖析的成果 tamoxifen.DB_2 <- dba.report(tamoxifen,contrast=2) tamoxifen.DB_2 write.csv(tamoxifen.DB_2, file=\"diffBind_result.csv\") ##传统的上下调在找差异peak中称为“Gain”或“Loss” #检查第1组的差异peak数量“Fold>0或<0”操控是“Gain”或“Loss” sum(tamoxifen.DB$Fold>0,contrast=1) sum(tamoxifen.DB$Fold<0,contrast=1) #检查第2组的差异peak数量“Fold>0或<0”操控是“Gain”或“Loss” sum(tamoxifen.DB$Fold>0,contrast=2) sum(tamoxifen.DB$Fold<0,contrast=2)
- 因为我是运用两组进行测验的,所以能够运用contrast=1或contrast=2别离检查,因为都是相同的代码,下面进行可视化的时分,只挑选contrast=1进行可视化
#韦恩图可视化 dba.plotVenn(tamoxifen, contrast=1, bDB=TRUE, bGain=TRUE, bLoss=TRUE, bAll=FALSE) #PCA dba.plotPCA(tamoxifen) #这儿能够运用一切样本进行PCA dba.plotPCA(tamoxifen, contrast=1, label=DBA_FACTOR)#独自对分组1进行PCA #MA plots dba.plotMA(tamoxifen, contrast=1) #Volcano plots dba.plotVolcano(tamoxifen, contrast=1) #Box plots dba.plotBox(tamoxifen, contrast=1) #Heatmap hmap <- colorRampPalette(c(\"red\", \"black\", \"green\"))(n = 13) #对一切样本的一切的差异peak画热图 dba.plotHeatmap(tamoxifen,correlations=FALSE,scale=\"row\",colScheme = hmap) #独自对分组1中一切的差异peak画热图 dba.plotHeatmap(tamoxifen, contrast=1, correlations=FALSE, scale=\"row\", colScheme = hmap) dba.plotHeatmap(tamoxifen,correlations=FALSE,scale=\"row\",colScheme = hmap)
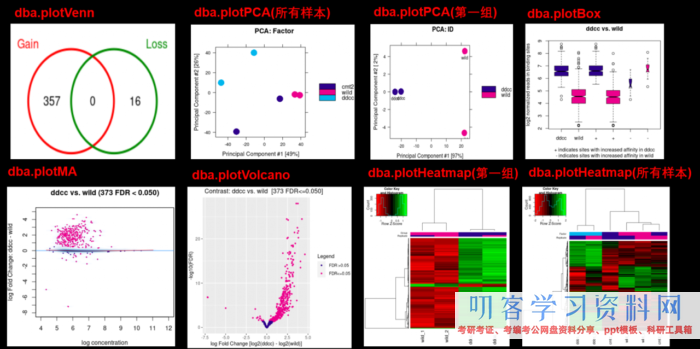
- 想画下面的图,可是一向提示could not find function dba.plotProfile,因而还没有成功,用官方供给的图展现一下吧,大家能画出来能够和我说一声哦。这是【官方文档】能够参阅
#Profiling and Profile Heatmaps dba.plotProfile(tamoxifen)
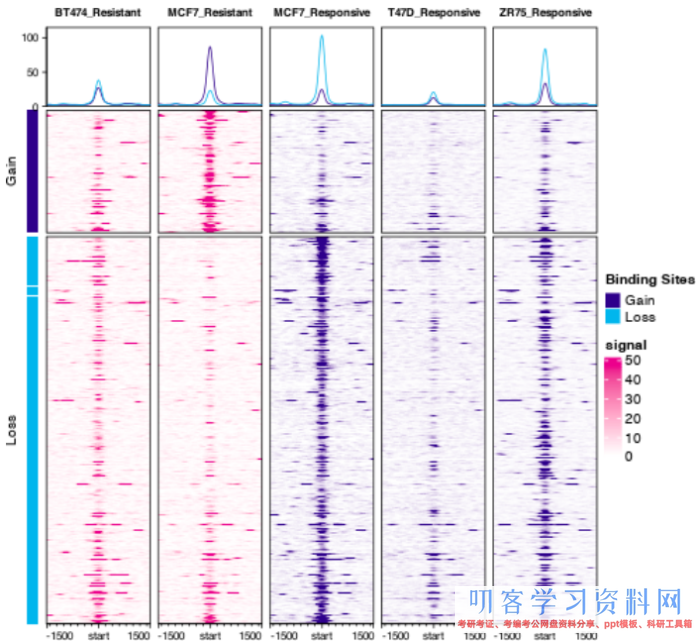
8. footprint剖析
现在我还没有找到运用于一切物种的软件,HINT-ATAC用来做footprint剖析的话,应该只支撑不几个物种,比较鸡肋。ATACseqQC应该是一款不错的软件,最近还在探究
三、ATAC-seq与多组学数据联合剖析
转录因子ChIP-seq:因为大部分转录因子结合的是染色质敞开区域,所以ATAC-seq的Peak或许和转录因子ChIP-seq的Peak存在部分堆叠的状况,而且ATAC-seq得到的Peak长度往往更长,因而ATAC-seq数据和转录因子ChIP-seq数据能够相互验证。转录因子在ChIP-seq中独有的Peak暗示这个转录因子或许是结合在异染色质区域的驱动型转录因子(Pioneer
TFs),驱动型转录因子随后招募染色质重塑复合体以及其它转录因子开端转录。别的,联合剖析现已报导的ChIP-seq数据能够更精确地剖析转录因子的足迹。
组蛋白润饰ChIP-seq:ATAC-seq数据相同能够和组蛋白润饰ChIP-seq数据进行联合剖析,其间转录激活性润饰(H3K4me3,H3K4me1和H3K27ac等)与染色质敞开程度呈正相关,转录按捺性润饰(H3K27me3)与染色质敞开程度呈负相关。联合已知的增强子和启动子之间的相互作用数据也能够帮助构建调控网络。
RNA-seq:ATAC-seq数据能够经过联合剖析RNA-seq数据来发现哪些差异表达的基因是受染色质可及性调控的,进一步能够估测这些差异表达的基因哪些是受敞开染色质中具有motif和footprint的转录因子调控的,因而ATAC-seq与RNA-seq的联合剖析有助于破译基因调控网络和细胞异质性。
- 本文固定链接: https://www.maimengkong.com/zu/1319.html
- 转载请注明: : 萌小白 2023年1月1日 于 卖萌控的博客 发表
- 百度已收录
