近年来,越来越多的研究表明,单一或者少数参考基因组不能涵盖整个种群全部的遗传信息,所以,构建物种的泛基因组成了当前基因组学研究的热点和前沿。本篇文章,小编和大家一起来了解一下泛基因组的构建方式、泛基因组的热点研究内容。
一、泛基因组的构建策略
泛基因组构建主要分为以下三种方式[1]:
(1)选取一个参考基因组,各个材料的测序reads分别比对,将未比对上的reads进行组装,通过“比对-组装”的迭代过程构建;
(2)所有材料进行基因组从头组装,通过从头组装的基因组进行相互比较来构建泛基因组;
(3)基于变异图的泛基因组构建;
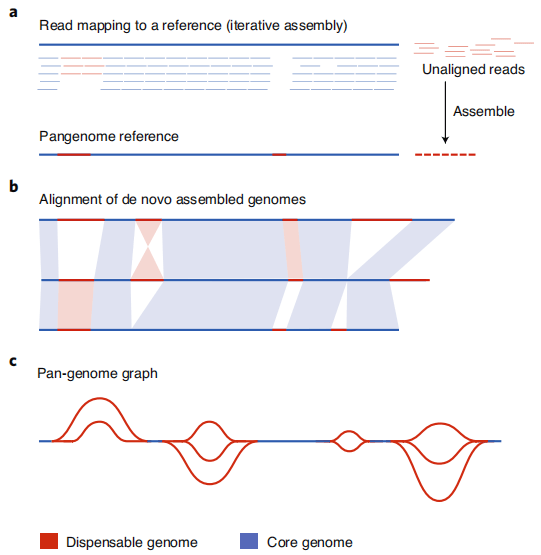
图1 构建泛基因的三种策略
当前第一种和第二种策略使用的比较广泛,例如,番茄的100泛基因就是采用的第一种策略构建的泛基因组[4];27个大豆项目、7个拟南芥,14个果蝇等都是采用第二种策略[3,5,6]。大豆的泛基因组也初步尝试使用了第三种策略,并进行了GWAS关联分析[3]。第三种策略是理想的构建策略。它基于图论的组装方法,利用有向图把基因组分成若干部分,很好地展示每部分序列在泛基因组中的位置关系。相对于第一种和第二种策略,构建泛基因组图的技术和算法尚未成熟,因此目前应用还较少,不过,未来可期。
二、泛基因组的应用与研究
1. 泛基因组揭示遗传变异与表型变异的关系
泛基因组可以结合GWAS数据,有效提高GWAS 的准确性,捕获到更加完整的遗传变异信息。通常,GWAS分析是将个体材料与参考基因组进行比对,然后将得到的 SNP 与表型进行关联分析,当GWAS 分析参考基因组不存在的功能基因引起的表型时,会出现 GWAS 定位区间与实际功能基因之间较大偏差甚至检测不到的情况。但以泛基因组为参考基因组,把结构变异作为基因型数据用于 GWAS 分析,可以有效解决这类因单一参考基因组而导致的信息不全问题。例如,油菜以泛基因组鉴定到的 PAV 为基因型进行GWAS 分析,直接鉴定到基因BnaA9.CYP78A9启动子区存在一段 3.9 kb 的插入,该变异严重影响油菜的角果长和粒重[2]。结构变异相关的GWAS研究更具有新意,也是后续研究的热点。
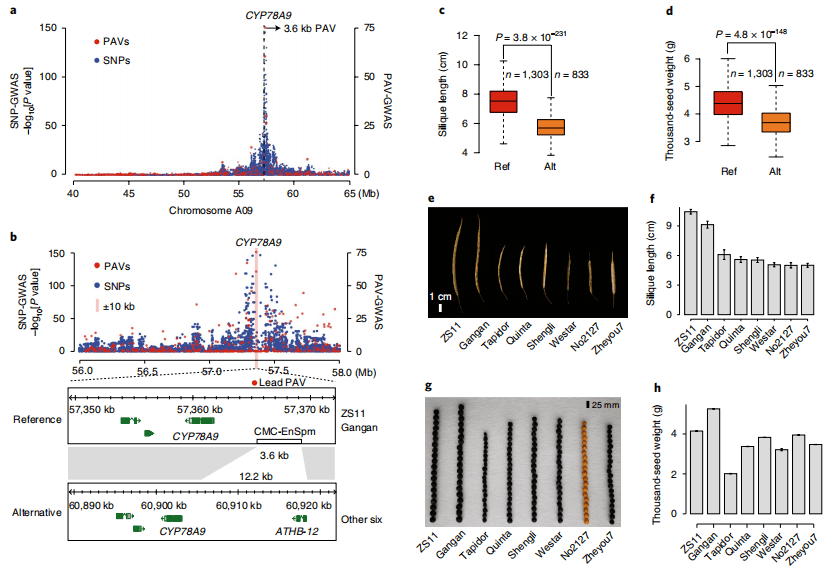
图2 油菜角果长和粒重的GWAS分析
2.泛基因组揭示了结构变异影响基因差异表达
结构变异可能会通过改变基因区或基因侧翼区的序列影响基因的转录调控及表达。如,基因的串联重复可能会引起基因剂量的变化,外显子的复制可能会引起可变剪接,顺式作用元件区的插入或缺失可能会破坏或者改变转录调控因子结合位点。例如,大豆14号染色体上存在一个调控Fe2+/Zn2+转运蛋白的基因SoyZH13_14G179600,一些品种的基因组中,该基因的启动子区存在一个1.4kb的缺失,RNA-seq数据分析显示,该启动子缺失会使转运蛋白表达量降低[3]。
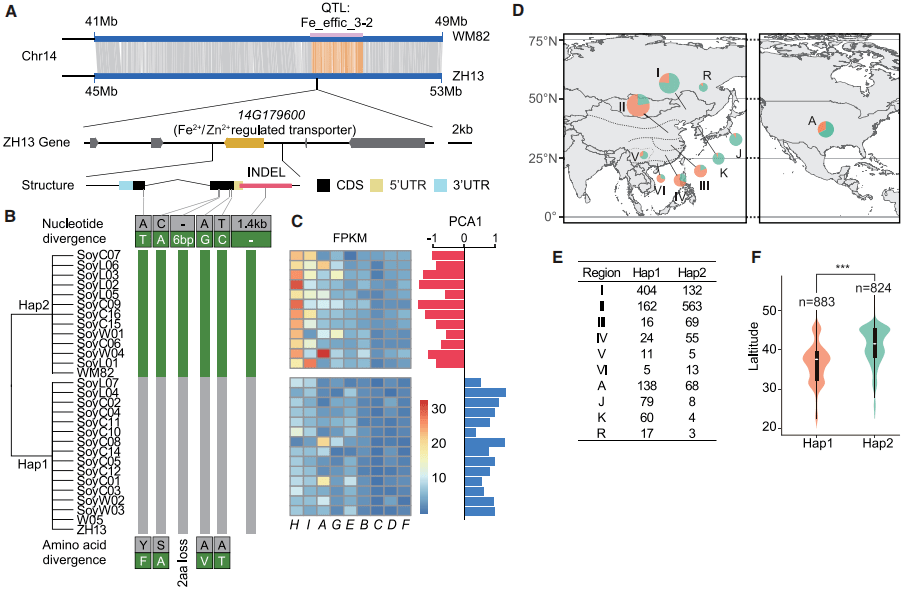
图3 大豆启动子缺失影响基因表达
3.泛基因组揭示了结构变异在种群驯化中的作用
泛基因组拥有群体完整的基因组多样性,可以为植物进化与驯化研究提供更完整、更宽泛的支持。如大豆的种皮颜色是大豆驯化过程中的一个重要表型性状,从野生大豆的黑色种皮演变成栽培大豆的黄色种皮,是因为一个查尔酮合成酶基因发生序列倒置或序列复制后倒置导致了该基因沉默。通过大豆泛基因组单倍体型进化分析,将29份材料分为5个主要的单倍型(H1- H5),并对不同单倍体型驯化时间和顺序进行了研究,剖析了大豆种皮颜色驯化的历史[3]。
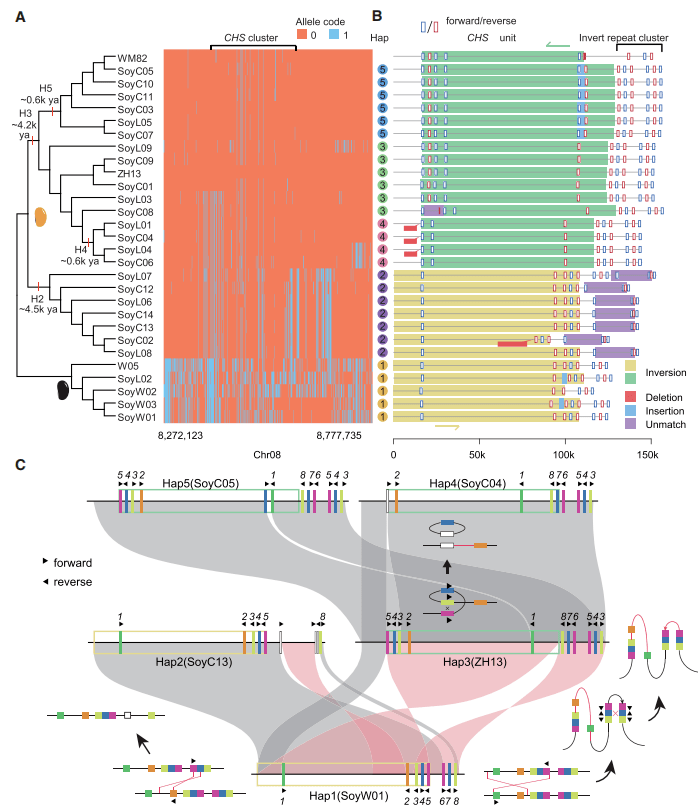
图4 大豆种群驯化
问题和展望
泛基因组是近年来研究的热点, 然而目前泛基因组领域还需解决以下两方面的问题。首先,不同泛基因组研究中,样本量的选择,基因组的测序、组装,泛基因组的构建策略,序列注释,判断基因有无变异(PAV)的方法等都有很大的差别,这就导致了即使同一种物种,不同实验室构建的泛基因组序列及基因有无变异结果等也不能直接进行比较。其次,如何更好地利用泛基因组鉴定出的变异数据,将遗传变异和重要的表型性状关联起来, 是泛基因组应用研究的关键和挑战。
泛基因组作为参考基因组,相对传统的单一参考基因组,能呈现更加全面的种群基因组信息。随着泛基因组的构建方式和分析策略的逐步完善,未来利用泛基因组,会高效地辅助解决功能基因组学研究的难题,为育种家在寻找合适的育种亲本和育种方法上提供支持。
文:LHL
参考文献
[1] Philipp E. Bayer, et al. Plant pan-genomes are the new reference. Nature Plants, 2020.
[2] Jia-Ming Song, et al. Eight high-quality genomes reveal pan-genome architecture and ecotype differentiation of Brassica napus. Nature Plants, 2020.
[3] Yucheng Liu, et al. Pan-Genome of Wild and Cultivated Soybeans.Cell,2020.
[4] Michael Alonge, et al. Major Impacts of Widespread Structural Variation on Gene Expression and Crop Improvement in Tomato. Cell, 2020.
[5] Mahul Chakraborty, et al. Structural variants exhibit widespread allelic heterogeneity and shape variation in complex traits. Nature Communications, 2019.
[6] Wen-Biao Jiao, et al. Chromosome-level assemblies of multiple Arabidopsis genomes reveal hotspots of rearrangements with altered evolutionary dynamics. Nature Communications, 2020.
- 本文固定链接: https://www.maimengkong.com/kyjc/845.html
- 转载请注明: : 萌小白 2022年2月11日 于 卖萌控的博客 发表
- 百度已收录
