Python作为胶水语言的强大之处已经不需要我过多描述了,它在机器学习领域、大型网站搭建框架等都有非常广泛的应用,比如TensorFlow的python接口,基于Python的Tornado和Django框架,国内的知乎和豆瓣、以及Youtube大部分是基于这些python的框架。当然最为重要的是它在生物信息领域的应用也较为广泛,比如基于扩增子的测序,还有一些全基因组测序等后期分析的软件很多都是基于python的。Python写成的Qiime软件,在扩增子测序的序列处理方面,几乎是行业内的标杆。不仅如此,Python在数学计算,数据模拟等工程领域和matlab可以说是不相上下,虽然matlab很多专业的工具箱目前为止还是不可代替的,但是一提到matlab那个庞然大物,我就提不起兴趣了。Python本身非常小巧,强大之处在于依赖大量的第三方库,非常著名的包括numpy、scipy、matplotlib、pandas等。这几个第三方库的官方文档都有将近两千页,相信为了用python的强大功能,很多工科的小伙伴们没少花时间看这些文档。Python对于完全没有计算机基础的同学来说起点稍高,不仅需要基础的面向对象编程的基础思维,还需要能够熟练掌握语法,更具有挑战性的是,强大的第三方库都有各自的数据类型和对象属性以及方法,实际上每次学习一个新的库,就需要重新去熟悉库函数的用法,输入和输出数据的属性、以及支持的方法,消耗大量的时间。关于如何学习python安装、基础语法、版本选择、第三方库安装等就不再赘述了,可以参考基础的教程。
今天我们要讨论的是如何通过Python画出和在R中用ggplot2画出的同样优美的统计图。科研作图在科研工作中是必不可少的,而且比如Nature、Science、PNAS和Cell这样的杂志对作图的要求非常高。不仅要美观,而且颜色搭配要合适,字体和布局也要合适。先看两篇最新发表在nature和PNAS上的,都是和基因研究相关的论文https://www.nature.com/articles/nature24621、http://www.pnas.org/content/114/38/10166.full这两篇论文都是采用Python作图,第一篇文章前期的数据分析和可视化python代码总共加起来上万行,里面所有的图和数据都在文章的GitHub库中提供了,大家可以参考学习,这也是这两年的文章中能够看到的比较完整的Python作图的code了。不难看出这些nature论文的图主要特点就是颜色看起来舒服,字体大小合适,如果你随便找一篇普通的二三流杂志的论文看一看,区别很明显。
首先介绍一下三个主要的画图包,第一个就不用说了,从matlab的plot库移植过来的,学习的时间成本比较高,功能的确很强大。第二个包是几年前创建,也是由于matplotlib过于复杂,这个包的作者估计也是用够了,索性写了这样的一个包,但是不得不说,在图形的主题,坐标轴标签和ticks的修改这方面还比较缺乏,官方的文档没有解释清楚,和它基于的matplotlib相比已经是前进了一大步了,直到今年四五月份,plotnine的作者将R语言ggplot2的API在python中重新实现,实际上是将ggplot2的绝大多数功能复制过来了,语法上有些差异,比如aes(x=cyl)修改为aes(x='cyl'),还有一些细微的差别,目前plotnine还在更新之中,相信完整的版本完全可以100%替代ggplotw2.
接下来说说R语言作图,这两年生物信息学的爆炸,R语言由于在多元统计方面的优势,以及比较简单的学习过程,受到很多人的青睐,Hadley的ggplot2风靡一时,好多文章纷纷使用,主要是因为基于Grammar of Graphics的思想,将画图简单化、符合人们的思维方式。然而python的作图库matplotlib则完全相反,虽然非常强大,但是学习起来难度很大。给个code对比就可以发现:
importmatplotlib# Say, "the default sans-serif font is COMIC SANS"matplotlib.rcParams['font.sans-serif'] ="Helvetica"# Then, "ALWAYS use sans-serif fonts"matplotlib.rcParams['font.family'] ="sans-serif"importnumpyasnpimportmatplotlib.pyplotaspltfrommatplotlib.tickerimportMaxNLocatorfromcollectionsimportnamedtuplen_groups =5means_men =(20, 35, 30, 35, 27)std_men =(2, 3, 4, 1, 2)means_women =(25, 32, 34, 20, 25)std_women =(3, 5, 2, 3, 3)plt.figure(num=3, figsize=(7, 4.5),)fig, ax =plt.subplots()index =np.arange(n_groups)bar_width =0.35opacity =1error_config ={'#999999': '0.3'}rects1 =ax.bar(index, means_men, bar_width, alpha=opacity, color='#0F73B0', yerr=std_men, error_kw=error_config, label='Men')rects2 =ax.bar(index +bar_width, means_women, bar_width, alpha=opacity, color='#D45E1A', yerr=std_women, error_kw=error_config, label='Women')ax.set_xlabel('Group',fontsize=14)ax.set_ylabel('Scores',fontsize=14)ax.set_xticks(index +bar_width /2)ax.set_xticklabels(('amoa', 'amya', 'mcra', 'mcraA', 'mcraD'),fontsize=14)plt.yticks(fontsize=14)ax.grid(linewidth=0.25, linestyle='--')ax.legend(fontsize=14)plt.savefig("plt4_menwomen_1.pdf",bbox_inches="tight")plt.show()
这是画的图(上传的高清的被压缩了):
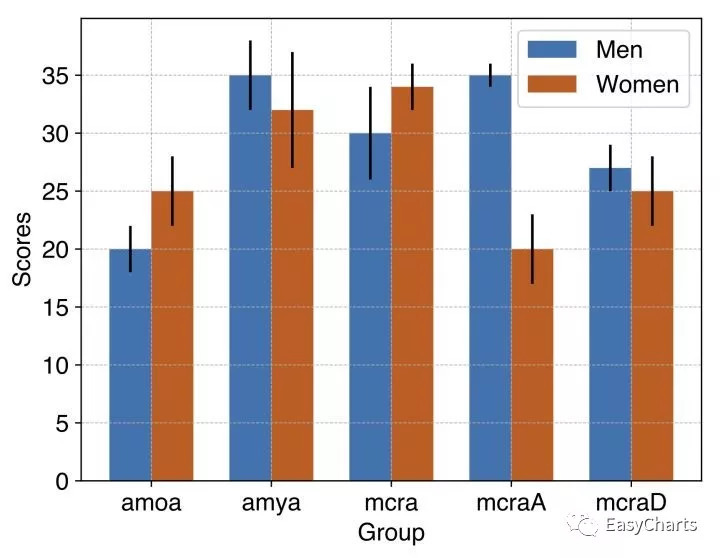
我们可以发现几十是作最简单的bar plot,matplotlib需要很多元素去修饰这样的一个图,而且每次修改作图参数都非常繁琐,一不小心就容易出错,相反,如果我们用plotnine来作图的话就相当简单,读取数据之后直接作图,由于根据图像的语法中的规则,规定图的类型、分类方式、颜色、坐标轴的名称和字体大小、以及图例的位置等:
importpandasaspdimportnumpyasnpfromplotnineimport*fromplotnine.dataimport*ToothGrowth_summary =pd.read_csv('ToothGrowth_summary.csv',header=0,index_col=0)q5 =(ggplot(ToothGrowth_summary, aes(x='factor(dose)', y='len', fill='supp')) +geom_bar(stat="identity", position=position_dodge()) +theme_classic()+geom_errorbar(ToothGrowth_summary,aes(ymin='ymin', ymax='ymax'), width=.2, position=position_dodge(.9))+theme(legend_position=(0.3,0.8))+theme(axis_text=element_text(size=14,color='black',family='sans-serif'),axis_title=element_text(size=14,color='black',family='sans-serif'),legend_text=element_text(size=14,color='black',family='sans-serif'))+labs(x='dose (mg)',y='length (cm)',fill='')+theme(legend_background=element_rect(alpha=0))+scale_y_continuous(expand=(0, 0, .1, 0))+labs(fill='')+scale_fill_manual(values=['#D45E1A', '#0F73B0']))q5.save('Tooth_boxploterrorbar.pdf',width=4, height=5.5)
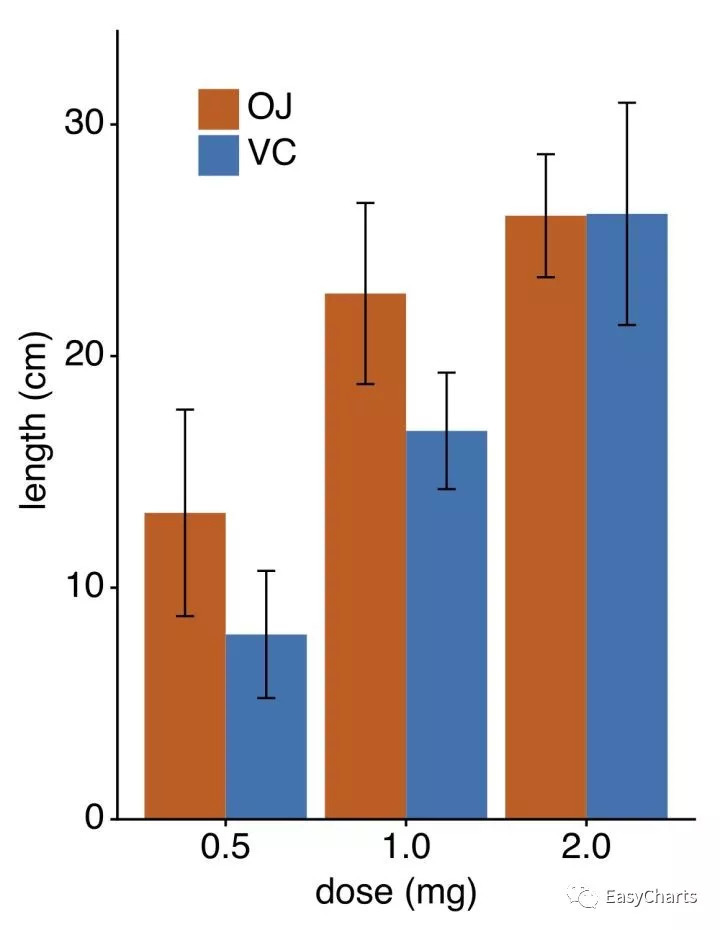
虽然代码长度差不多,但是理解起来却非常直观,而且对许生物统计来说这种分组数据有误差存在,还需要作方差分析和事后比较的的时候,用matplotlib的方式就显得很累赘,而这种方式就非常容易理解,同样的如果采用Seaborn这个包的话,也相当简单:
importmatplotlib.pyplotaspltimportseabornassnssns.set_style('ticks', {'font.family':'sans-serif', 'font.serif':'Helvetica'})# Load the example Titanic datasettitanic =sns.load_dataset("titanic")print(titanic.head())# Draw a nested barplot to show survival for class and sexg =sns.factorplot(x="class", y="survived", hue="sex", data=titanic, kind="bar", palette="muted")# g.despine(left=True)g.set_ylabels("survival probability")g.set_xlabels("Cagegories")plt.show()
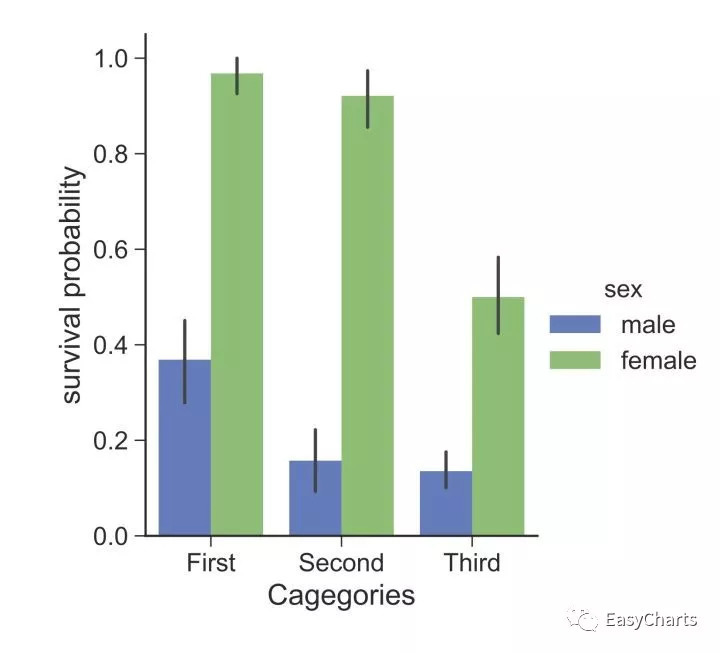
Seaborn是Stanford的一个同学写的后来包放到PyPi上,在plotnine今年公布之前主要python的流行作图就是使用这个包,前面提到的两篇论文也是主要采用的这个包的作图函数和风格。
接下来是散点图,使用matpotlib作散点图相对而言简单一些,但是如果需要分组,同样的会面临复杂的配置:
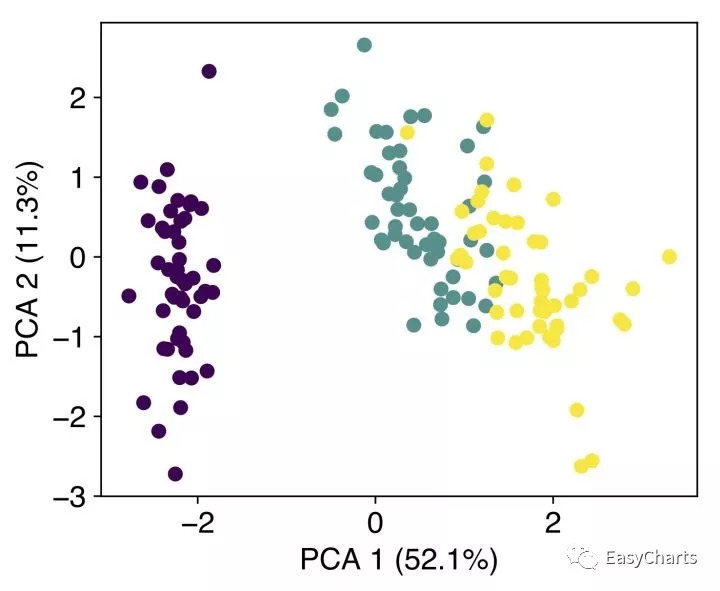
importmatplotlib# Say, "the default sans-serif font is COMIC SANS"matplotlib.rcParams['font.sans-serif'] ="Helvetica"# Then, "ALWAYS use sans-serif fonts"matplotlib.rcParams['font.family'] ="sans-serif"importnumpyasnpimportmatplotlib.pylabaspylabimportmatplotlib.pyplotaspltparams={ 'axes.labelsize': '14', 'xtick.labelsize':'14', 'ytick.labelsize':'14', 'lines.linewidth':1, 'legend.fontsize': '14', 'figure.figsize': '6, 6'}pylab.rcParams.update(params)importnumpyasnpfromsklearn.datasetsimportload_irisfromsklearn.preprocessingimportscaleimportscipyimportmatplotlib.pyplotaspltimportpandasaspdfromsklearnimportdecomposition# Load Iris datadata =load_iris()x =data['data']y =data['target']Se_targets =pd.Series(load_iris().target, index =["iris_%d"%i fori inrange(load_iris().data.shape[0])], name ="Species")print(y)# Since PCA is an unsupervised method, we will not be using the target variable y#scale the data matrix x to have zero mean and unit standard deviation. The rule of thumb is that if all your columns are measured in the same scale in your data and have the same unit of measurement, you don’t have to scale the data. This will allow PCA to capture these basic units with the maximum variation:x_s =scale(x,with_mean=True, with_std=True, axis=0)# Calculate correlation matrixx_c =np.corrcoef(x_s.T)# Find eigen value and eigen vector from correlation matrixeig_val,r_eig_vec =scipy.linalg.eig(x_c)#print "PCP': "#print r_eig_vec.dot(x_c).dot(r_eig_vec.T);# Select the first two eigen vectors.w =r_eig_vec[:,0:2]#Project the dataset in to the dimension from 4 dimension to 2 using the right eignen vectorx_rd =x_s.dot(w)fig =plt.figure(1, figsize=(5, 4))# Scatter plot the new two dimensionsplt.scatter(x_rd[:,0],x_rd[:,1],marker='o',c=y)plt.xlabel("PCA 1 (52.1%)")plt.ylabel("PCA 2 (11.3%)")plt.savefig("pca_plot_012.pdf",bbox_inches="tight")plt.show()## using sklearnfig =plt.figure(2, figsize=(5, 4))pca =decomposition.PCA(n_components=2)pca.fit(x)x =pca.transform(x)df=pd.DataFrame(x,columns=['PC1','PC2'],index=Se_targets.index)df.to_csv('iris_pca_result.csv',header=True, index=True)print(x)plt.scatter(x[:,0],x[:,1],marker='o',c=y)#plt.show()# using ggplot2fromplotnineimport*fromplotnine.dataimport*df['group']=Se_targets# print(df)p1 =(ggplot(df, aes(x='PC1', y='PC2',color='factor(group)',shape='factor(group)'))+theme_matplotlib()+geom_point(size=3)+scale_shape_manual(values=['^','o','s'])+scale_color_manual(values=['#FC0D1C', '#1AA68C', '#F08221'])+theme(axis_text=element_text(size=14,color='black',family='sans-serif'),axis_title=element_text(size=14,color='black',family='sans-serif'),legend_text=element_text(size=12,color='black',family='sans-serif'))+theme(legend_position=(0.8,0.25),legend_background=element_rect(alpha=0))+guides(color=guide_legend(nrow=3),shape=guide_legend(nrow=3))+labs(color='',shape='') )p1.save('iris_pcoa_plotnine.pdf',width=6, height=5.5)
如果采用plotnine作图的话和matplotlib相比基本可以达到相同的效果,精确控制每一个图像元素,上图图为matplotlib的结果,下图为plotnine的效果。
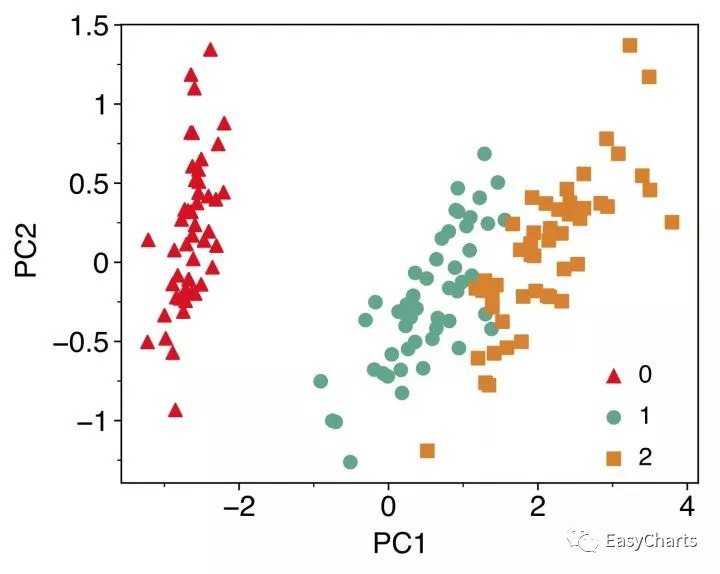
经过以上几个例子不难看出,无论plotnine还是seaborn都是对matplotlib更高层次的封装,调用起来更加人性化而且数据组织的方式符合我们实验中得到的形式,不需要再去转换,因而方便使用。Seaborn支持非常少的封装,比如如果需要改变图例的位置,还是需要通过matplotlib.pyplot模块,我们以这个violin plot为例:
importmatplotlib# Say, "the default sans-serif font is COMIC SANS"matplotlib.rcParams['font.sans-serif'] ="Helvetica"# Then, "ALWAYS use sans-serif fonts"matplotlib.rcParams['font.family'] ="sans-serif"importmatplotlib.pylabaspylabparams={ 'axes.labelsize': '14', 'xtick.labelsize':'14', 'ytick.labelsize':'14', 'lines.linewidth':1, 'legend.fontsize': '14', 'figure.figsize': '6.5, 6'}pylab.rcParams.update(params)# using seaborn to plot resultsimportmatplotlib.pyplotaspltimportseabornassnssns.set_style('ticks', {'font.family':'sans-serif', 'font.serif':'Helvetica'})# Load the example tips datasettips =sns.load_dataset("tips")# Draw a nested violinplot and split the violins for easier comparisong=sns.violinplot(x="day", y="total_bill", hue="sex", data=tips, split=True, inner="quart", palette={"Male": '#1AA68C', "Female": '#F08221'})plt.legend(loc=(0.02,0.85))plt.show()
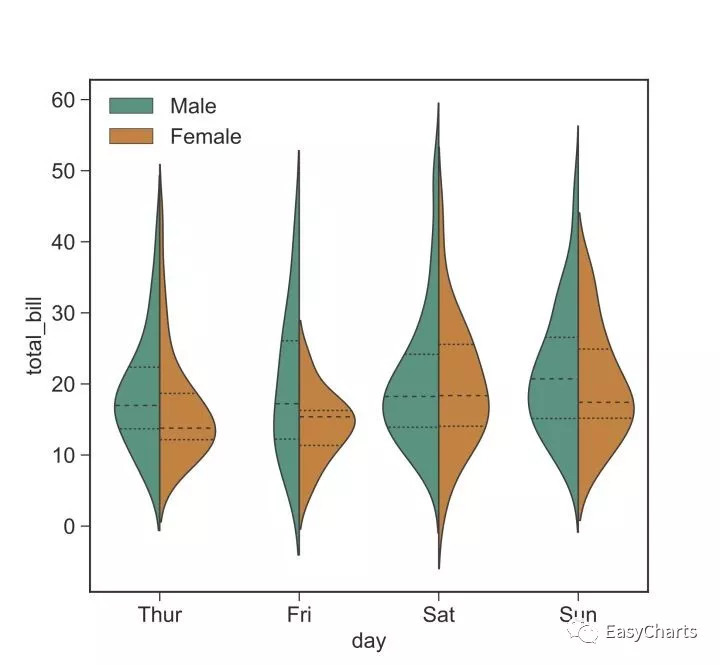
如果需要在plotnine中改变legend的位置则可以非常方便,使用theme(legend_position=(0.2,0.8))即可。还有如果需要在图中添加文字,seaborn则非常不方便。而使用plotnine中的geom_text()函数则可以轻松完成。Seaborn的一些主题和plotnine相比还是很相似的,但是缺点就是相对死板,修改绘图参数不容易。这一点上plotnine继承了ggplot2的精髓,修改绘图参数直观而简单。
接下来看看画图的一些基本要求,比如颜色的选择等,颜色对于一个图的效果非常重要,比如莫兰蒂色系可以大面积用色,而那些饱和度较高的颜色则不适合大面积用色。下面的图本来的颜色是基础的红黄蓝绿,非常刺眼,我修改之后则明显优雅很多:
在mac上推荐大家使用Sipcolor的软件,将Rcolorbrewer等配色方案中的颜色都记下来,以供后来使用,常用的有莫兰迪色系等,可以找一本设计师配色的书籍学习更多的适合作图的颜色。无论是这种柱状图,需要大量用色,还是以下的线图,仅仅需要少量的用色,都需要合理考虑颜色的使用。
ggplot2的功能非常强大,因此plotnine同样可以完成复杂的图,下面的六边形图和柱状图的结合,可以通过plotnine一次完成。
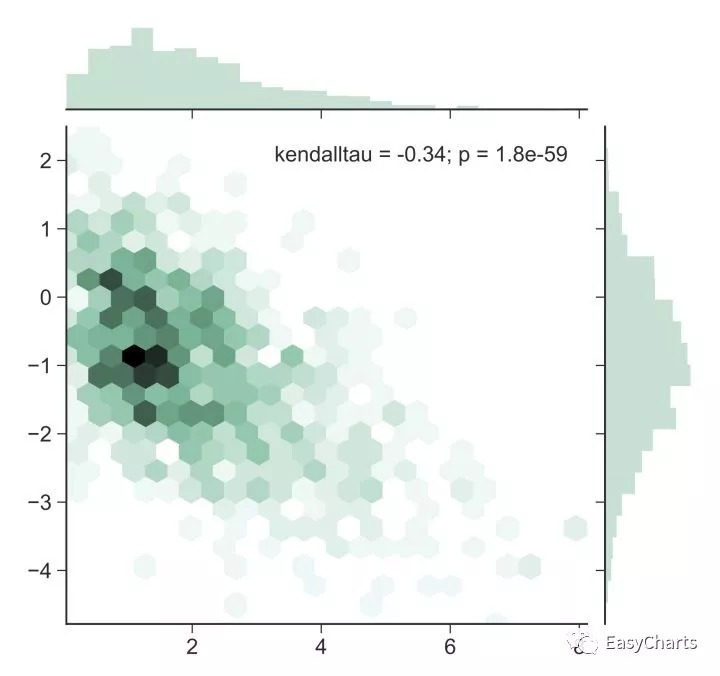
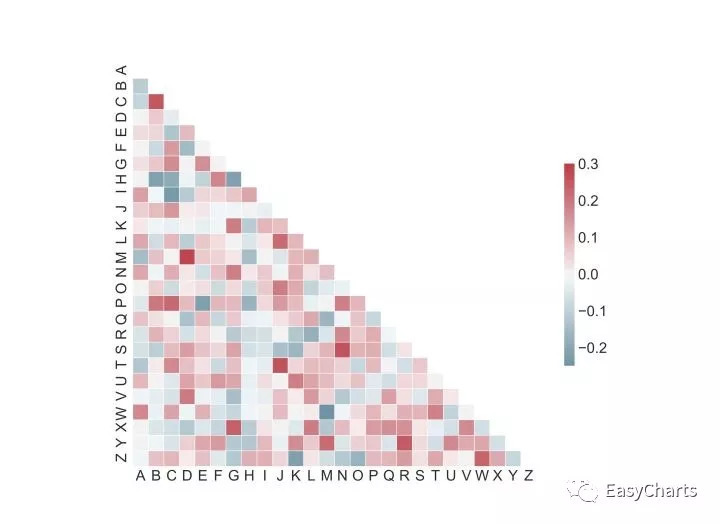
同样的相关性图也可很快完成,通过plotnine和seaborn都可以。
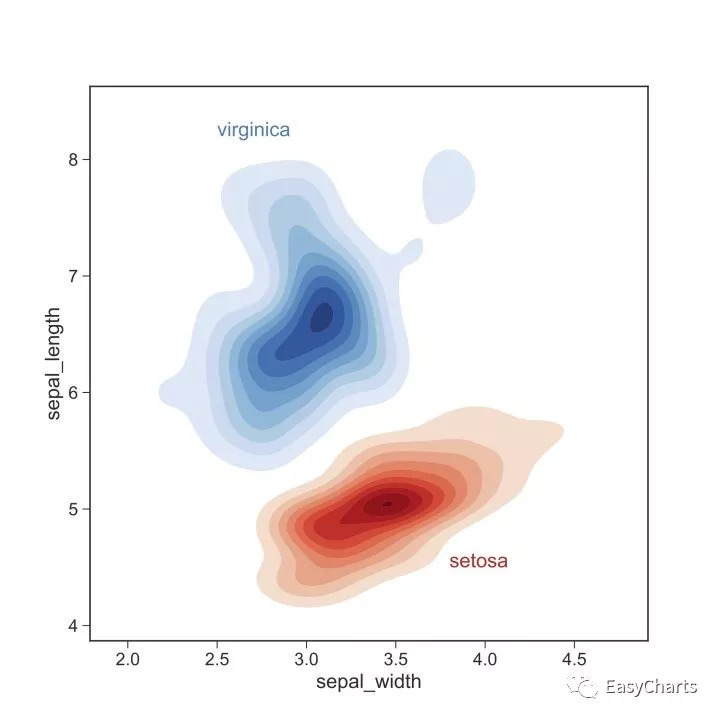
kde图也可以通过两个包画出来。
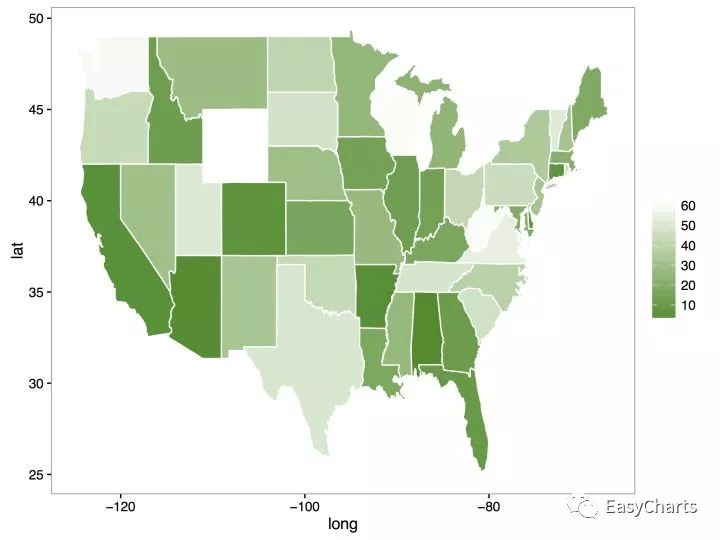
同样的geom_polygon()可以创作地图,但是新版的plotnine功能还没有更新。这是用R创作的美国地图。
密度分布图,code也是相当简单。
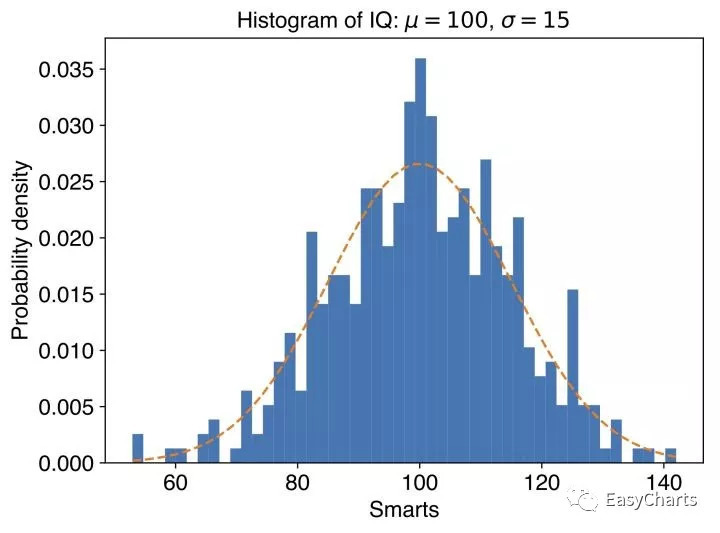
值得一提的是,将density plot和分布图结合起来,在R中可以轻易实现,plotnine还不能完全做到。
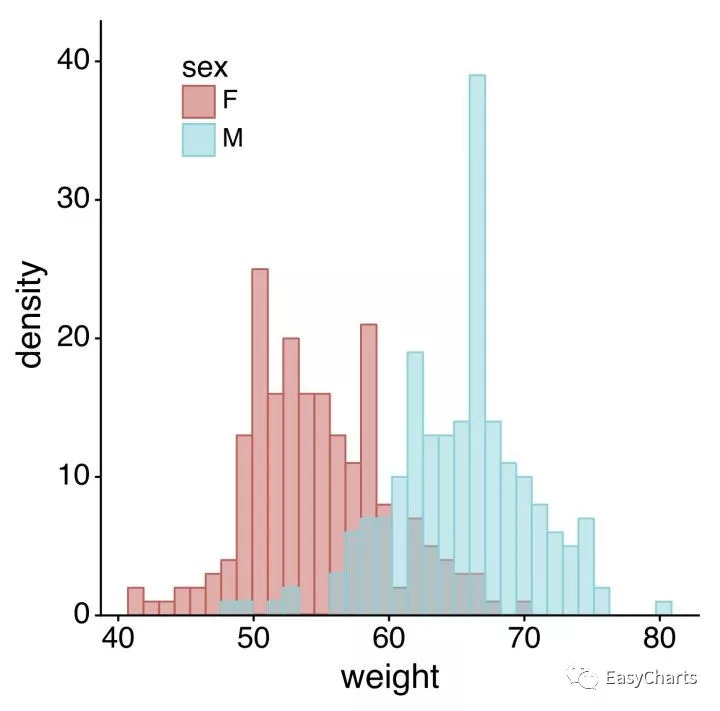
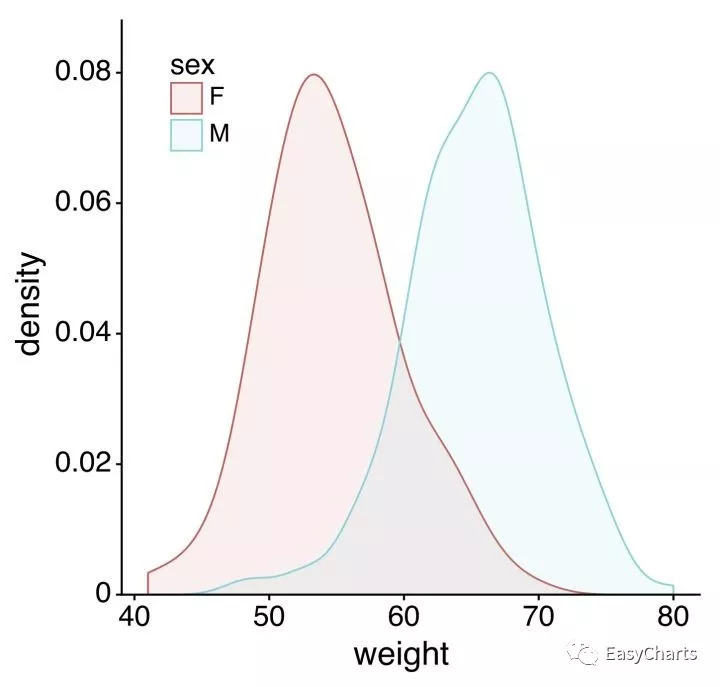
同样的,分组画图使用plotnine,非常简单优美:
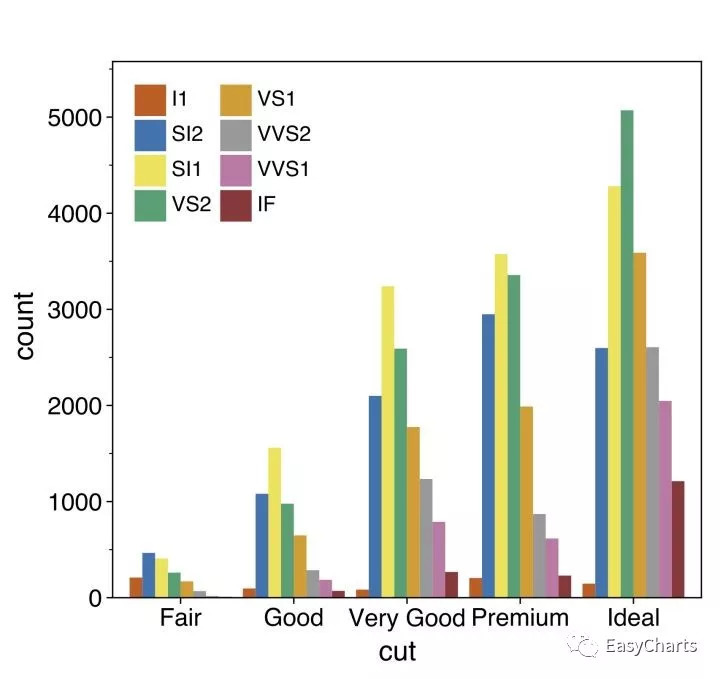
Violin plot 和boxplot:
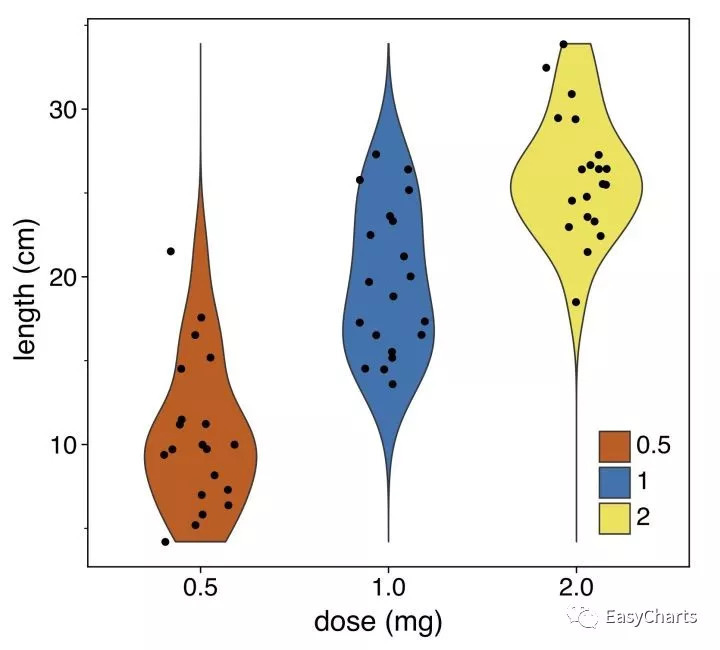
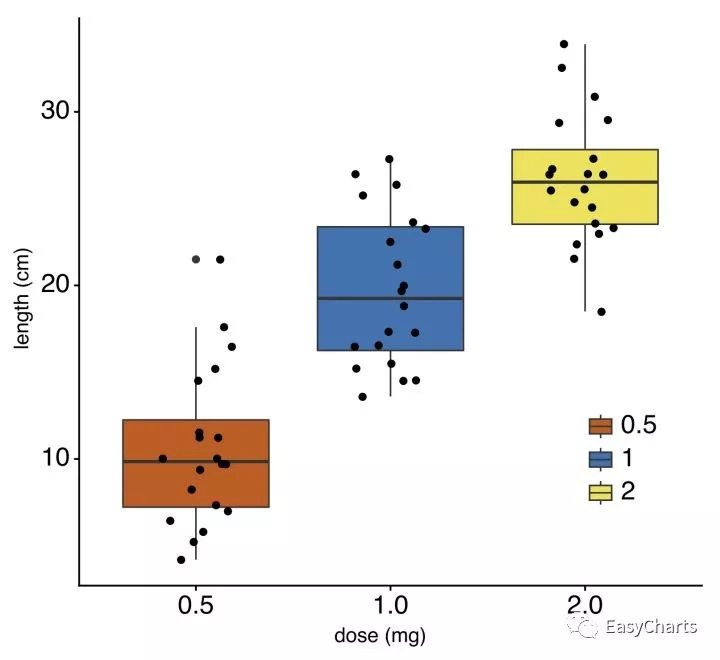
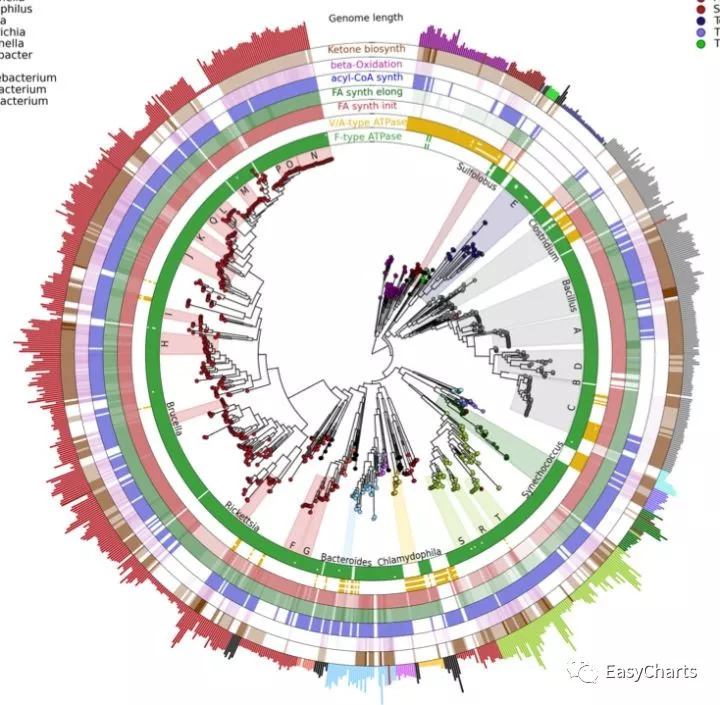
当然还可以基于python的maplotlib自己创作自己的图,Graphlan可以做出发育情况的图:
开始学习吧,和各种生物信息的软件结合起来,量化投资,金融,大数据处理可视化,这一些在python中都可以轻而易举。
plotnine:python数据可视化版ggplot2
plotnine数据可视化手册的领取方式:
1. 将该文章转发到微信朋友圈或者微信群,获得10个以上的赞;


转自EasyCharts团队
- 本文固定链接: https://www.maimengkong.com/image/1101.html
- 转载请注明: : 萌小白 2022年7月8日 于 卖萌控的博客 发表
- 百度已收录
