高质量scRNA-seq数据检索/注释工具:Cell BLAST
嗨,小伙伴们大家好!哈哈~新的一年祝大家牛年大吉,多发paper,多拿基金,工作科研两开花,家庭事业双福气!为配合风哥单细胞测序分析R语言全代码系列,知你所求懂你所需,本着饭喂到嘴边的少妇原则,弘毅给大家带来相关的无代码数据库介绍。这周就从Cell BLAST开始,一款高质量scRNA-seq数据检索/注释工具,一起来康康吧~!
序章:单细胞测序简介
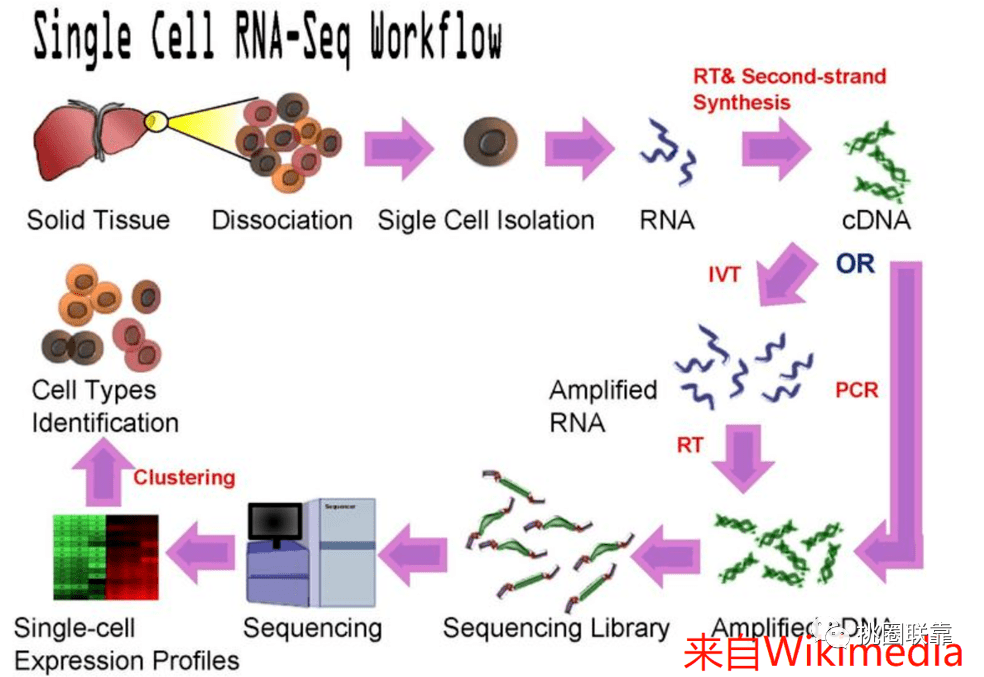
在这个测序技术应用和发展风起云涌的时代,DNA/RNA测序早已成为科研工作中的家常便饭。以往,RNA测序技术是基于检测样本中所有RNA转录本的方式来发现新型RNA分子或进行基因表达差异分析,获得的是组织样本或多细胞群的成千上万个细胞的综合结果,不同细胞亚群之间的差异往往被掩盖,尤其是在肿瘤异质性和免疫细胞亚群相关研究中,传统的测序技术显得捉襟见肘。正如同世界上没有完全相同的两片树叶一样,生物体中没有两个细胞是完全相同的,于是单细胞测序(single-cell sequencing)技术应运而生,自2013年被Nature Methods评为年度技术以来,近些年异军突起的单细胞测序技术逐渐显露出当红小鲜肉的潜质,并俘获一大批芳心青睐。单细胞RNA-seq最大的优势在于,能够独立地提供每个单细胞RNA表达谱数据,揭示每个细胞独特的微妙变化,使不同细胞类型得以精细区分,甚至可以鉴定并揭示新的细胞类型,使得在单细胞水平进行分子机制研究的设想成为可能。单细胞RNA-seq流程大的方面包括三个步骤,分别简述如下。
1
单细胞分离
即从异质性细胞群体中分离出单细胞,方法众多。高通量技术,如荧光标记流式细胞分选(fluorescence activated cell sorting,FACS)和免疫磁珠细胞分选(MACS)技术,可根据细胞大小/形状或表面标志物进行有偏向的选择。基于微流体和液滴的技术,如Fluidigm C1、10x Genomics Chromium和Bio-Rad ddSEQ Single-Cell Isolator系统可实现细胞无偏向分离。若对通量要求不高,可选择显微镜下手动挑选细胞,或采用激光捕获显微切割系统(aser capture microdissection,LCM),都是基于细胞形态或荧光报告基因表达的有偏向选择,优点在于可以了解单细胞所处细胞环境,缺点在于需要精细操作以免切到细胞或细胞核。相比之下,细胞有限稀释获得单细胞克隆的方法就是无偏向的。
2
RNA抽提、建库和测序
首先,裂解分离出的单细胞并纯化获得总RNA,可通过自动化设备或市售试剂盒来完成,设备如前文Fluidigm C1、10x Genomics Chromium和Bio-Rad ddSEQ Single-Cell Isolator,试剂盒如Thermo Scientific Single Cell Lysis Kit等。然后,大多数方案是通过polyA选择来富集mRNA,并以修饰的oligo dT引物进行逆转录。逆转录过程中,有些方案利用独特分子标识符(UMI)对单分子标记,可以更精确地定量单细胞中mRNA分子初始量。之后,通过体外转录或PCR扩增cDNA构建文库,用于高通量测序。目前测序方法众多,更新换代较快,2020年4月Nature杂志一篇题为Benchmarking single-cell RNA-sequencing protocols for cell atlas projects的论文阐述了不同单细胞测序技术的差异,并重点比较了13种常用scRNA-seq和单核RNA-seq实验方案优缺点。
目前主流的3种测序技术分别是:
(1) 以10x Genomics为代表的3‘/5’转录组测序:本方案对转录本3‘/5’端600nt以内的片段测序,可满足一般转录组定量分析需求,细胞通量较高,测序成本相对较低,缺点是其他信息被大量丢失;
(2) Smart-seq:本方案将cDNA打断后对转录本所有片段测序,优势是测序灵敏度高,可以检测转录本全长片段信息,缺点是细胞通量较低,单个细胞的测序成本较高,而且检测的并不是真实的全长转录本;
(3) 单细胞三代转录组测序:获得单细胞全长cDNA,经过扩增和建库后,用Nanopore平台或者Pacbio平台测序,cDNA不会被打断,避免了短reads拼接过程带来的错误,因此检测的是真实转录本全长信息,缺点是测序通量太低,错误率较高,并且花费过高。
3
数据分析
单细胞数据往往存在批次效应,给不同数据集之间的整合分析带来困难。导致批次效应的原因非常多,包括不同单细胞转录组建库技术带来的转录本捕捉效率和序列偏好差异,不同实验批次的操作差异,不同测序批次的测序深度差异,不同物种表达调控的差异,甚至是不同生物信息学分析流程带来的差异等等。因而跨数据集分析之前做好数据整合或矫正尤为重要。目前单细胞数据分析流程已经有非常完善的框架,大体上可以分为两步:
(1)数据处理:细胞和基因的预筛选,外源基因/线粒体DNA/核糖体DNA过滤,去文库标准化和去批次效应等;
以上是单细胞测序的简要介绍,以便小伙伴们有一个直观的认识,数据分析环节是我们需要重点学习的内容,风哥给大家带来一系列R语言全代码的丰盛大餐,弘毅呢就小小的锦上添个花,来介绍一下无代码的单细胞数据库,一起来品尝第一道甜点吧~!
数据库概览
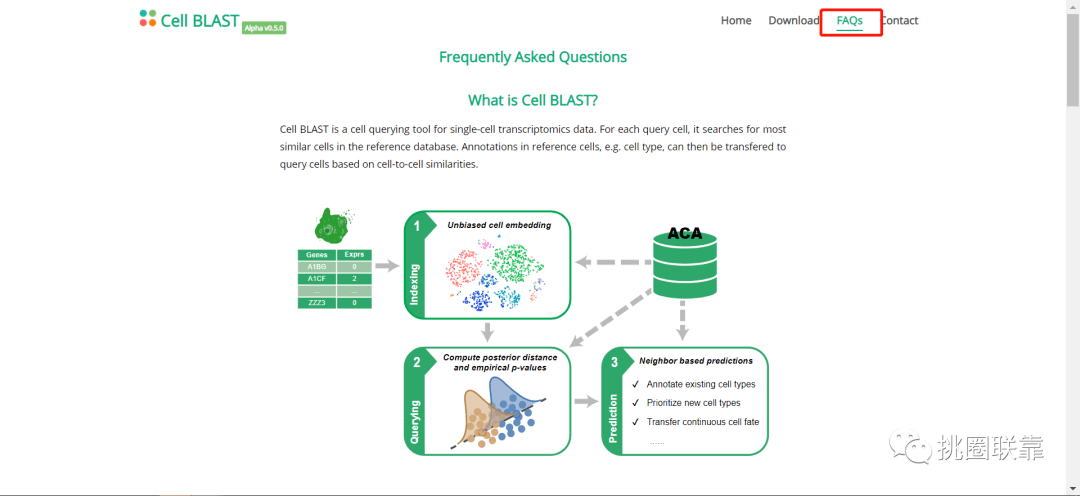
页面下拉,Cell BLAST很贴心的提供了数据库使用方法。
第一步,准备包含需要查询的基因表达矩阵文件,目前支持csv,tsv,h5ad和loom四种文件格式;
第二步,将数据文件上传到Cell BLAST,如果使用csv或tsv文件,系统将提示选择矩阵方向,即cell by gene或gene by cell,并显示前5行×5列的数据预览,以验证是否正确解析基因表达矩阵;
第三步,执行单细胞数据查询,确认已正确解析数据矩阵后,进入到BLAST选项卡进行参数设置,完成后单击BLAST按钮提交;
第四步,单细胞数据注释,获得检索结果后,可下载为csv格式文件,或者选择需要预测的特征参数后,点击PREDICT按钮获得自动注释结果。

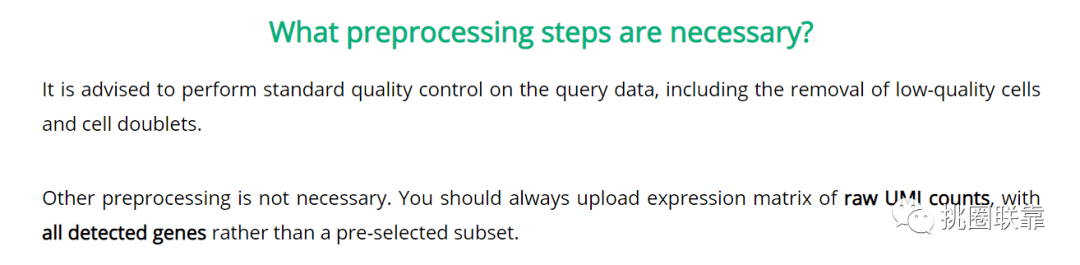
需要注意的是,在上传表达矩阵之前建议对查询数据执行标准的质量控制,可删除低质量的细胞数据和双峰数据,除此之外不需要其他预处理,应该上传原始UMI计数的表达矩阵,包含所有检测到的基因,而不是预先选择的数据子集。
另外,由于机密性原因不希望上传数据,或者更喜欢个性化定制,Cell BLAST提供Python软件包供使用,可以在自定义的参考数据集上进行模型操作、检索和个性化定制分析。目前网页版不支持跨物种分析,也需要借助Python软件包实现。

数据库功能及操作演示
1
检索功能:HOME
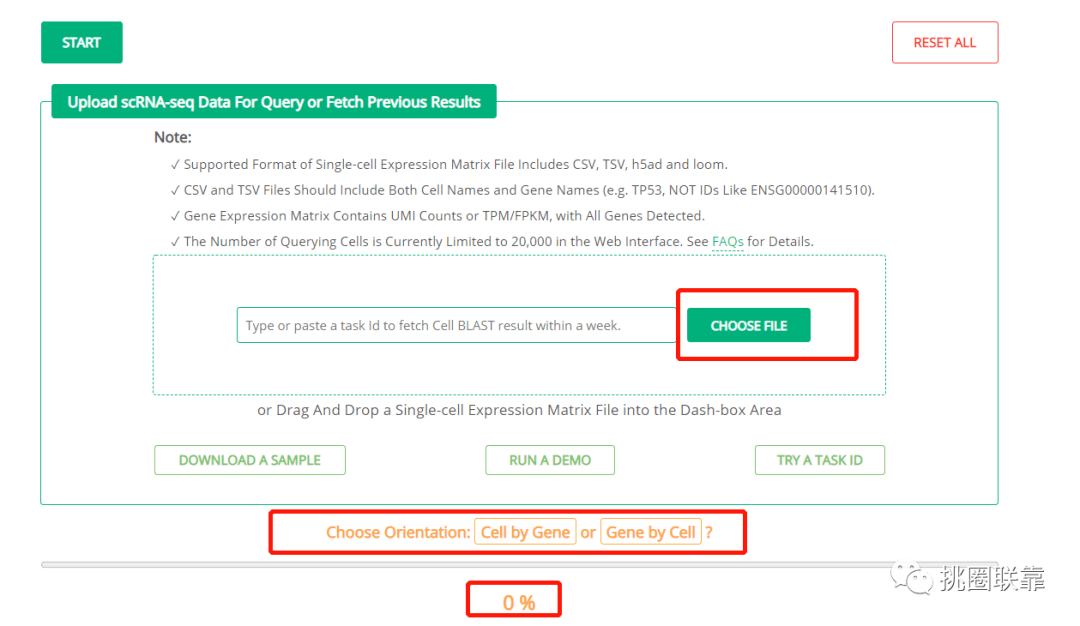
点击HOME进入数据库主页,检索框输入的表达矩阵须满足:
(1)上传的文件格式为csv,tsv,h5ad和loom,其中CSV或TSV文件须指定基因名称和细胞名称;
(2)包含UMI计数或TPM/FPKM,并且是检测到的所有基因,而不是预选的基因子集;
(4)当前Web界面的细胞数限制为20000。
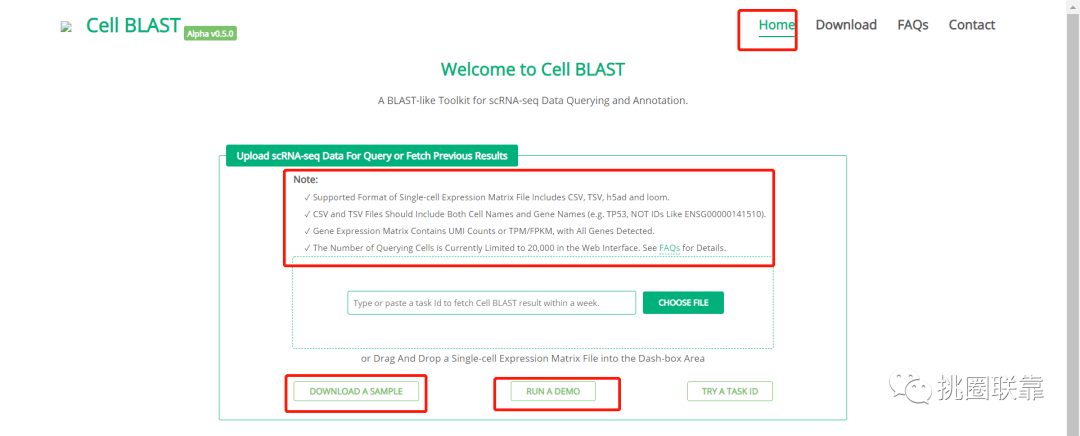
点击DOWLOAD下载示例数据,或者点击RUN DEMO,均可查看网站要求的表达矩阵文件的数据格式。
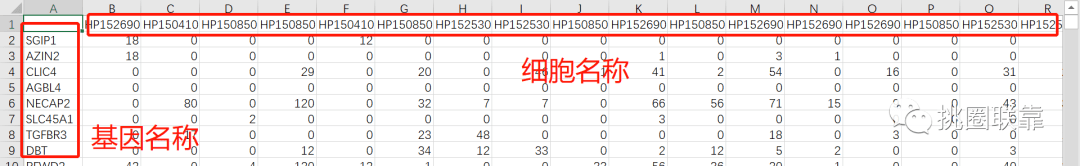
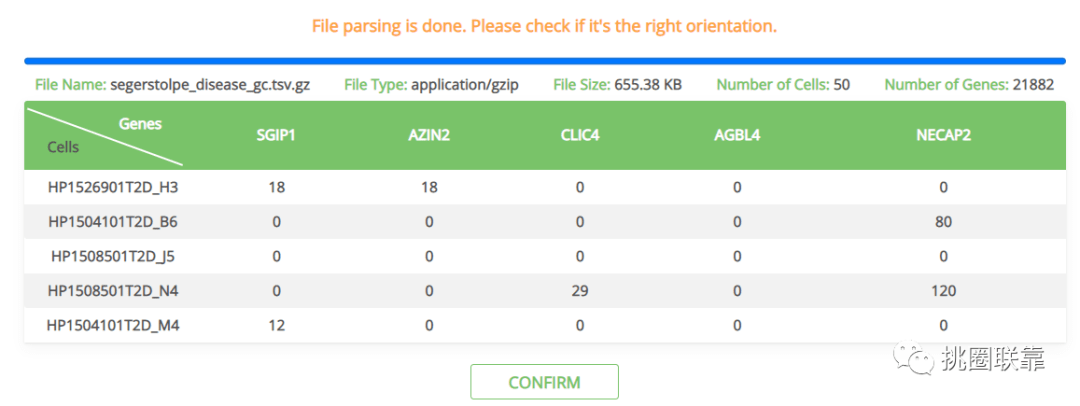
以示例数据为例进行演示,下载后解压,点击CHOOSE FILE,选择示例数据集TSV格式文件,页面弹出Choose orientation,此处选择gene by cell,进度条完成后可见表达矩阵详细信息:文件名称为segerstolpe_disease_gc.tsv,文件类型是application/gzip,文件大小为4.54MB,细胞数为50,基因数为21882。
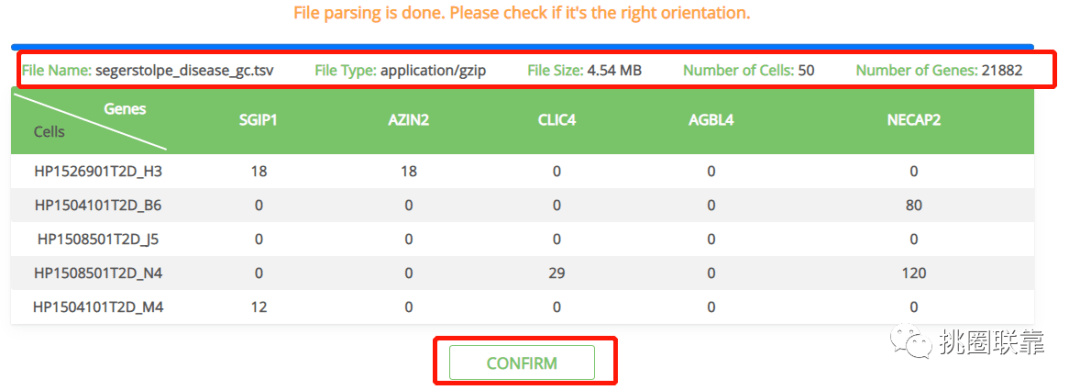
点击CONFIRM进入参数设置界面,依次选择数据集对应的物种和组织器官,以及需要匹配的参考数据集,接收结果的邮箱可留可不留,底下的高级设置默认即可,点击BLAST获得检索结果。
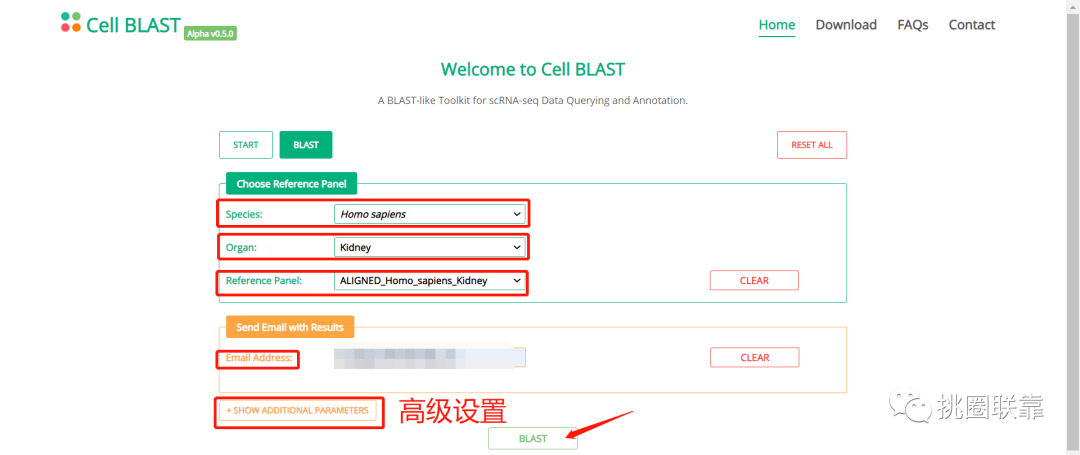
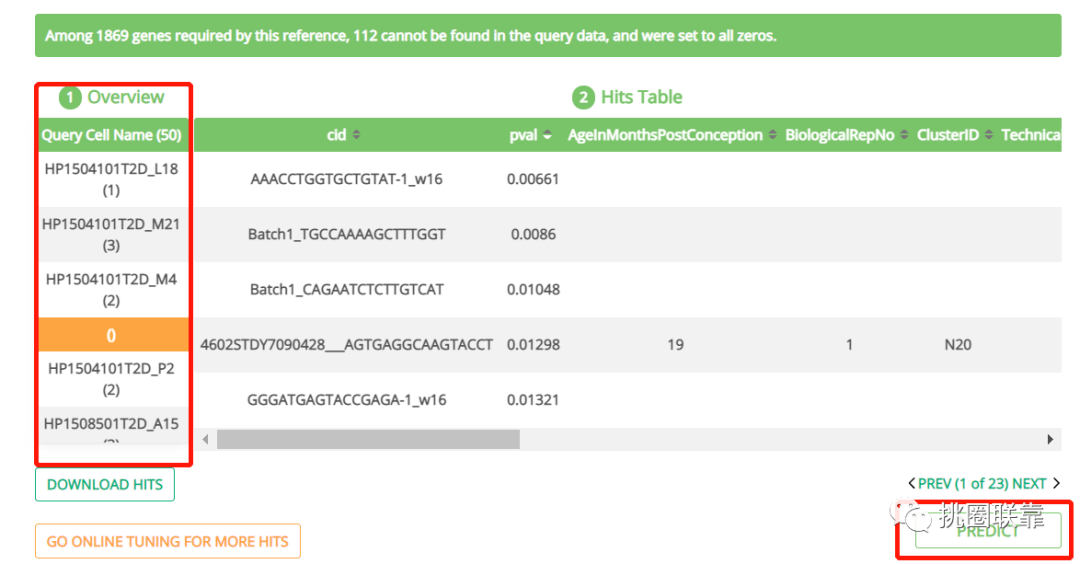
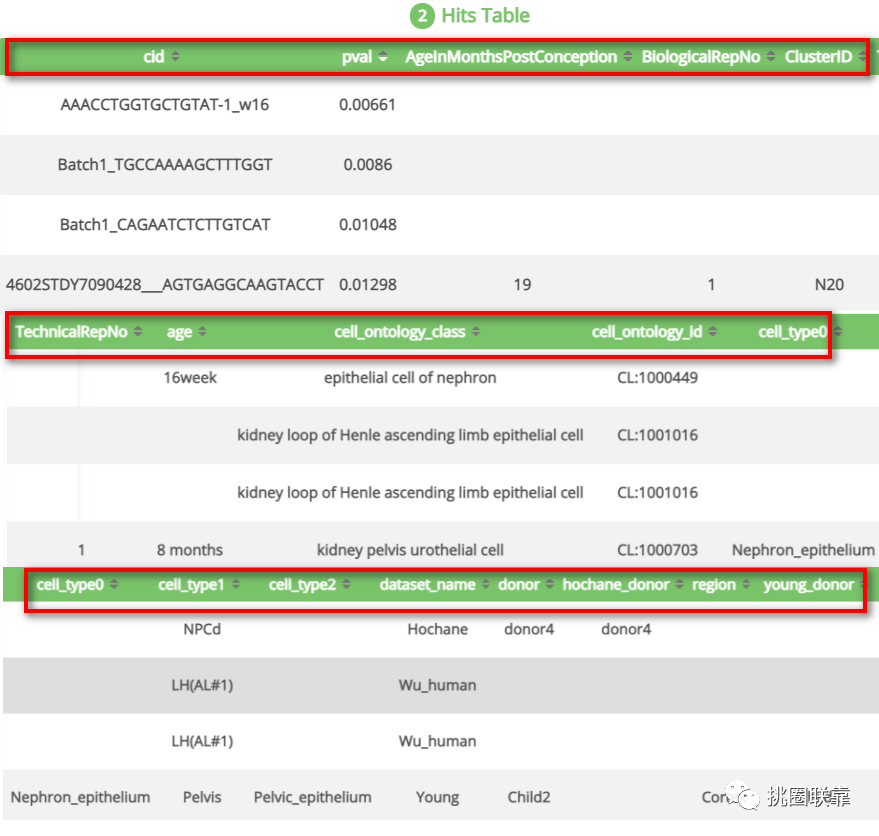
Query Cell Name栏为参考数据集中已注释的细胞,细胞名称下面的数字表示匹配到的基因数目,参考数据集1869个基因中有112个未匹配到,均以0表示,可作为潜在的新的细胞类型。点击表格下方DOWNLOAD HITS可下载检索结果,点击表格下方GO ONLINE TUNING FOR MORE HITS可返回上级界面重新设置检索参数。Hits Table表格显示各个类型细胞的详细信息。
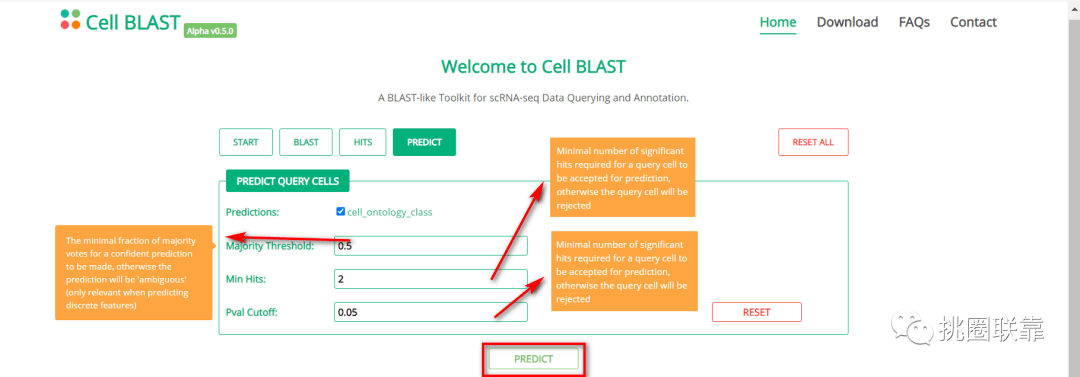
点击PREDICT进行细胞预测,鼠标悬浮在输入框可显示各个参数的意义,此处默认设置,点击PREDICT获得预测结果。
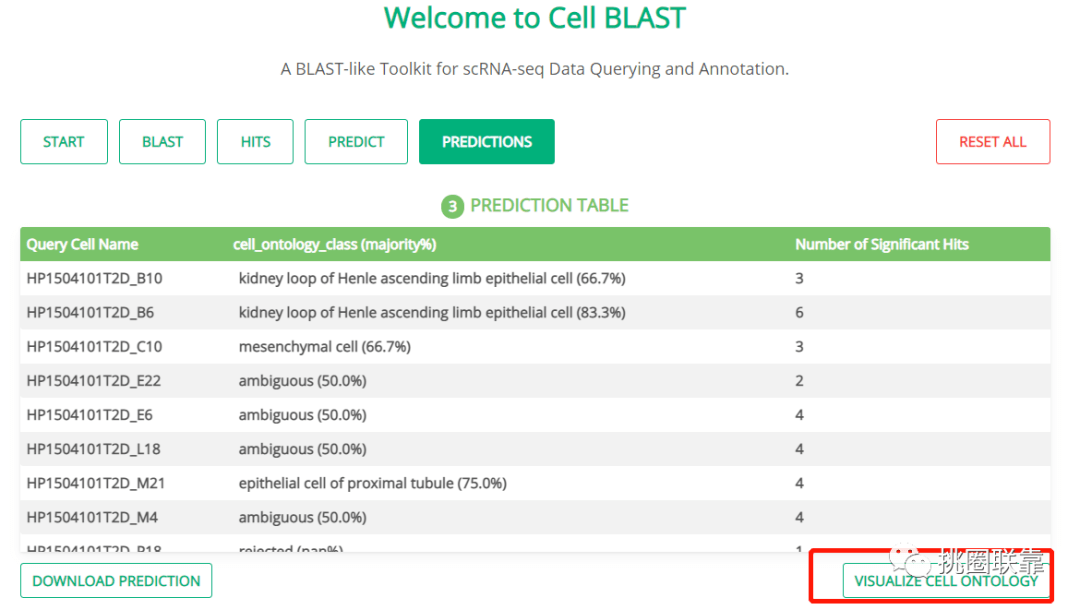
点击VISUALIZE CELL ONTOLOGY查看预测结果的可视化展示,节点表示不同细胞类型,节点颜色表示匹配到的基因数。双击某个节点,可重点显示该节点详细信息,及其相关的细胞。
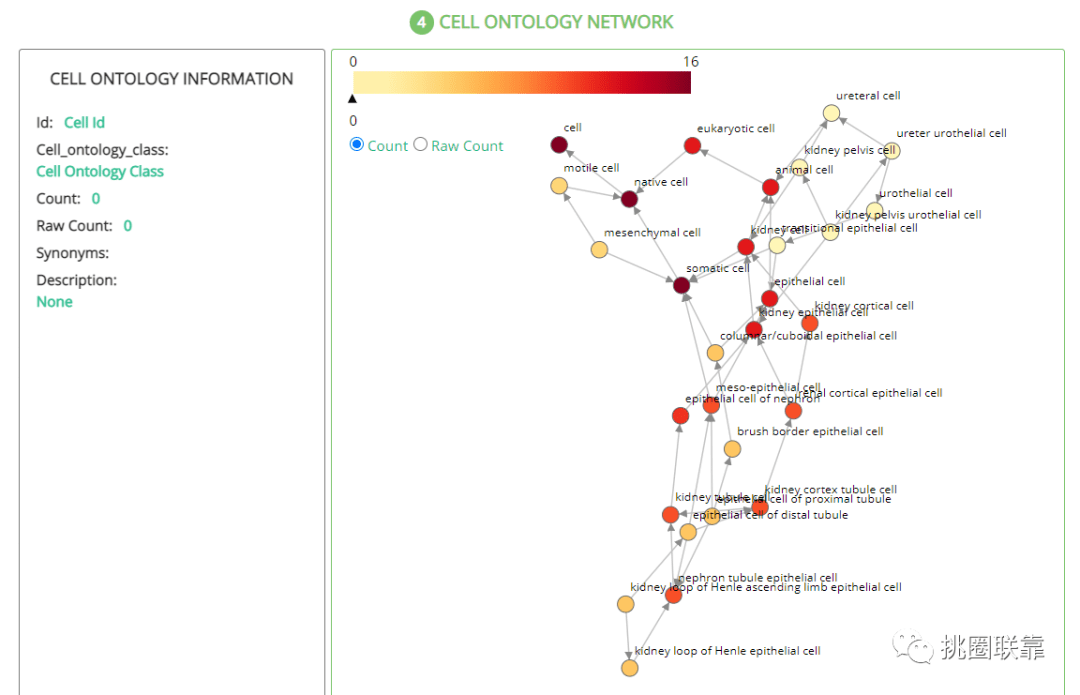
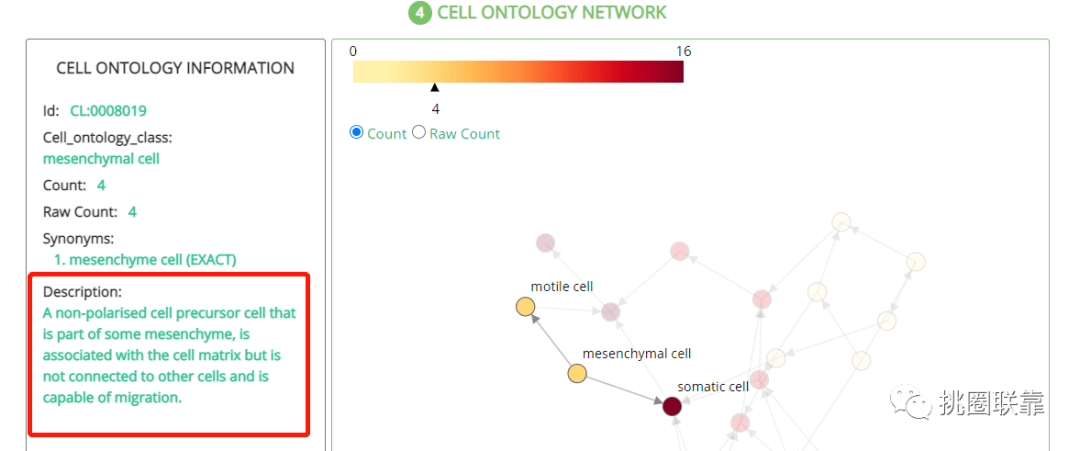
2
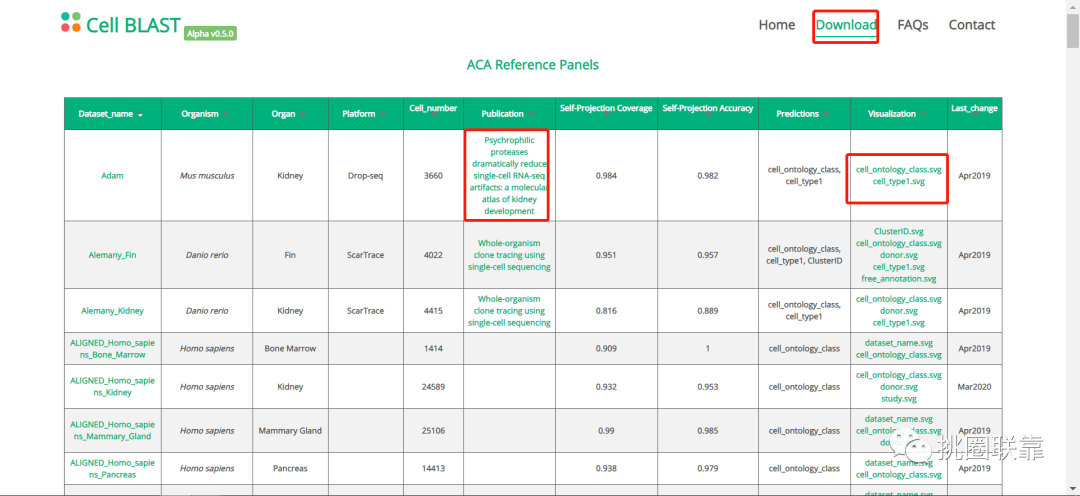
数据下载:Download
点击Download进入参考数据集ACA数据下载界面,提供数据集名称、物种信息、器官、检测技术平台、细胞数量和出版信息等。点击Visualization下链接可查看该参考数据集细胞类型可视化结果。
以上就是Cell BLAST数据库数据库的全部内容,目前引用文献不多,小伙伴们可以抓紧时间利用起来!另外,开发并维护数据库不易,小伙伴们使用时别忘记引用以下参考文献哦!~
Cao ZJ, Wei L, Lu S, Yang DC, Gao G. Searching large-scale scRNA-seq databases via unbiased cell embedding with Cell BLAST. Nat Commun. 2020 Jul 10;11(1):3458. doi: 10.1038/s41467-020-17281-7. PMID: 32651388; PMCID: PMC7351785.
转自:挑圈联盟
- 本文固定链接: https://www.maimengkong.com/zu/1664.html
- 转载请注明: : 萌小白 2024年2月4日 于 卖萌控的博客 发表
- 百度已收录
