基因序列是大多数生物医学研究僧经常碰到的一种的数据。不同于长达几kb的序列,在一些情况下,我们需要在文中展现局部的,比如仅有10-20bp,序列突变情况,这就构成了一个seqlogo。
那seqlogo是啥?长啥样,请看下图:

在openi网站,输入关键词sequence logo,就可以发现有非常多的seqlogo,这些图都来自于已经发表的SCI论文(是不是又给大家透露了一个科研神器)。
简单来说,seqlogo展示的就是一小段序列的多样性。对于DNA序列来说,每个位点仅有AGTC四种可能,因此,图中每个位点的字母的大小代表的是该位点该碱基出现的概率。当然,对于氨基酸序列来说,由于存在20种不同的氨基酸,因此序列的多样性会更加丰富一些。但是,seqlogo的原理是一样的。
接下来,Leopard将为大家介绍如何绘制这种seqlogo,主要涉及两种类型。
目前可及的绘制seqlogo的工具不算很多,常见的有网页工具Weblogo,seqLogo包,gglogo包,以及最近发表的ggseqlogo包。综合比较下来,ggseqlogo包功能最强,绘制出的图形最好看。
使用ggseqlogo之前,请先下载并加载该包。

模式一
展示基因序列多样性
Ggseqlogo的使用非常简单,主函数为ggseqlogo(),该函数可以直接接受已经比对好的DNA或者氨基酸序列,但是有一点需要注意,比对好的序列中可以有gap,但是所有序列的长度必须相同,否则会报错。
下面Leopard为大家展示如何快速生成一副seqlogo。此处我用的是ggseqlogo包中的示例数据集,后面再给大家演示如何利用自己的数据进行绘制。
如你所见,seqs_dna中的MA0001.1对象其实是一个含有97个碱基序列的向量,而且,这97个碱基序列其实都是独立的字符串元素。因此,如果大家利用R随机生成一串字符数相同的字符串向量,也是可以用来绘制seqlogo的。
下面展示的是MA0001.1的seqlogo:
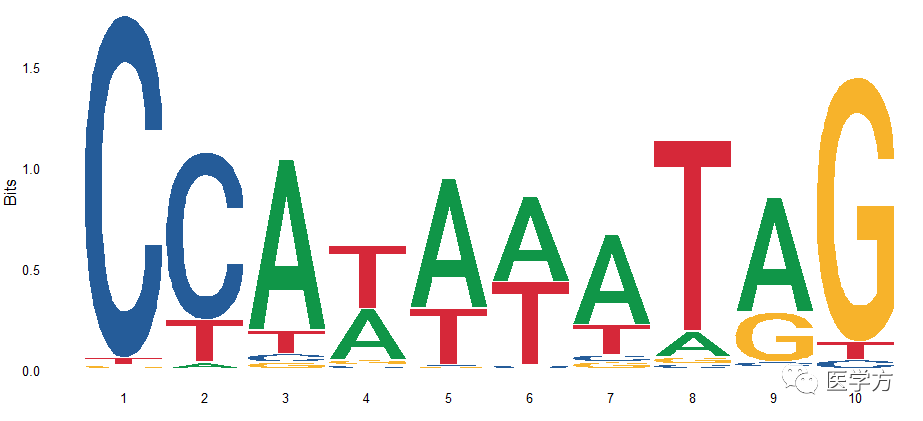
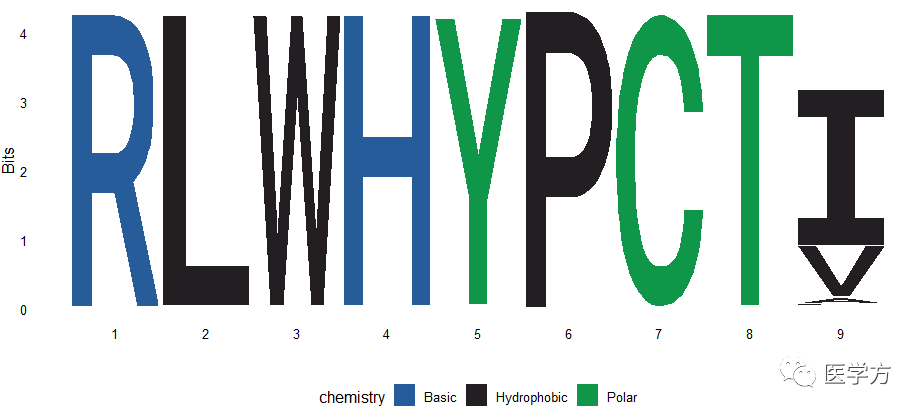
在上面的seqlogo中,横轴是每一个碱基的位点,纵轴默认情形下是Bits,可以替换成“probability”。关于bits和probability的区别,我在此不做赘述,大家可以去维基百科查询。总之,记住一点,不管是什么指标,都是用来衡量某个位点某个碱基出现的可能性的大小。显然,碱基的字符越高(宽度是相同的),其出现的可能性越大。
以上就是最简单的seqlogo。ggseqlogo还提供了色彩调配的函数,有兴趣的读者可以自己去尝试,我个人觉得这个包的默认配色已经很棒了。
下面我为大家展示如何利用我们自己的序列数据进行seqlogo的绘制。下图展示的是氨基酸序列。
绘图之前,首先要做的就是把序列读进R语言。
Fasta格式的序列读进R语言后,并非字符串向量,因此在绘图之前,我们还需要把序列转换成字符串向量。
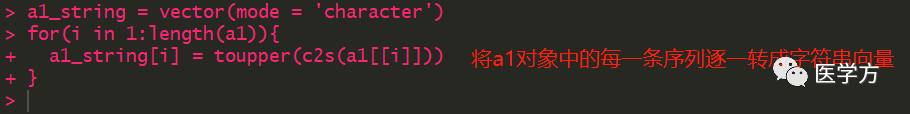
完成这一步后,绘图就很简单了。直接使用ggseqlogo(a1_string)即可。图形如下:
模式二
绘制单个位点的序列多样性
模式一给大家展示的是最常见的seqlogo,即横轴都是序列的位点。但是,有些时候,我们仅仅需要展示单个位点的多样性,比如HCV耐药位点等。此时横轴仅有一个位点,所以我们想把横轴换成其他的信息,比如HCV的不同亚型,然后以seqlogo的形式展示HCV不同亚型中同一个耐药位点的突变情况。
碰到这种情况,比模式一稍微复杂一点点,但是也很简单。下面为大家介绍如何实现这种类型的seqlogo。
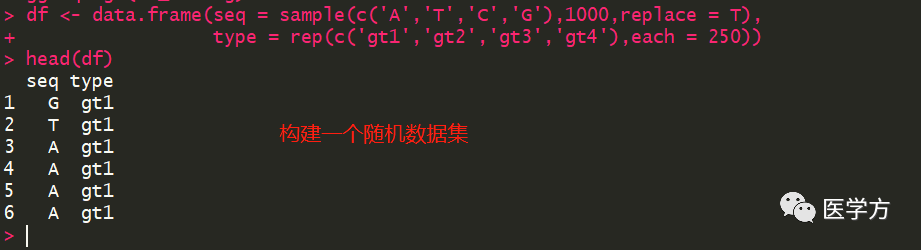
在df中,seq表示的是某位点的碱基,type表示的是序列亚型。注意,此处df中展示的碱基都是在同一个位点。
下一步我们需要根据df的信息,构建一个矩阵,行名是碱基名称,列名是序列亚型。如下所示:

最后一步,利用构建好的矩阵绘图即可:
图形如下:

如你所见,很简单的,我们就实现了第二种模式的seqlogo。
通常来讲,seqlogo的情形主要是以上两种。由于ggseqlogo可以进行图层叠加,因此我们也可以在seqlogo上进行注释等等,用法与ggplot2中的用法一样,在此不做赘述。
希望ggseqlogo能为各位做序列分析的童鞋带来帮助。
- 本文固定链接: https://www.maimengkong.com/image/1407.html
- 转载请注明: : 萌小白 2023年3月25日 于 卖萌控的博客 发表
- 百度已收录
