早在1991年,一位来自法国的生物学家Michelmore首次将BSA技术应用到莴苣的霉病抗性基因位点的验证之中,他利用RFLP和RAPD两种标记对两个不同抗性的莴苣混合样本进行探究,根据不同混池样本的RFLP和RAPD凝胶电泳成像结果,最终找到了3个与霉病抗性基因紧密相关的标记[1],这一结果标志着BSA的“诞生”,由此拉开了BSA的序幕。
高通量测序技术的不断发展,推进了BSA技术的应用,先后催生了SHOREmap[2],NGM[3],Mutmap[4]以及QTLseq[5]等BSA系列分析技术。BSA的应用也从模式物种拓展到了非模式物种,发表文章也越来越多。可以说,基于高通量测序技术的BSA性状定位方法已经成为当前分子遗传育种重要方法之一。
BSA 的基本原理
BSA(Bulked Sergeant Analysis),即分离体分组混合分析法,也称为集群分离分析法或混合分组分析法,是从近等基因系分析法演变而来的。近等基因系(NIL)指一组遗传背景相同或相近,只在个别染色体区段上存在差异的株系。BSA法克服了许多作物没有或难以创建NIL的限制,其原理是从一个分离群体中选择目标性状表型极端的10~20个单株,混合构建2个DNA“池”,这两个池应在感兴趣的性状方面存在差异,除了感兴趣基因所在的位点外,所有的位点均随机化。换句话说,两个DNA池间的差异相当于两近等基因系基因组之间的差异,仅在目标区域不同,而整个遗传背景是相同的。对两个池筛选标记,多态性标记可能表示与感兴趣的某个基因或QTL连锁。在检测两个DNA池之间的多态性时,通常应以双亲的DNA作对照,以利于对实验结果的正确分析和判断。
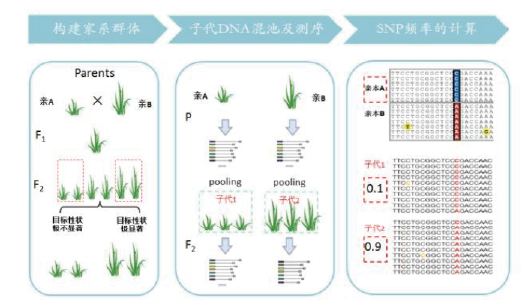
图1. BSA基本原理
BSA 方案设计

BSA 影响因素
1)参考基因组:参考基因组组装质量好坏以及注释是否完全对BSA结果有一定的影响。建议尽量选择组装至染色体水平,且基因组注释较完全的参考基因组。
2)分离群体:理论上任何一对具有相对性状的亲本杂交后产生的分离后代都适用于集群分离分析法。但常用的分离群体为:F2代群体、回交群体、重组自交系、双单倍体群体。
3)亲本选择:亲本间目标性状要求差异显著,尽量选用纯度高的亲本,并进一步通过自交进行纯化;并且,除了关注的1个目标性状外,其他性状尽量一致,无其他性状发生分离。
4)混池个体数:混池的样本数通常选取极端性状(群体总样本数的5~10%内)的个体进行混池。混池中若极端性状个体数太少,可能造成样品数不够,不具备代表性;若样本数过高可能会引入杂合个体,产生干扰。
5)表型统计:表型统计不准确,或表型由多个微效基因控制,可能会引起定位效果不佳。
BSA 代表文献
BSA性状定位相关研究发表文献影响因子水平通常为3~5分。已有许多相关研究成果发表在《Nature Biotechnology》(IF:39.08),《The Plant Journal》(IF:6.582),《Theor Appl Genet》(IF:3.507),《BMC Genomics》(IF:4.397)等SCI著名期刊上。
BSA 常见问题
问:1)子代混池的样品个数和测序深度如何设计?为什么?
答:2013年,James et al.[6]对混池个体数和测序深度对候选基因数的影响进行了评估(图2)。当子代个数超过20个,测序深度达到25X以后,候选基因个数会趋于平衡,不会有更明显的提升效果。基于以上文章经验以及已完成的BSA项目经验,通常推荐20个个体进行混池,每池20X的测序深度。
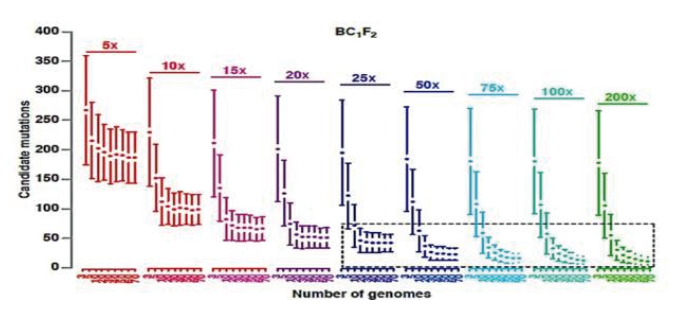
图2 混池样本个数和测序深度对候选基因数的影响
(横坐标是混池的样本个数,纵坐标是候选基因数)
问:2)能否只测亲本?或者只测子代池?或者只测1个子代池?
答:分两种情况:A) 如果研究的性状是数量性状,最好测2个亲本和2个子代池;若只测两个子代池,假阳性可能会较高,候选SNP可能会较多。
B)如果研究的性状是EMS诱导的突变性状,可以只测两个子代池(野生型+突变型),或者测1个亲本(野生型)和1个子代池(突变型)。
问:3)能否保证定位到的候选区域的大小和候选基因的个数?
答:候选区域的大小和候选基因个数的多少,与群体的大小、亲本材料差异的大小、目标性状特点、测序深度多少、分析物种的基因组水平等多项因素相关,因此很难给出统一的标准,但可以参考已完成的项目经验进行估计。
问:4)通过BSA得到候选基因后,如何进一步筛选目标基因?
答:通过BSA 方法得到候选基因后,可以对候选基因进行进一步筛选,常见筛选方法如下:
(1)根据基因功能注释结果筛选,找到的几十个基因不一定都和研究的性状相关,去除无关基因;
(2)结合已有的SSR遗传图谱进行QTL定位的结果,将候选区间缩小;
(3)构建更大的群体进行定位,增加取样数,增加测序深度;
(4)通过多组重复实验,取交集。
问:5)通过BSA 得到候选基因后,如何进行基因验证?
答:经过候选基因筛选,得到目标基因后,可以进一步对SNP或者基因的功能进行验证,常见验证方法如下:
(1)验证SNP
(A)将候选SNPs转化成CAPS或dCAPS等标记进行验证。即:对候选的SNPs进行限制性内切酶识别位点分析,筛选出引起酶切识别位点改变的SNPs,使用相应的引物扩增这些SNPs所在的片段, 然后对扩增产物进行酶切和电泳检测,将SNPs转化为CAPS标记;对CAPS标记进行多态性分析,以验证标记的可用性;
(B)将候选SNP所在区段进行PCR扩增,然后利用Sanger测序验证扩增产物;
(2)利用RT-PCR验证候选基因表达量在不同表型中是否有变化;
(3)基于转录组的差异表达分析,看基因表达是否有显著差异;
(4)RNAi分析:使用RNAi技术特异性剔除或关闭特定基因的表达。
问:6)多倍体物种可以进行BSA性状定位吗?
答:有参考基因组的多倍体物种,例如:“油菜、烟草、棉花”,可以用BSA进行性状定位。
- 本文固定链接: https://www.maimengkong.com/1219.html
- 转载请注明: : 萌小白 2022年10月3日 于 卖萌控的博客 发表
- 百度已收录
