单细胞转录组学测序是目前比较火热的组学技术,单细胞测序在很多高分文章中露脸的次数非常频繁。然而数据拿到手,分析后的可视化也是发文章时的重点内容。看到CNS或者其他高质量文章中单细胞的可视化总是一种高大上的感觉,可是自己手里的数据可视化却是与别人有很大差距。那么,接下来就随着笔者初探单细胞作图的修饰,抛弃默认出图,修饰作图,提高档次,向好的文章看齐!也让自己的数据有一个更好的呈现!
为了方便演示,示例数据我们采用Seurat官方提供的PBMC单细胞数据。首先加载数据。
1 .library( SeuratData) #加载 seurat数据集 2 .InstallData(" pbmc3k") 3 .data(" pbmc3k") PBMC< -pbmc3k.final
可能到这里会发现一个错误。
1.#ERROR 2.# Errorineval(call( "@", object, slot)): 3.# "images"槽名不存在于 "Seurat"类别对象中
这是因为加载的数据时Seurat较早的版本,目前已经更新到4.0了,所以需要将其转换一下。( 知识点:例如在公共数据库挖掘时,拿到别人Seurat2的数据时,就可以用下面的函数转化)
PBMC = UpdateSeuratObject(object = PBMC) #旧版的seurat对象转变为新版
数据准备完成了,就开始真正的操作吧!
1、UMAP聚类图的修饰
所有的画图软件都会有一个默认的配色,单细胞作图的seurat也不例外。但是如果你阅读了足够多的单细胞好文章,就会发现,这些文章基本无一例外的,都不会使用默认的配色,而是选择自己配色,让图形可视化更加好看。
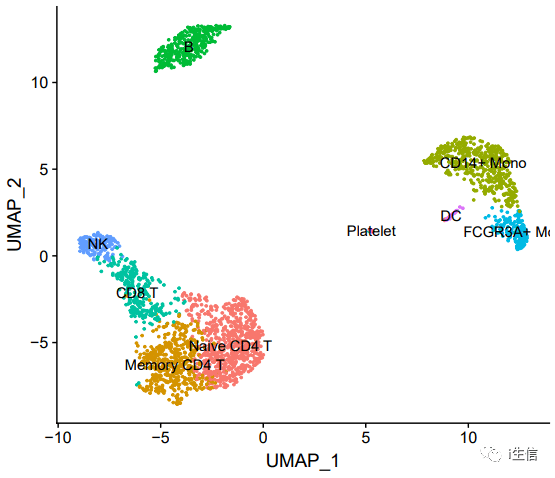
一般默认情况下,seurat做出的UMAP图如下,这种配色其实很普通,如果整个文章都是这个色调,看起来不是很好。
1. #普通展示细胞聚类图 DimPlot(PBMC, label = T)+NoLegend
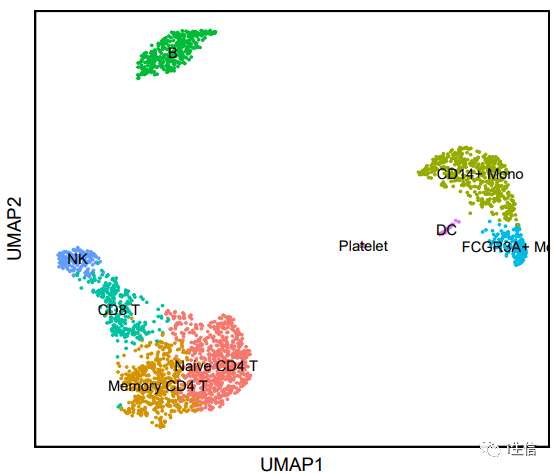
如果稍加修饰,修改坐标轴,去除坐标轴刻度,自定义添加标题,给UMAP图加上边框等等修饰,立马就能看出区别。但是相比于高分文章,还是有点欠缺。
1.DimPlot(PBMC, label = T)+ 2. NoLegend+labs(x = "UMAP1", y = "UMAP2") + 3. theme(axis.text.y = element_blank, 4. axis.ticks.y = element_blank, 5. axis.text.x = element_blank, 6. axis.ticks.x = element_blank)+ 7. theme(panel.border = element_rect(fill=NA,color="black", size=1, linetype="solid")) #加边框
其实关键的还是对颜色的修饰,以两篇高分文章的配色为例看看效果:
#第一种配色方案来源于《Cell》文章:DOI:10.1016/j.cell.2021.05.013。
1.library(paletteer) #提供了 R 编程环境中提供的数百种其他调色板的组合集合,详情可以查看此包,总有你满意的方案 2.pal <- paletteer_d( "ggsci::nrc_npg")[c(1,3,4,9,5,2,6,8,10)] #有几群细胞需要标记就选几种颜色 3.DimPlot(PBMC, label = T, 4. cols= pal, #设置颜色 5. pt.size = 1.5, #设置点的大小 6. repel = T)+ #标注有点挤,repel=T可以让排列更加合理 NoLegend
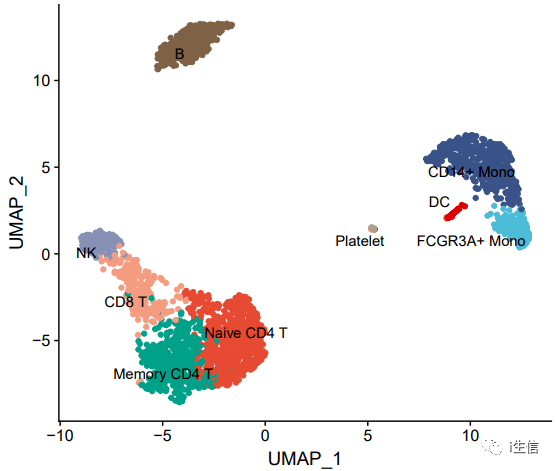
是不是感觉有那“味”了!
1.library(RColorBrewer) #配置自己需要的颜色 2.cell_type_cols <- c(brewer.pal(9, "Set1"), "#FF34B3", "#BC8F8F", "#20B2AA", "#00F5FF", "#FFA500", "#ADFF2F", "#FF6A6A", "#7FFFD4", "#AB82FF", "#90EE90", "#00CD00", "#008B8B", "#6495ED", "#FFC1C1", "#CD5C5C", "#8B008B", "#FF3030", "#7CFC00", "#000000", "#708090") 3.DimPlot(PBMC, label = T, 4. cols= cell_type_cols, #设置颜色 5. pt.size = 1.5, #设置点的大小 6. repel = T)+ #标注有点挤,repel=T可以让排列更加合理 7. NoLegend
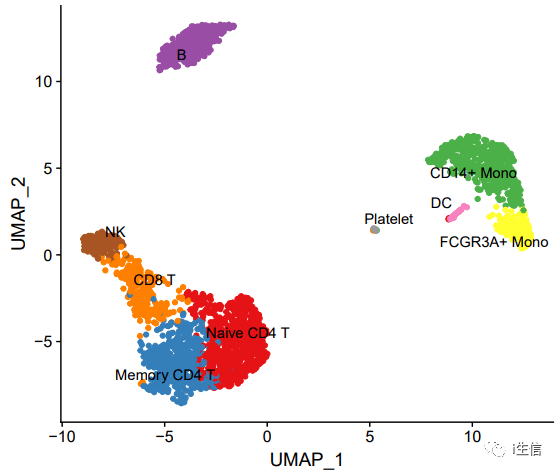
这样做出的图更加好看,符合高分文章的标准!
UMAP图还有一个重要的作用是是展示某些基因在cluster中的表达。
1. #默认作图 2.FeaturePlot(PBMC, features = "S100A8") 3. #修饰图片 4.mycolor <- c( 'lightgrey', 'blue', 'seagreen2') #设置颜色 5.FeaturePlot(PBMC, features = 'S100A8',cols = mycolor, pt.size = 1.5)+ theme(panel.border = element_rect(fill=NA,color= "black", size=1, linetype= "solid")) #加边框
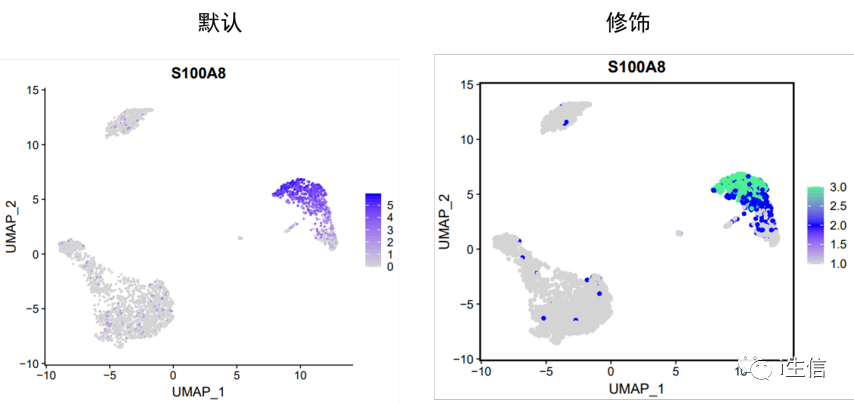
2、热图修饰
热图是单细胞测序数据分析中展示marker基因的重要方式,一般使用DoHeatmap函数实现。默认的图片也是可以的,但是缺乏生动的感觉,所以很少文章用默认的画图,多多少少也是进行了修饰。
这里用每个cluster前top3基因演示,默认的绘图如下:
1.DoHeatmap(PBMC, 2.features = as.character(unique(top3$gene)), 3.group. by= "seurat_clusters", 4.assay = "RNA")
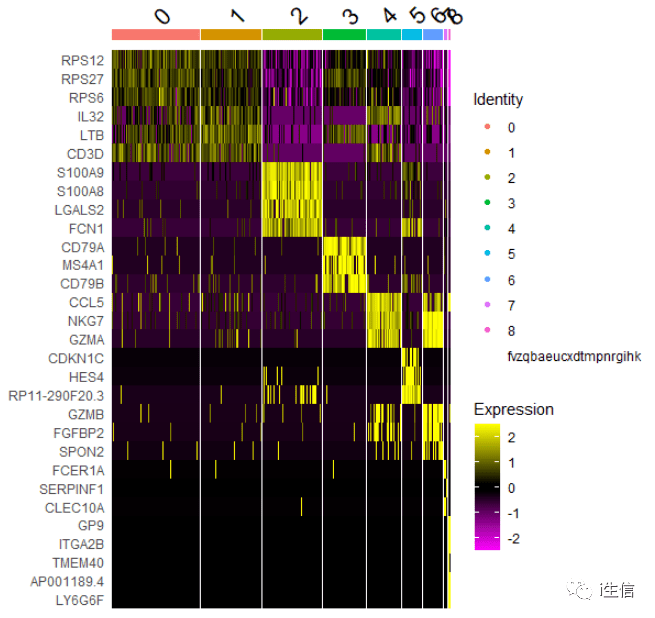
我们对热图配色进行修改,看起来会顺眼很多!
1.DoHeatmap(PBMC, 2.features = as.character(unique(top3$gene)), 3.group. by= "seurat_clusters", 4.assay = "RNA", 5.group.colors = c( "#C77CFF", "#7CAE00", "#00BFC4", "#F8766D", "#AB82FF", "#90EE90", "#00CD00", "#008B8B", "#FFA500"))+ #设置组别颜色 6.scale_fill_gradientn(ccolors = c( "navy", "white", "firebrick3")) #设置热图颜色
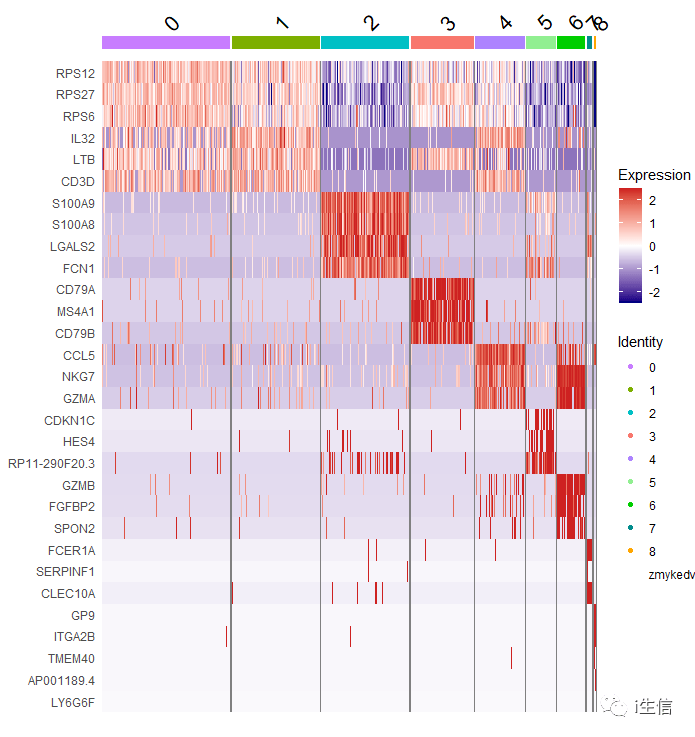
看这个热图,其实我们可以发现,4群和6群的细胞marker基因表达一致,但是热图是按照0-8这个顺序排列的,如果将顺序调整一下,自定义cluster的排序,会让结果看起来会更加明显。
1.PBMC$seurat_clusters <- factor(x = PBMC$seurat_clusters, levels = c( '0', '1', '2', '3', '4', '6', '5', '7', '8')) 2.DoHeatmap(PBMC, 3.features = as.character(unique(top3$gene)), 4.group. by= "seurat_clusters", 5.assay = "RNA", 6.group.colors = c( "#C77CFF", "#7CAE00", "#00BFC4", "#F8766D", "#AB82FF", "#90EE90", "#00CD00", "#008B8B", "#FFA500"))+ #设置组别颜色 7.scale_fill_gradientn(ccolors = c( "navy", "white", "firebrick3")) #设置热图颜色
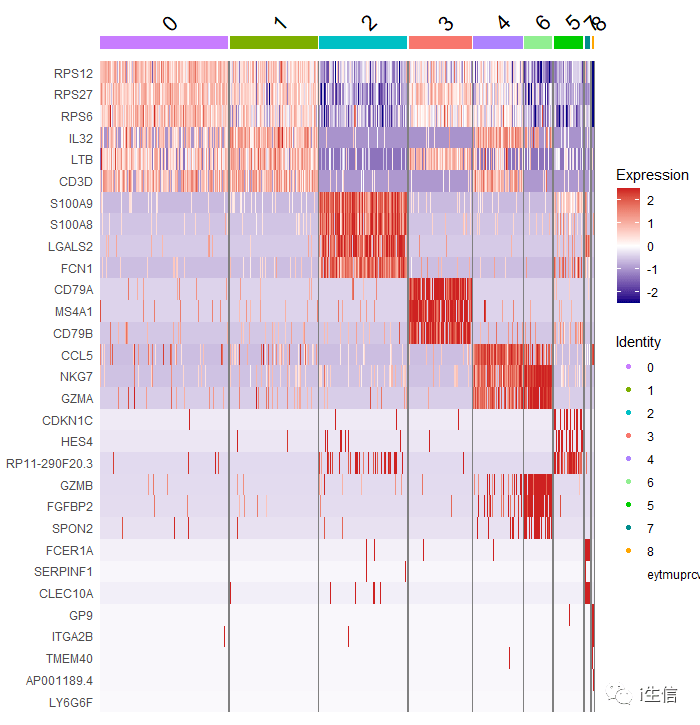
当然了,以上的改变我们都是在Seurat默认画图函数的基础上修饰得到的,如果要通过marker基因从头画热图,则需要提取表达矩阵和分组信息,用Heatmap来做。具体已经有人做了这个工作,可以参考链接:
3、改造小提琴图
小提琴图也是文章中最常见的图,例如下图,也是比较好看的,可以用于展示marker基因。
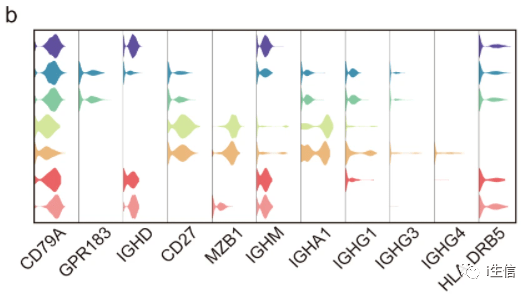
(文章截图)【1】
但是R语言画出来的小提琴图只能单独展示一个基因的表达,笔者在查阅后发现,文章中这种小提琴图基本是用Python画的,这就很不友好,总不能为了一个图学python吧,也太费时间了。不过还好,有人已经通过改造,出了一个包,帮我们解决这个问题(感谢分享者)。
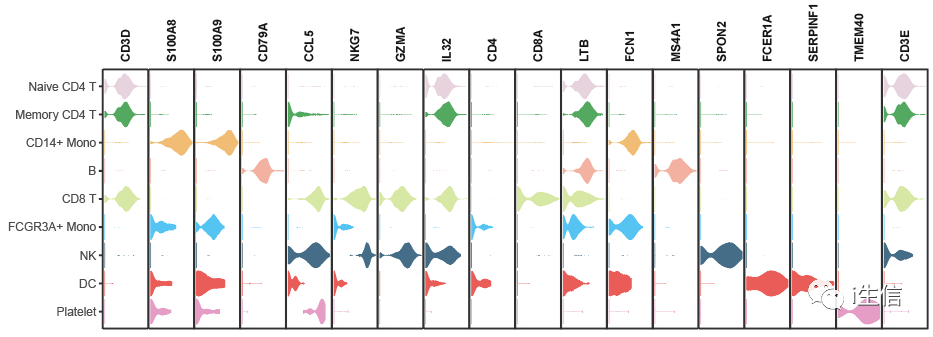
1.install.packages( "remotes") 2.remotes::install_github( "lyc-1995/MySeuratWrappers") #通过链接安装包 3.library(MySeuratWrappers) 4.#需要展示的基因 5.markers <- c( 'CD3D', 'S100A8', 'S100A9', 'CD79A', 'CCL5', 'NKG7', 'GZMA', 'IL32', 'CD4', 'CD8A', 'LTB', 'FCN1', 'MS4A1', 'SPON2', 'FCER1A', 'SERPINF1', 'TMEM40', 'CD3E') 6.my36colors <-c( '#E5D2DD', '#53A85F', '#F1BB72', '#F3B1A0', '#D6E7A3', '#57C3F3', '#476D87', '#E95C59', '#E59CC4', '#AB3282', '#23452F', '#BD956A', '#8C549C', '#585658', '#9FA3A8', '#E0D4CA', '#5F3D69', '#C5DEBA', '#58A4C3', '#E4C755', '#F7F398', '#AA9A59', '#E63863', '#E39A35', '#C1E6F3', '#6778AE', '#91D0BE', '#B53E2B', '#712820', '#DCC1DD', '#CCE0F5', '#CCC9E6', '#625D9E', '#68A180', '#3A6963', '#968175') #颜色设置 7.VlnPlot(PBMC, features = markers, 8.stacked=T,pt.size= 0, 9.cols = my36colors, #颜色 10.direction = "horizontal", #水平作图 11.x.lab = '', y.lab = '')+ #横纵轴不标记任何东西 12.theme(axis.text.x = element_blank, 13.axis.ticks.x = element_blank) #不显示坐标刻度
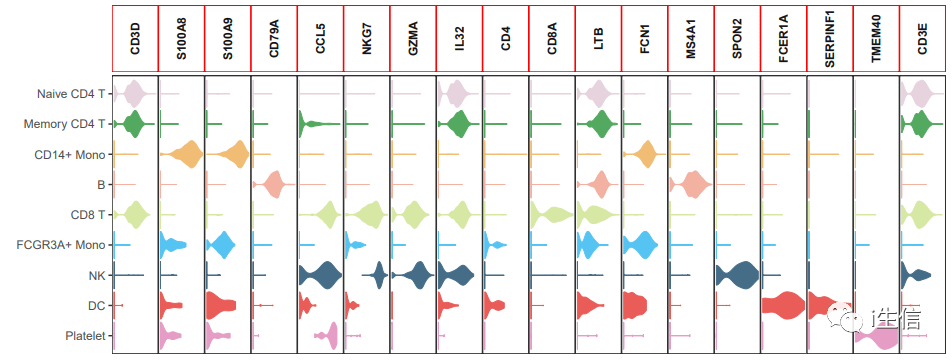
最后利用AI将图片稍加修饰,就可以得到与别人文章中一样的图了。
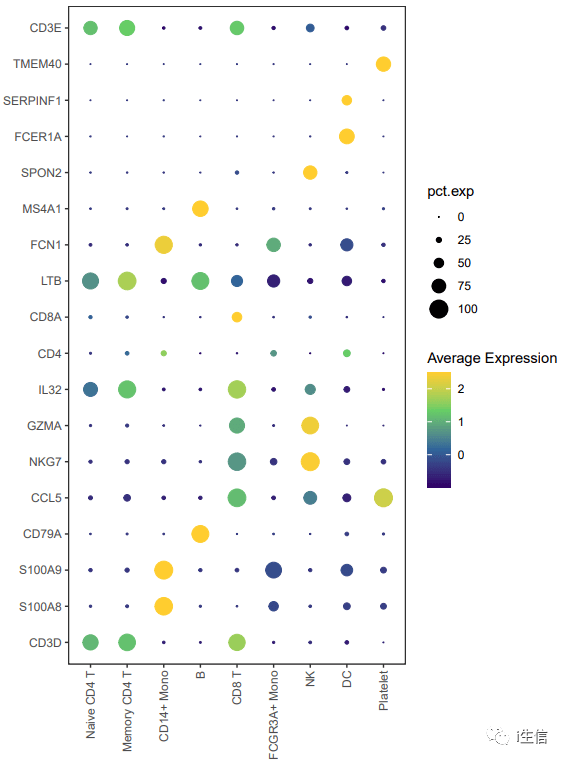
4、Dotplot气泡图的修饰
DotPlot函数可以展示感兴趣基因的表达量,也是发文章比较喜欢用的图,默认的图形如下:
虽然看起来也可以,但是看起来没有很惊艳,如果对于颜色和外形做点修饰,会更加吸引眼球,为自己文章增色不少!
1. #dotplot的改造 2.DotPlot(PBMC, features = markers)+coord_flip+theme_bw+ #去除背景,旋转图片 3. theme(panel.grid = element_blank, 4. axis.text.x=element_text(angle= 90,hjust = 1,vjust= 0. 5))+ #文字90度呈现 5. scale_color_gradientn( values= se q(0,1,0.2),ccolours = c( '#330066', '#336699', '#66CC66', '#FFCC33'))+ #颜色渐变设置 6. labs( x=NULL, y=NULL)+guides(size=guide_legend(order= 3))
以上就是单细胞基础作图的一些修饰,只要用心,多看文章,向高分文章看齐,让自己的文章也更加显眼和惊艳!数据分析很重要,可视化也是非常重要的一部分,呈现效果好的话,对于自己的文章和数据都是锦上添花的作用。












