
裸花紫珠Callicarpanudiflora Hook. et Arn为马鞭草科紫珠属植物,干燥叶入药,主产于海南、广东、广西等省份,是海南省道地药材和黎药大品种,为《中国药典》2020年版唯一新增收载药材,具有消炎、解肿毒、化湿浊、止血等功效[1]。课题组前期对海南岛裸花紫珠种质资源进行调查,发现生产上尚无优势品种,人工栽培种苗大部分来源于自留种,长期以来会导致种质混杂,药材产量与质量参差不齐[2],因此对现有种质资源进行鉴定、厘清种质种源亲缘关系,对于后续指导裸花紫珠药材新品种选育显得尤为重要。
DNA分子标记是研究种质资源遗传多样性及分子辅助育种的重要手段,主要包括随机扩增多态性DNA标记(random amplified polymorphic DNA,RAPD)、扩增片段长度多态性(amplified fragment length polymorphism,AFLP)、简单重复序列(simple sequence repeats,SSR)等[3]。早期的RAPD、AFLP等传统标记技术由于多态性信息含量低、分型不稳定、重复性差不适合做种质资源鉴定,而SSR分子标记因其多态性高、重复性好的特点被广泛应用于遗传多样性分析和亲缘关系鉴定等[4]。近年来,SSR分子标记逐步应用于裸花紫珠研究当中,于福来等[5]对裸花紫珠进行基因组调研及SSR特征分析,发现其单、双、三核苷酸重复序列占比较高。彭云露[6]利用MISA软件对6份裸花紫珠进行SSR位点搜索,筛选出22对引物进行多态性分析,发现在分子水平上的变异较小,因使用的样本数量较少,难以全面反映出裸花紫珠基因型差异。
鉴于此,本研究扩大样本数量,基于基因组和转录组信息,利用SSR分子标记对来自我国3省23县(市)共计103份裸花紫珠种质进行遗传多样性分析,建立了裸花紫珠DNA指纹图谱,为裸花紫珠种质鉴定、优异种质筛选以及分子辅助育种等提供理论依据,并为后续充分发掘利用裸花紫珠种质资源、选配育种亲本等工作奠定基础。
1材料与仪器
1.1材料
1.1.1药材实验样品为来自3省23县(市)的103份裸花紫珠野生种或半野生种,集中种植于农业部儋州热带药用植物种质资源圃,由中国热带农业科学院热带作物品种资源研究所于福来副研究员鉴定为马鞭草科紫珠属植物裸花紫珠C. nudiflora Hook. et Arn,其中93份来自于海南,5份来自于广东,5份来自于广西,具体信息见表1,103份种质中不同编号代表来自不同乡镇、村落的裸花紫珠种质。

1.1.2试剂2×Taq PCR Master Mix(美国基因泰克公司),DL 2000 DNA Marker(美国基因泰克公司),琼脂糖(美国基因泰克公司),10 µL/200 µL移液吸头(爱思进生物技术有限公司),96孔PCR板(爱思进生物技术有限公司),磁珠法基因组DNA提取试剂盒(天根生化有限公司),普通引物/荧光标记引物(天一辉远有限公司),GeneScan 500 LIZ Size Standard(美国应用生物系统公司),POP-7 Polymer(美国应用生物系统公司),Hi-Di Formamide(美国应用生物系统公司)。
1.2仪器
Mikro 120型台式离心机(MIKRO公司),Veriti 96型PCR仪(美国应用生物系统公司),DYY-6C型电泳仪(北京六一生物科技有限公司),Gel Doc TM XR+型紫外分析仪(美国伯乐公司),King Fisher™ Flex型核酸提取仪(赛默飞有限公司),3730XL型基因分析仪(美国应用生物系统公司)。
2方法
2.1总DNA提取与检测
采集供试材料的幼嫩叶片,液氮冷冻保存,使用磁珠法基因组DNA提取试剂盒(天根生化)提取103份裸花紫珠嫩叶样品的总DNA,使用Nano DROP 8000超微量分光光度计检测核酸浓度和纯度,A260/A280均在1.8~2.0,表明DNA提取纯度较高,最终调整DNA浓度为50~200 ng/L用于进一步研究。
2.2SSR引物的合成、筛选与PCR扩增
依据课题组前期测序结果,由天一辉远生物科技有限公司合成384对SSR引物。选择编号分别为1、2、8、12、50、54、61、77、81、86的10份遗传背景差异较大的裸花紫珠种质的混合DNA样本,对合成的SSR引物进行初步筛选,并对初筛引物进行复筛和验证,最终筛选出14对条带清晰、多态性高及稳定性好的核心引物(表2)。

SSR的PCR扩增反应体系(10 μL)为:2×Taq PCR Master Mix 5.0 µL,基因组DNA(~20 ng)1 µL,上游引物(浓度10 pmol/μL)0.5 µL,下游引物(浓度10 pmol/μL)0.5 µL,ddH 2O 3.0 µL 。PCR反应在Veriti 96 PCR仪(Applied Biosystem)上进行。反应程序为:95 ℃预变性5 min;95 ℃变性30 s,62~52 ℃梯度退火30 s,72 ℃延伸30 s,10个循环,每个循环下降1 ℃;95 ℃变性30 s,52 ℃退火30 s,72 ℃延伸30 s,25个循环;72 ℃延伸20 min,4 ℃保存。
2.3扩增产物检测
荧光PCR扩增完成后,取3 µL荧光PCR产物利用1%琼脂糖凝胶电泳检测,检测PCR条带是否单一、片段大小是否与预期一致。条带单一且大小相符的,对照DNA Marker的浓度进行定量,将所有产物稀释至相同的浓度范围。取10 µL甲酰胺和0.5 µL内标混匀,加入1 µL PCR稀释产物,上ABI 3730xl仪器进行毛细管电泳检测。
2.4数据处理及分析方法
将电泳结果导入GeneMarker分析软件中,进行基因型数据的读取,并按位点名称分别导出Excel基因型原始数据和PDF分型峰图文件。利用GenALEx 6.502将SSR分子标记的结果转化为txt文本格式。Popgene 32 [7] 计算各SSR位点的等位基因数(number of alleles,Na)、有效等位基因数(effective number of alleles,Ne)、观测杂合度(observed heterozygosity,Ho)、期望杂合度(expected heterozygosity,He)、Shannon's信息指数(shannon's information index,I)以及基因多样性指数(nei's gene diversity index,H),PowerMarker 3.25 [8] 计算PIC值。NTSYS-version 2.10e [9] 计算遗传距离,并绘制遗传距离三维图。MEGA 6.06 [10] 绘制环形遗传聚类图谱。GenAlEx 6.502 [11] 绘制主坐标分析图。
3 结果
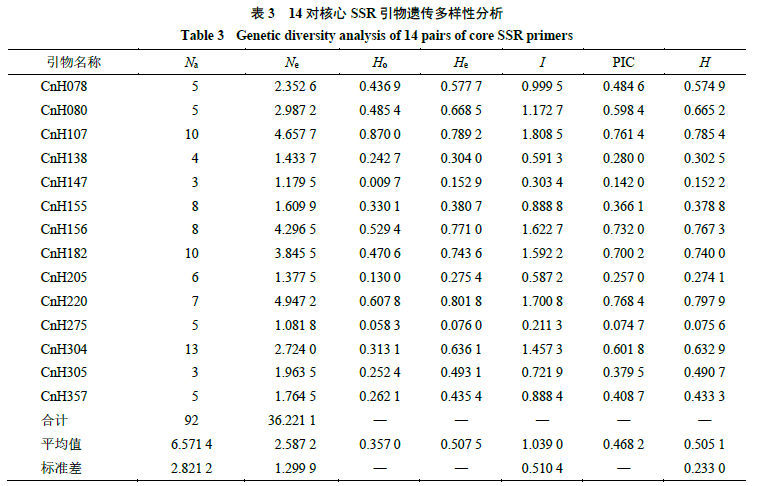
3.1SSR引物遗传多样性分析
对所筛选出的14对引物进行PCR扩增,通过DNA分析仪对扩增产物进行分析,准确获得裸花紫珠不同种质在各位点等位基因的片段大小及相应的 电泳图,部分检测结果如图1所示。14对核心SSR引物参数见表3,103份样品中共扩增92个Na,平均每对引物扩增6.571 4个,变化范围3~13个;Ne变化范围在1.081 8~4.947 2,Ne变异占比39.37%;I范围在0.211 3~1.808 5,其中4对引物达到1.5以上;He变化范围在0.076~0.801 8,平均为0.507 5(He>0.5),提示供试材料遗传多样性较高[12];Ho变化范围在0.009 7~0.87,平均为0.357 0,仅1个引物的He小于Ho外,其余引物He均大于Ho,这说明供试种质内杂合子过剩现象少[13];平均多态性信息含量(polymorphism information content,PIC)值变化范围在0.074 7~0.768 4,平均为0.468 2,具有中度多态性(0.25<PIC<0.5),其中6对引物具有高度多态性(PIC>0.5),6对引物为中度多态性(0.25<PIC<0.5)。本研究中,高PIC值的引物占42.9%,表明这些SSR位点可从分子水平解释基因型差异,具有丰富的遗传差异性,可作为有效标记来构建裸花紫珠种质DNA指纹图谱。
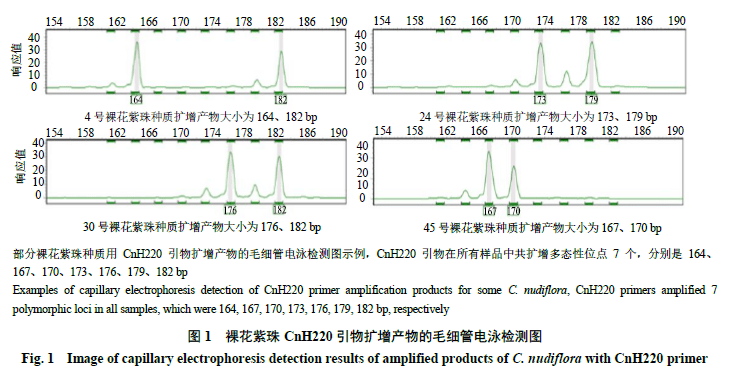
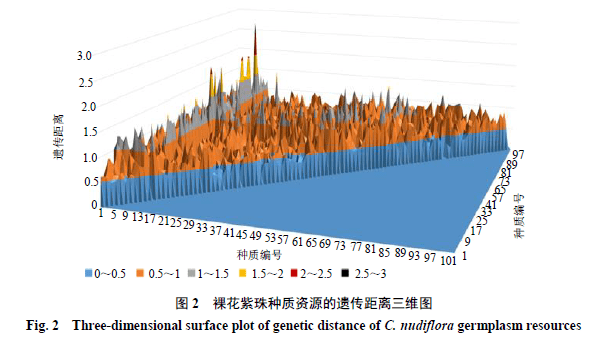
3.2裸花紫珠种质资源遗传距离分析
基于NTSYS-pc 2.10软件计算103份裸花紫珠种质间遗传距离及构建遗传距离矩阵,绘制三维曲面图(图2),103份供试材料间遗传距离范围在0~2.791 7,大多数种质遗传距离在0.5~1.0,平均遗传距离为0.634 2,遗传距离较大,表明各供试材料间具有丰富的遗传多样性。其中编号为93(海南昌江县)的种质和编号为2(海南五指山市)的种质遗传距离最大(2.791 7),表明2个种质间遗传背景差异大,亲缘关系远。另外,来自广东的种质平均遗传距离为0.644 3,来自广西的种质平均遗传距离为0.661 3,来自海南的种质平均遗传距离为0.616 5,3个省份供试材料间平均遗传距离差异较小,表明遗传距离分析准确性强,可信度高。
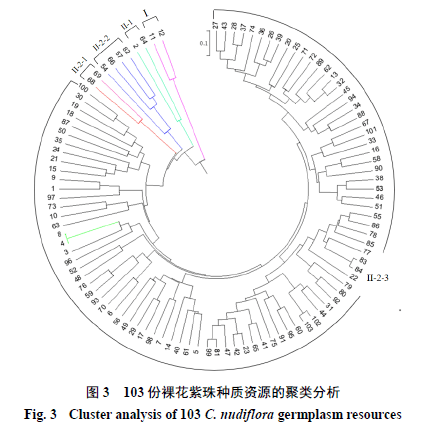
3.3裸花紫珠种质资源的聚类分析
根据种质间遗传距离,利用UPGMA法对供试材料进行分类并得到聚类图(图3)。结果表明,101份裸花紫珠种质被有效区分,编号4和编号8的种质(均来自海南白沙)未被区分开,聚类在可信度0.3515处将103份种质分为2大类(类群I、类群II)。类群I包括2份裸花紫珠种质,分别是编号11和编号12(均来自海南五指山);类群II被分为2个亚类群(II-1和II-2):亚类群II-1包括编号2(海南白沙)与编号64(海南东方)的种质,亚类群II-2包括其他99份种质,在可信度0.093 7处再次将亚类群II-2分为3个小类群,小类群II-2-1包括编号为68和编号69的种质(均来自海南东方),小类群II-2-2包括编号为54(海南乐东)、66(海南万宁)、57(海南海口)、82(广西南宁)的种质,小类群II-2-3含括其他93份种质,包括来自广西北海和广西钦州的4份、广东省5份以及海南省18市(县)的84份种质。从聚类分析中可以看出,有些位于同一地区的种质并未完全聚在一起,而一些不同地区的则被聚为一类,这可能是由于不同地区种质进化来源相同,也有可能是不同种质在长期自然选择过程中存在基因交流的现象。
3.4裸花紫珠种质资源的主成分分析
为了更好地验证裸花紫珠种质间的遗传关系,利用GenAlEx 6.502对供试材料进行主成分分析,以第1主成分和第2主成分为二维坐标图的横坐标和纵坐标,构建二维主成分分析图(图4),其中主成分1解释率为12.33%,主成分2解释率为8.96%。主成分分析将103份种质划分为3个类群,每一类群均分布在不同象限内。
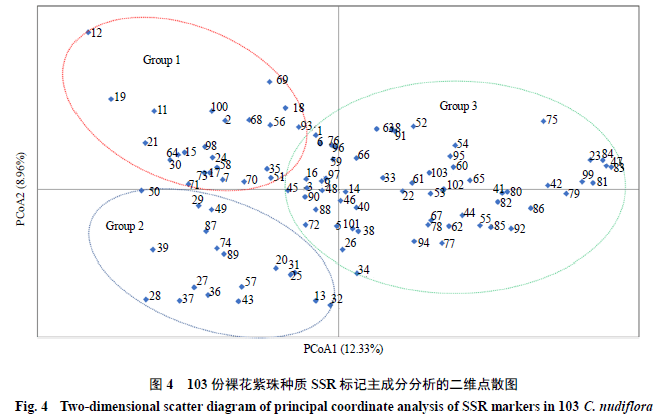
第1类包括24份种质,主要分布在第2象限,其中包含聚类分析中类群I的2份种质、亚类群II-1的2份种质、小类群II-2-1的2份种质、小类群II-2-2的4份种质以及小类群II-2-3中的16份种质。第2类主要分布在第3象限,共聚集18份种质,包含小类群II-2-2中的1份种质以及II-2-3中的17份种质。第3类材料最为丰富,共61份种质,在4个象限中均有分布,但在第1、4象限分布较为集中,包括聚类分析小类群II-2-2中的3份种质以及II-2-3中的58份种质,其中来自广东、广西的10份种质被聚集在一起。主成分分析从不同角度直观反映出供试材料间的亲缘关系,且种质在主坐标二维图中分布均匀,表明其遗传多样性较为丰富[15-16],聚集在一起的裸花紫珠材料代表种质间遗传背景相似,亲缘关系较近。本研究将PCoA主成分分析与UPGMA聚类分析相结合,发现类群内对具体种质的分布高度一致,两者分析结果可以相互佐证。
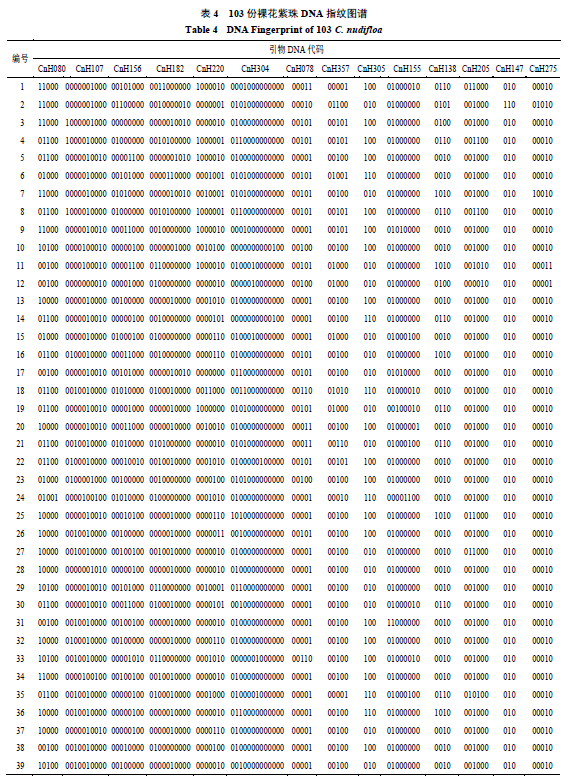
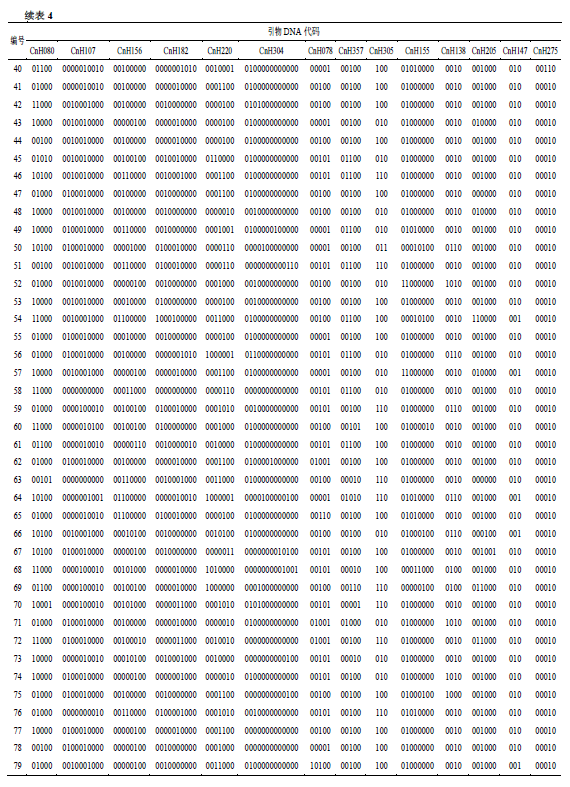
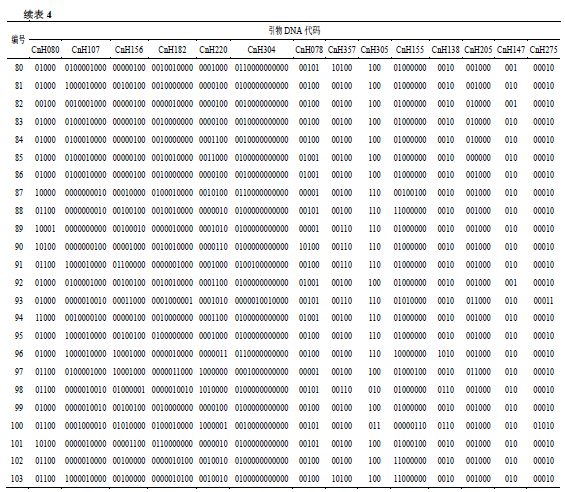
3.5 构建裸花紫珠DNA指纹图谱
DNA指纹图谱表现形式采用闵学阳等[17]的方法,根据扩增结果将有条带、无条带分别记为“1”和“0”,根据Botstein [18]提出的理论,指纹分析无法仅用1对引物将某个种质与其他种质区分开,因此需要利用引物组合的方式进行区分。对14对引物进行多样性分析后,选取具高度多态性的引物区分所有种质,如不能全部鉴别,增加一个引物进行区分,直到将所有种质区分开。从表3可以看出,CnH080、CnH107、CnH156、CnH182、CnH220、CnH304引物多态性信息含量高(PIC>0.5),引用上述6对引物组合共区分出99个种质,还有4份种质未被区分。因此,综合PIC值和等位基因数等因素,增加CnH078引物进行鉴别,共区分出101个种质,将14对引物全部转换成指纹图谱(表4),仍有2个种质(编号4和编号8)无法完全区分开,这与聚类分析结果一致,这表明该2份材料可能为同一种质,也可能与供试种质较多,引物鉴别力下降有关[19]。本研究首次利用14对引物构建出101份裸花紫珠种质资源DNA指纹图谱代码,使每份种质都有各自唯一的一套DNA指纹,可在分子水平上辅助解决裸花紫珠同名异种或者同种异名等种质混乱现象[20],也使裸花紫珠种质鉴定更加准确高效。
4 讨论
4.1SSR引物有效性评价
SSR标记是用来鉴定品种真实性和遗传多样性的有力工具[21],遗传多样性水平高可代表物种群体健康、适应性强[22],而SSR引物有效性高则可以更好地评价物种遗传多样性。本研究利用14对SSR引物对来自不同省份的103个裸花紫珠种质进行遗传多样性分析,较前人研究[6]极大的扩增了样本数量,且来源几乎涵盖所有裸花紫珠的主产区,样本具有较好的代表性。分析获得的Na平均值(6.571 4)和PIC平均值(0.468 2)较高,说明引物多态性高、有效性强,有利于不同遗传背景供试种质的区分,在种质遗传多样性分析中表现出较大潜力。研究检测到的Na值较高(92个),Ne占比较大(39.37%),这可能与供试种质来源广、类型丰富有关,且采用的方法精确、灵敏,能够有效评价裸花紫珠遗传多样性。基于以上分析,本研究所筛选的引物有效性强,区分效果好,使得裸花紫珠种质鉴别更加准确,为后续裸花紫珠遗传变异检测及杂交育种奠定试验基础。
4.2 裸花紫珠遗传关系
供试材料间的遗传距离可以直观反应种质的遗传多样性,间接体现出种质间遗传背景相似度。赵雅楠等[23]利用10对多态性引物对92份内蒙古绿豆进行遗传距离分析,发现内蒙古绿豆遗传多样性丰富,个体差异水平较高。郑涛等[24]利用12对SSR引物对12份血叶兰进行遗传分析,发现种质间遗传距离最大为0.74,研究有效验证了种质间亲缘关系与遗传距离密切相关。本研究供试材料的遗传距离在0~2.791 7,遗传距离变异幅度大,遗传背景差异明显。少部分来源相同的种质遗传距离较近,如来自海南三亚的102号和103号种质遗传距离为0.052 7,来自广西钦州的83号和84号种质遗传距离仅为0.032 3,原因可能是一些地理来源较近的种质经过长时间自然驯化,导致遗传变异下降。可见SSR分子标记能够在一定程度上反应种质地理来源情况。从遗传距离分析,有些来源不同的种质遗传距离却比较接近,如来自海南临高的13号种质与来自海南儋州的89号种质遗传距离仅为0.160 2,这可能与种质具有相似的环境背景或常年引种有关,导致个别种质在进化过程中出现DNA分子差异水平下降的情况。因此,在今后的育种工作中一方面要结合分子标记技术开展各地方种质遗传多样性研究,发掘亲缘关系相对较远的种质;另一方面要加大种质资源的收集及保存,拓宽裸花紫珠育种的遗传背景。
4.3裸花紫珠类群结构
物种的群体遗传结构与物种的起源进化、自然分布等有很大关系,对研究物种之间的进化关系有很大帮助[25]。Feng等[26]运用SSR技术对40份野生黄精进行聚类分析,认为野生黄精群体间遗传差异小,主坐标分析有效地验证了聚类分析的结果。Iqbal等[27]运用SSR技术对13个橄榄品种进行聚类分析及主坐标分析,明确了13份材料间亲缘关系的远近,证明橄榄品种间遗传变异在一定程度上收到地理环境的影响。本研究将UPGMA聚类分析及主坐标分析相结合,发现各类群呈现一定地域相关性,即相近地区材料间遗传关系较近,来自同一地区的种质聚集在一起的几率较大,如图3中类群II-2-3将来自广东省的所有种质以及广西省的4份种质均聚集在了一起,图4的主成分 分析将广东的5份种质聚为一类。不同地区来源种质也存在相互聚集的情况,如类群II-2-2中聚集了海南岛的3份种质和广西的1份种质,主成分 将广东的5份种质和广西的5份种质聚集在一起,这体现出该种质可能来源于相同(相似)的祖先,或在发展种植过程中不同地理来源的种质为适应环境从而发生基因交流的现象。未被聚集在一起的种质代表种质间亲缘关系较远,遗传背景差异大,因此,在今后对裸花紫珠进行种质改良时,可以利用亲缘关系远、品质性状优的种质进行杂交选育,既可获得遗传差异丰富的后代,又能在一定程度上提高裸花紫珠种质遗传多样性。
4.4 裸花紫珠DNA指纹图谱构建
目前,构建植物分子指纹图谱主要有特征谱带法、引物组合法和单引物法3种方式[28]。其中,引物组合法一般用于种质数量多的指纹图谱构建,该方法不仅可以高效、快速地鉴别种质,而且极大地提高引物的鉴别能力。目前,SSR标记在裸花紫珠指纹图谱构建中的应用尚未见报道,鉴于此,本次研究运用SSR标记技术,利用14对高多态性引物,采用引物组合法,首次构建了103份裸花紫珠种质DNA指纹谱图,并精准鉴别出101份裸花紫珠种质。然而研究中选用的14对引物并未将103个种质全部区分,4号种质与8号种质的指纹图谱不具备唯一性(表4)。究其原因,一方面可能是由于多态性引物的开发还不够全面,另一方面可能与所收集的该种质地理来源相近、种质间遗传背景相似有关。
未来随着裸花紫珠种质收集范围的逐渐扩大与种质信息的不断更新,本研究中的SSR引物可能会无法全部鉴别,因此应进一步优化SSR引物或采用更多的多态性引物组合,为DNA指纹图谱应用于裸花紫珠种质鉴别提供方便快捷的记录方式的同时,扩展更加完善的指纹图谱。
本研究利用14对SSR标记对裸花紫珠种质资源进行遗传多样性分析,结果表明,每个SSR标记平均6.571 4个Na,PIC平均为0.468 2,说明引物多态性较高,鉴别能力强,能够精准评价所有种质的遗传多样性。对供试材料进行遗传距离分析发现,遗传距离在0~2.791 7,遗传变异幅度大,遗传背景差异明显。聚类分析将供试材料分为2大类群,类群II又分为2个亚类,与主成分分类结果基本一致,两者有效验证了种质间亲缘关系的远近。利用14对引物组合首次建立103份裸花紫珠DNA指纹图谱,能够精准鉴定101份种质,区分全面并具代表性,对裸花紫珠种质溯源管理及原产地保护具有重要意义。
综上所述,本研究印证了SSR标记技术应用于裸花紫珠遗传多样性分析的可行性,这些SSR标记不仅为裸花紫珠遗传多样性分析和DNA指纹图谱构建提供有价值的候选标记,也为后续裸花紫珠种质分子鉴别、优良种质的分子辅助育种提供分子技术手段。
利益冲突所有作者均声明不存在利益冲突
参考文献(略)
来 源:张红瑞,李 鑫,陈振夏,胡文斌,李 伟,黄 梅,于福来,杨林立.基于SSR分子标记的裸花紫珠种质资源遗传多样性分析及DNA指纹图谱构建 [J]. 中草药, 2023, 54(12):3971-3982.
- 本文固定链接: https://www.maimengkong.com/zu/1639.html
- 转载请注明: : 萌小白 2023年10月15日 于 卖萌控的博客 发表
- 百度已收录
