近期,中国农业科学院畜牧研究所分别在DNA Research以及Scientific Reports上发表了关于贵州小型猪circRNA及lncRNA的相关研究。两篇文章分别从lncRNA和circRNA两方面进行系统的鉴定、特征分析等,构建了全面的基因转录图谱,并进一步揭示了转录后调控的机制。值得注意的是,两篇文章都是基于同一批样本一次建库获得的数据结果。1次测序,2篇文章产出,怎么实现的呢?
下面小编结合2篇文章的主要内容及整体研究思路,了解作者是如何设计文章的。
思 路 总 结
首先选取贵州小型猪9个组织(心脏,肝脏,脾脏,肺,肾,卵巢,睾丸,肌肉和脂肪)以及3个发育时期(出生后0d,30d,240d)的骨骼肌样本分别构建去rRNA的链特异性文库,一次性获取了mRNA、lncRNA以及circRNA共3部分的数据,并进行了全基因组范围内lncRNA及circRNA的鉴定和特征分析,保守性分析,以及组织、时空特异性分析,关联分析等。

结 果 展 示

猪lncRNA时空表达及遗传保守性分析
S.scrofa lncRNA鉴定及特征分析
鉴定了10,813个lncRNA,其中多数(78%)为基因间区转录本(lincRNAs)。特征分析显示,S.scrofa lncRNA平均长度、外显子数量以及表达量显著低于蛋白编码基因。
S.scrofa lncRNA 组织及时空特异性分析
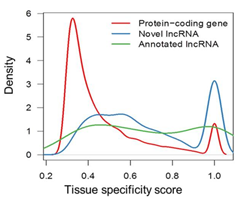
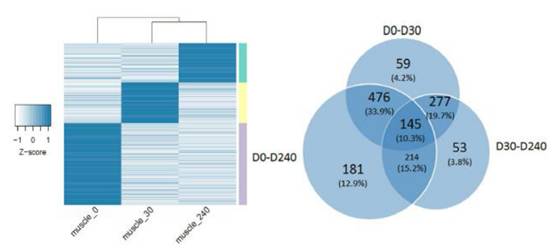
lncRNA组织特异性明显高于蛋白编码基因,其中睾丸和卵巢组织中组织特异性lncRNA占比最高(68.8%),表明lncRNA在生殖发育过程中具有潜在作用。通过比较3个不同发育阶段骨骼肌lncRNA的变化,筛选出1405个发育特异性lncRNA。

S.scrofa lncRNA保守性分析
与人鼠lncRNA相比较,6213个lncRNA在三物种中保守。物种保守的lncRNA往往比S.scrofa特异性lncRNA具有更高的组织特异性。另外,睾丸组织中含有最高比例的保守性lncRNA(~50%)。
保守性lncRNA及蛋白编码基因共表达网络分析
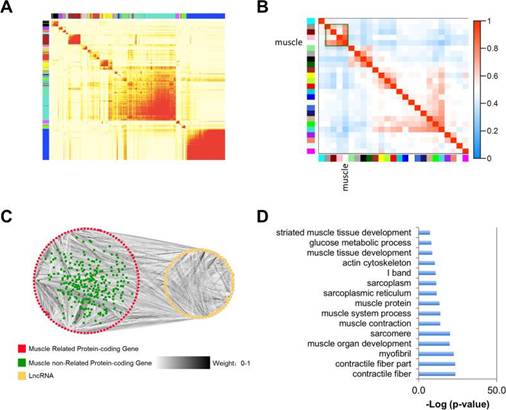
保守性lncRNA及相关蛋白编码基因WGCNA分析显示,共聚成25个基因模块,筛选相关性最强的模块进行分析。GO富集分析显示,S.scrofa lncRNA共表达的蛋白编码基因主要参与骨骼肌发育过程。

不同组织及发育时期猪circRNA分析
circRNA鉴定及特征分析
鉴定5,934个circRNA,circRNA大多处于编码区(68.40%)和基因间区(21.93%),当然也有部分在UTR区域。家猪的circRNA中位数为509bp,人的是547bp。71.5%的circRNA 长度短于2Kb,GC含量为42.67%。
家猪、人和小鼠circRNA序列保守性分析
多于20.2%的猪circRNA跟人具有直系同源性,16.96%跟小鼠具有同源性,表明家猪的circRNA跟小鼠和人的circRNA发挥着相同的功能。
S.scrofa circRNA 潜在作为miRNA的海绵结构
circRNA可能是在调控RNA表达时发挥miRNA的海绵作用。miRNA和circRNA的结合位点分析共鉴定出1842个circRNA跟miRNA进行结合(符合三个预测软件标准的)。
circRNA 组织特异性表达及骨骼肌发育特异性
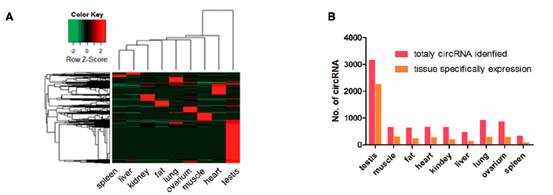
circRNA相对表达水平较低,具有明显的组织特异性,其中睾丸的circRNA是脾脏的九倍以上。GO及KEGG结果显示,心脏中特异性circRNA与肌肉发育、细胞分化以及心肌病、心肌收缩、wnt信号通路等相关。
不同发育阶段肌肉组织中共鉴定出1489个circRNA ,其中D0与D240的circRNA更相近。骨骼肌中最丰富的circRNA起源于骨骼肌生长和肥大相关的蛋白编码基因。

S.scrofa circRNA数据库的构建
整合circRNA注释、circRNA表达、潜在的circRNA功能和circRNA-miRNA-基因相互作用,最终构建了S.scrofa的circRNA数据库,为哺乳动物circRNA研究提供全面的数据信息。
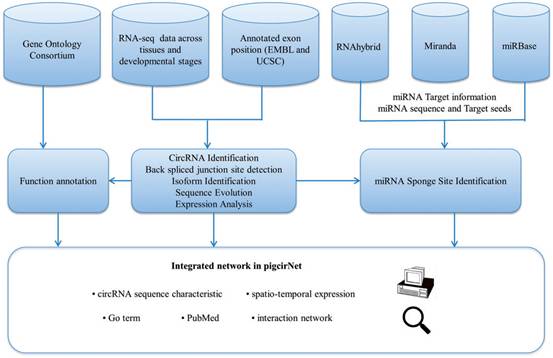
安诺基因全转录组测序技术,通过构建去rRNA链特异性文库,可一次性获取mRNA、lncRNA以及circRNA 三部分数据分析结果,包含非编码RNA鉴定、特征分析,保守性分析,组织/时间特异性分析以及WGCNA分析等多样化的分析内容,并可免费赠送ceRNA关联分析内容,私人定制个性化分析等,轻松揭示RNA分子表达模式及转录后调控机制。如此高性价比的测序文章策略,您值得拥有~
参考文献:
[1] Tang Z, Yang W, Yang Y, et al. Comprehensive analysis of long non-coding RNAs highlights their spatio-temporal expression patterns and evolutional conservation in Sus scrofa [J]. Sci Rep, 2017, 7:43166.
[2] Liang G, Yang Y, Niu G, et al. Genome-wide profiling of Sus scrofa circular RNAs across nine organs and three developmental stages.[J]. Dna Research An International Journal for Rapid Publication of Reports on Genes & Genomes, 2017.
文案:RNA产品经理 王亚男- 本文固定链接: https://www.maimengkong.com/zu/1603.html
- 转载请注明: : 萌小白 2023年8月14日 于 卖萌控的博客 发表
- 百度已收录
