大家好,这里是专注表观组学十余年,领跑多组学科研服务的易基因。
在全球范围内,牛为60多亿人提供了重要的营养来源。传染病是养牛生产的主要限制因素,且许多疾病人畜共患,因此与人类健康直接相关。近年来牛的许多复杂性状遗传基础得到了广泛研究,但不同动物的大量重要表型变化仍无法解释,人们越来越关注非遗传变异(包括基因表达、DNA甲基化和染色质)与重要牛表型的潜在相关性。为充分了解牛性状的非遗传成分、基因与环境互作的重要性以及遗传变异如何通过甲基化影响表型,有必要分析整个基因组中的染色质和甲基化状态,并了解其在动物和品种中的变化方式和位置。
2023年05月22日,爱丁堡大学皇家 (迪克) 兽医学院Liam J. Morrison和 James G. D. Prendergast团队在《Genome Biology》杂志发表了题为“Profiling the immune epigenome across global cattle breeds”的研究论文,该研究通过使用简化甲基化测序(RRBS)、ATAC-seq、RNA-seq绘制了Holstein Friesian(欧洲taurine)、 N’Dama(非洲taurine)和 Nelore(印度indicine)三个不同牛品种的DNA甲基化、染色质可及性和RNA表达谱。

标题:Profiling the immune epigenome across global cattle breeds(分析不同牛品种的免疫表观基因组)
时间:2023.05.22
期刊:Genome Biology
影响因子:IF 17.906
技术平台:RRBS、ATAC-seq、RNA-seq
样本实验:
Holstein Friesian牛(欧洲taurine)、 N’Dama牛(非洲taurine)、Nelore牛(印度indicine)三种不同雌性牛品种(每个品种三头)的七个主要血液免疫细胞类型(B细胞、CD4 αβ T细胞、CD8 αβ T细胞、γδ T细胞、NK细胞,单核细胞和粒细胞)
研究摘要:
了解对当地环境和病原体适应良好和适应性差的牛品种间差异对于培育具有改良气候和抗病表型的牛至关重要。尽管在鉴定品种间的遗传差异方面已经取得了相当大的进展,但表观遗传和染色质水平变化仍未表征。本研究以单碱基分辨率生成、测序和分析了150多个文库,以研究三个不同牛谱系中牛免疫系统的DNA甲基化和染色质可及性变化。研究结果表明发现taurine牛和indicine牛在免疫细胞类型上存在广泛的表观遗传差异,这与两个牛谱系之间的局部DNA序列差异水平有关。特异性细胞类型谱能够使用数字细胞术对复杂的细胞混合物进行反卷积。最后,根据CpG岛的染色质和甲基化谱分析了CpG岛的不同亚群类别,这些亚群区分了与离散转录状态相关的远端岛和基因近端岛。
本研究提供了三个不同牛品种的DNA甲基化、染色质可及性和RNA表达谱的综合资源。可能对设计有效的非欧洲品种牛表观基因组广泛关联研究产生不同的影响。
研究结果
(1)染色质可及性、DNA甲基化和转录组免疫图谱的生成和验证
分别以Holstein Friesian牛、N'Dama牛和Nelore牛的7种主要血液免疫细胞类型(B细胞、CD4αβT细胞、CD8αβT细胞、γδT细胞、NK细胞、单核细胞和粒细胞)为靶点,使用RRBS和ATAC-seq分析全基因组DNA甲基化和染色质可及性(图1A)。由于N’Dama牛品种在样本采集和细胞分离中的挑战,该品种的4个ATAC-seq和3个RRBS样本被排除在研究之外。因此,三个品种九头牛的B细胞、CD4αβT细胞、γδT细胞和单核细胞数据总共产生了119个文库。为了在下游分析中进行比较,还为来自Holstein Friesian牛和Nelore牛的细胞类型子集生成了26个RNA-seq文库。
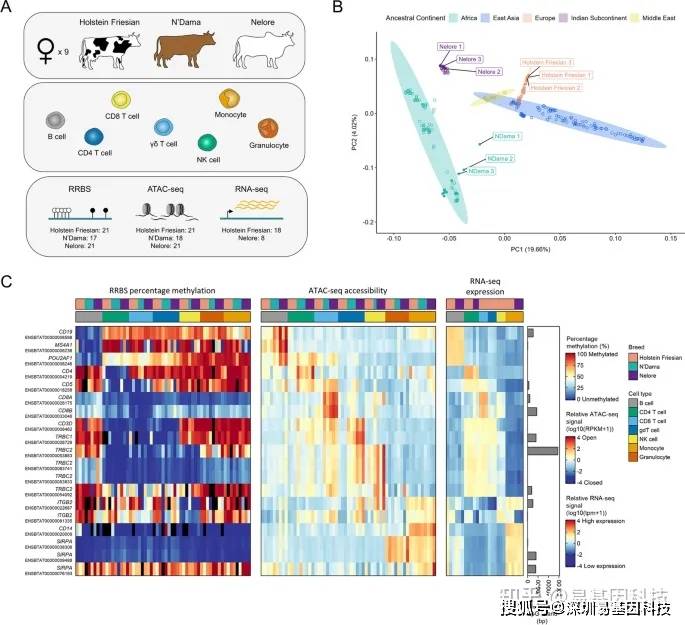
图1:三个遗传多样性牛品种原代血细胞中染色质景观研究
- 从Holstein Friesian牛、N'Dama牛和Nelore牛(每个品种三头)共九头牛中收集的RRBS,ATAC-seq和RNA-seq细胞类型概述示意图。底部显示了每个品种收集的RRBS、ATAC-seq和RNA-seq样品总数。牛基因型数据的主成分分析(PCA)。使用298头牛的27379808个变体进行分析。免疫相关基因的甲基化水平、染色质可及性和基因表达。热图显示免疫相关基因启动子的甲基化(左)和染色质可及性(中)及其相应表达(右)(TSS上游1000 bp和下游500 bp)。右侧条形图显示了每个转录本的TSS与最近CGI的距离。甲基化热图中的黑条表示在给定启动子中发现少于5个CpG位点被至少5个reads覆盖的样品。SIRPA表示SIRPα,gdT细胞表示γδT细胞。该图表明与以前的报道一致,CD4+CD8+双阳性T细胞的数量可能很少,因为CD4和CD8表达在很大程度上靶向各自细胞类型。
- 与预期一致,ATAC-seq数据显示片段长度具有明显的周期性,与跨越不同核小体数量的片段一致。RRBS数据在CpG岛(CgIsland,CGI)显著富集,所有样本中CpG岛约56%的CpG位点至少有10reads覆盖,另25%位于CGI附近2 kb区域的CGI海岸(图2A)。相比之下,只有约1%的参考基因组含有CGI。与许多含有CGI的基因启动子一致,48%的CpG位点位于启动子区域(图2B)。
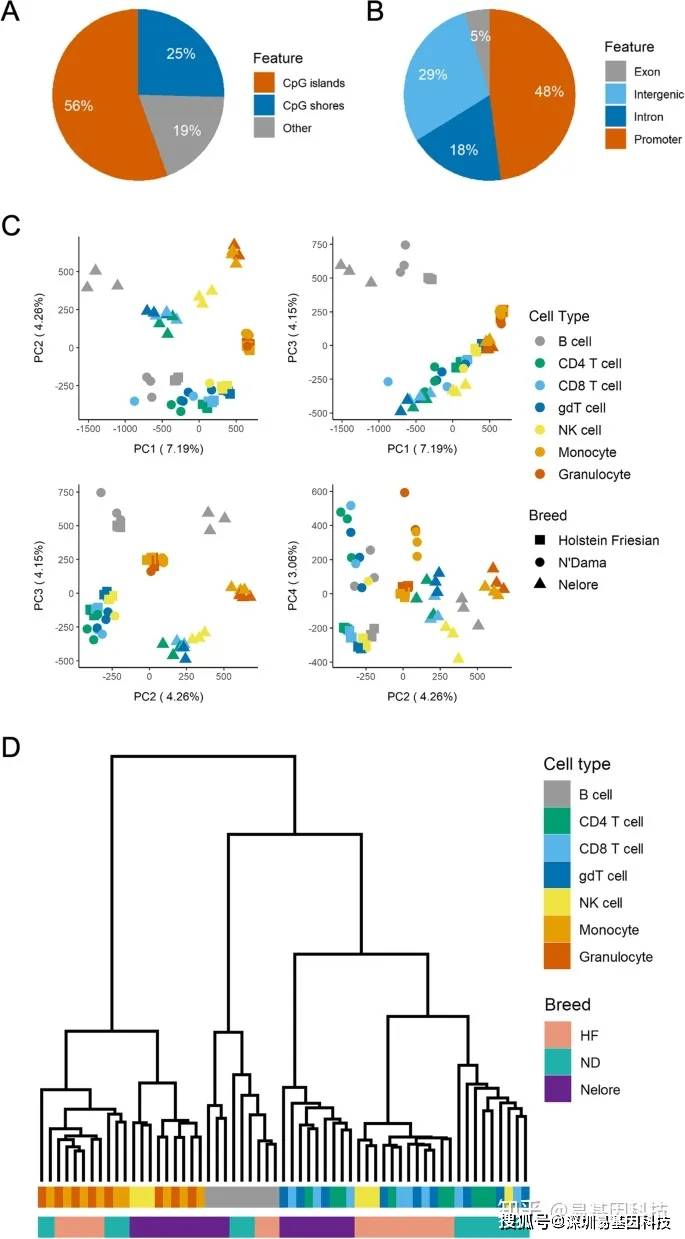
图2:细胞类型和牛谱系之间的DNA甲基化差异。所有样本的CpG位点(9084个位点)中至少有10reads覆盖位点。
A~B. CpG位点注释与定义的基因组特征重叠。当CpG位点与多个基因组特征重叠时,优先顺序:启动子>外显子>内含子。
C. CpG位点甲基化水平的PCA分析。
- 基于CpG位点甲基化水平的样品无监督分层聚类。根据Spearman相关系数对样本进行聚类,其中前50% CpG位点在所有样本(4542个位点)中表现出最高标准偏差。gdT细胞表示γδT细胞
(2)细胞类型和牛谱系之间的差异甲基化
通过对细胞类型、牛品种和牛谱系的成对比较来分析样本的CGI甲基化水平。对24598个CGI进行分析,其中3036个CGI在至少一个比较中存在显著差异(q值≤0.01,甲基化水平差异≥25%)。成对比较中差异甲基化CpG岛(DMI)的共存如图3A。这些DMI中的大多数(2675)是在B细胞与另一种细胞类型的比较中鉴定出来的。总的来说,在至少一个品种或谱系的比较中鉴定出348个DMI,与欧洲和非洲两个taurine品种相比,200个DMI在indicine中具有特异性甲基化谱。
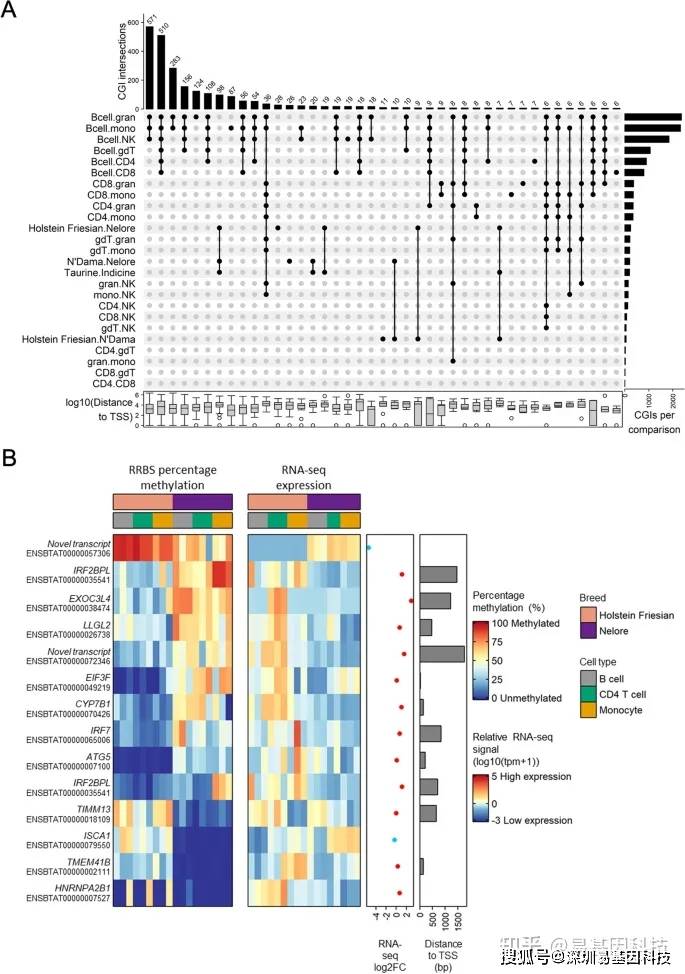
图3:细胞类型、牛品种和牛谱系之间差异甲基化CGI的比较。
- 在标记的成对比较中鉴定出差异甲基化CGI(甲基化差异≥25%,q值≥0.01)的Upset plot图。右侧条形图显示了每次比较中显著差异甲基化CGI数量。顶部注释条显示了在黑点指示比较中显著差异甲基化的CGI数量。仅显示至少6个显著差异甲基化CGI的比较集。底部箱形图显示了CGI在每个组合集中到蛋白质编码基因的最近TSS距离分布。左侧文本表示在每个成对比较中比较的组,其中“.”将组1和组2分开。Holstein Friesian牛(欧洲taurine)、 N'Dama牛(非洲taurine)、Nelore牛(印度indicine)之间差异甲基化的CGI热图。CGI仅限TSS 2000 bp内,且在所有样品中最近基因的平均表达均>10 TPM。RNA-seq log2FC是平均Holstein Friesian RNA-seq信号和平均Nelore RNA-seq信号之间的log2倍变化,其中正log2FC(红点)表示Holstein Friesian牛中高表达,负log2FC(蓝点)表示Nelore牛中较表达。Bcell =B细胞,CD4=CD4 T细胞,CD8=CD8 T细胞,gdT=γδT细胞,NK=NK细胞,mono =单核细胞,gran =粒细胞
(3)牛谱系之间遗传和表观遗传差异之间的相关性
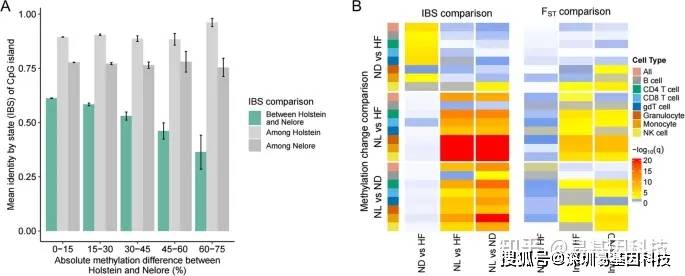
图4:种群间甲基化差异升高的CpG岛平均显示遗传差异升高。
A、CGI状态的平均同一性表明,将所有细胞类型放在一起比较,Holstein Friesian和Nelore品种之间的甲基化差异水平不同。显示平均值的标准差。与这对种群之间的其他CGI(绿色)相比,甲基化差异相对较高的CGI通常也显示出较高的遗传差异(IBS较低)。相反,种群间的甲基化差异在很大程度上与个体群体内计算的IBS评分无关(灰色)。
B。IBS(左)或FST评分(右)与细胞类型和群体比较甲基化差异之间的关联。每个细胞表示特定细胞类型中的甲基化差异与群体比较(行)以及成对群体(列)之间的遗传差异之间的关联强度。黄色/红色显示显著关联(校正FDR P<0.05),白色/蓝色显示不显著结果。HF=Holstein Friesian,ND=N'Dama,NL=Nelore,Ind=Bos indicus
(4) 将CpG岛分为六个簇,每个品种具有不同的表观遗传谱
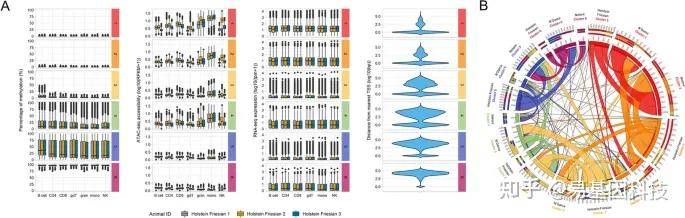
图5:CGI的无监督聚类分析鉴定出不同的染色质景观。
A、Holstein Friesian牛数据中 CGI甲基化水平和ATAC-seq信号聚类分析(RPKM)。聚类采用期望最大化(EM)算法拟合的有限高斯混合模型(GMM)进行。每个CGI仅配对一个TSS附近基因。显示每个CGI附近基因的RNA-seq表达以及每个CGI与TSS附近距离。簇通过增加中位甲基化水平进行排序,并根据此顺序编号。
B、 Circos图显示品种对之间不同聚类的CGI重叠程度。优先顺序如下:Holstein Friesian>N'Dama>Nelore。最外层标签表示聚类分析中使用的品种数据,后跟聚类编号。最外面条形图显示属于其他两个品种聚类的每个品种中CGI的相对重叠。
(5)基于DNA甲基化谱的复杂细胞混合物反卷积
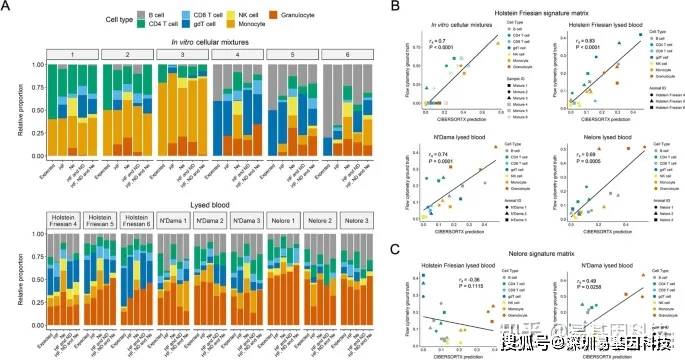
图6:基于DNA甲基化谱的细胞混合物反卷积
- 每个体外混合物和裂解血液样本中细胞亚群相对比例条形图。体外细胞混合物,“expected”是指用于生成混合物的FACS纯化细胞类型的已知比例。对于裂解的血液样本,“expected”是指通过流式细胞术分析的每个细胞亚群的比例。每个混合物的后续四条显示CIBERSORTx使用指定品种的参考样本分析的每种细胞类型比例。
B~C. 散点图比较流式细胞术与CIBERSORTx值,其中仅使用Holstein Friesian(B)或Nelore(C)参考样品预测混合物组成。通过Spearman秩相关(rs)和线性回归(实线)鉴定一致性。gdT细胞表示γδT细胞
易基因小结:
本研究生成了全面的数据集,为七种免疫细胞类型和三个牛品种的DNA甲基化、染色质可及性和基因表达互作提供了见解。本研究数据将有助于鉴定不同细胞类型、不同牛品种和物种的保守或不同的表观遗传模式和调控机制。这将有助于理解进化谱系表型变异的过程,在功能上验证与性状相关的遗传变异,并有助于牛的遗传改良计划,以培育具有更强大免疫系统的动物。
关于易基因简化基因组甲基化测序(RRBS)研究解决方案
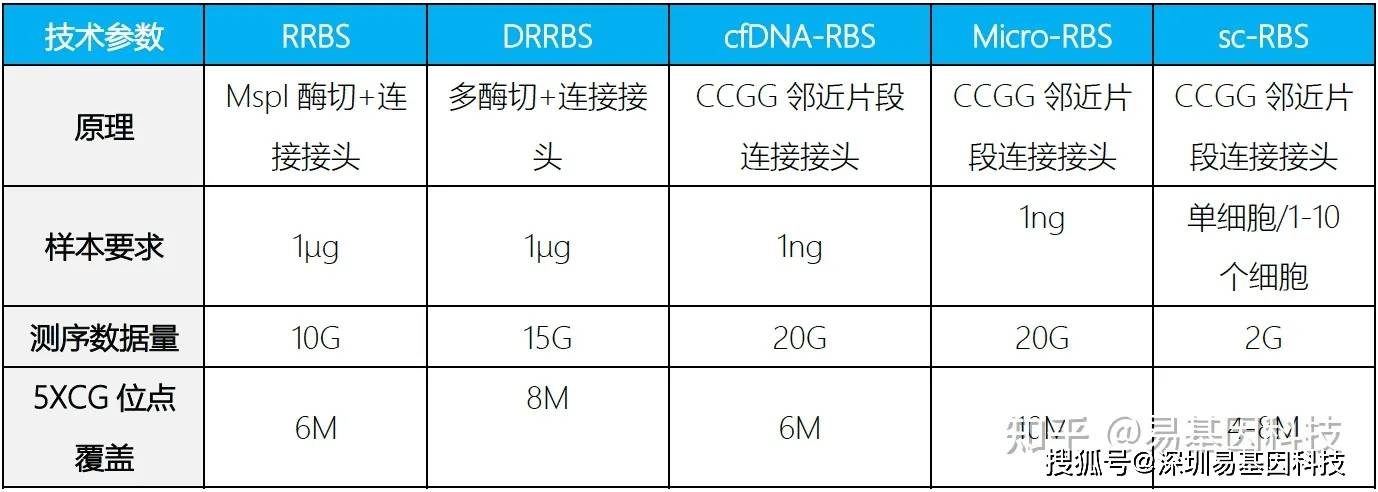
简化甲基化测序(Reduced Representation Bisulfite Sequencing,RRBS)是利用限制性内切酶对基因组进行酶切,富集启动子及CpG岛等重要的表观调控区域并进行重亚硫酸盐测序。该技术显著提高了高CpG区域的测序深度,在CpG岛、启动子区域和增强子元件区域可以获得高精度的分辨率,是一种准确、高效、经济的DNA甲基化研究方法,在大规模临床样本的研究中具有广泛的应用前景。
为适应科研技术的需要,易基因进一步开发了可在更大区域内捕获CpG位点的双酶切RRBS(dRRBS),可研究更广泛区域的甲基化,包括CGI shore等区域。
为助力适用低起始量DNA样本(5ng)量多维度甲基化分析,易基因开发了富集覆盖CpG岛、启动子、增强子、CTCF结合位点的甲基化靶向基因组测序方法:extended-representation bisulfite sequencing(XRBS),实现了高灵敏度和微量样本复用检测,使其具有高度可扩展性,并适用于有限的样本和单个细胞基因组CG位点覆盖高达15M以上。
技术优势:
- 起始量:100ng gDNA;单碱基分辨率;多样本的覆盖区域重复性可达到85%-95%、测序区域针对高CpG调控区域,数据利用率更高;针对性强,成本较低;基因组CG位点覆盖高达10-15M,显著优于850K芯片。
应用方向:
RRBS/dRRBS/XRBS广泛应用于动物,要求全基因组扫描(覆盖关键调控位点)的:
- 队列研究、疾病分子分型、临床样本的甲基化 Biomarker 筛选复杂疾病及肿瘤发病机制等甲基化研究模式动物发育和疾病甲基化研究
易基因科技提供全面的DNA甲基化研究整体解决方案。
参考文献:
Powell J, Talenti A, Fisch A, Hemmink JD, Paxton E, Toye P, Santos I, Ferreira BR, Connelley TK, Morrison LJ, Prendergast JGD. Profiling the immune epigenome across global cattle breeds. Genome Biol. 2023 May 22;24(1):127.
转自:深圳易基因
- 本文固定链接: https://www.maimengkong.com/zu/1564.html
- 转载请注明: : 萌小白 2023年6月16日 于 卖萌控的博客 发表
- 百度已收录
