做转录组分析时,我们通常会筛选差异表达基因进而对这些差异表达基因进行功能富集分析(下面简称常规富集分析)。不知道大家有没有遇到过以下情况:差异基因少而富集不出来感兴趣或相关的功能/通路,或者差异表达基因虽然很多,但是没有富集到感兴趣的通路或者GO功能?此时,可以试试GSEA分析。
![]()
那么问题来了,GSEA是什么?
全名Gene Set Enrichment Analysis,也就是基因集富集分析!GSEA是一个计算的方法,用来确定是否一个预先定义的基因集,能在两个生物学状态中显示出显著的一致性的差异。

![]()
Oh no!什么预定义基因集,什么生物学状态,什么一致性差异,这都什么鬼?
预先定义的基因集:首先它是一个基因集合,它包含的是感兴趣的基因,比如某个通路,某个GO term,或hall marker基因集两个生物学状态:即实验组和对照组,可以是癌症和正常,男和女一致性差异:也就是预定义的基因集中的基因在两个生物学状态中呈现出相似的差异状态;说白了就是某个通路/GO条目中的基因集在实验组和对照组中呈现出一致的上调或者下调趋势~

GSEA与常规富集分析的区别在哪里呢?(敲黑板,划重点)
常规富集分析必须先做差异筛选,用筛选的基因(无论多少)进行功能富集,这种方式可能由于筛选参数的不合理导致漏掉一些关键信息。
而GSEA无需做差异分析,直接拿所有基因的表达量即可找到实验组和对照组有一致性差异的感兴趣的通路。好处就是,不经筛差异可以保留了这些关键信息,进而找到那些差异不很明显但是基因差异趋势很一致的功能基因集。
当然,常规富集分析和GSEA分析没有说哪个更好,实际应用中能解决问题即可,引用一句名言:黑猫白猫,能抓住老鼠的就是好猫
那么问题又双叒来了,我该怎么用?
终于唠叨到了正题,小编的絮叨症又犯了!



安装篇
windows/mac用户可以打开GSEA网站:
http://software.broadinstitute.org/gsea/index.jsp
点击Download进行下载应用程序(要先注册再登录)。可以点击launch下载桌面小程序,或者点击download gsea-3.0.jar 文件。前者双击后直接会在桌面生成一个快捷方式,后者是一个jar文件放在哪里就直接双击即可使用。当然前提还是要更新你的java版本呦!

双击小程序或者jar文件就可以看到GSEA软件的使用界面啦!

使用篇
文件准备
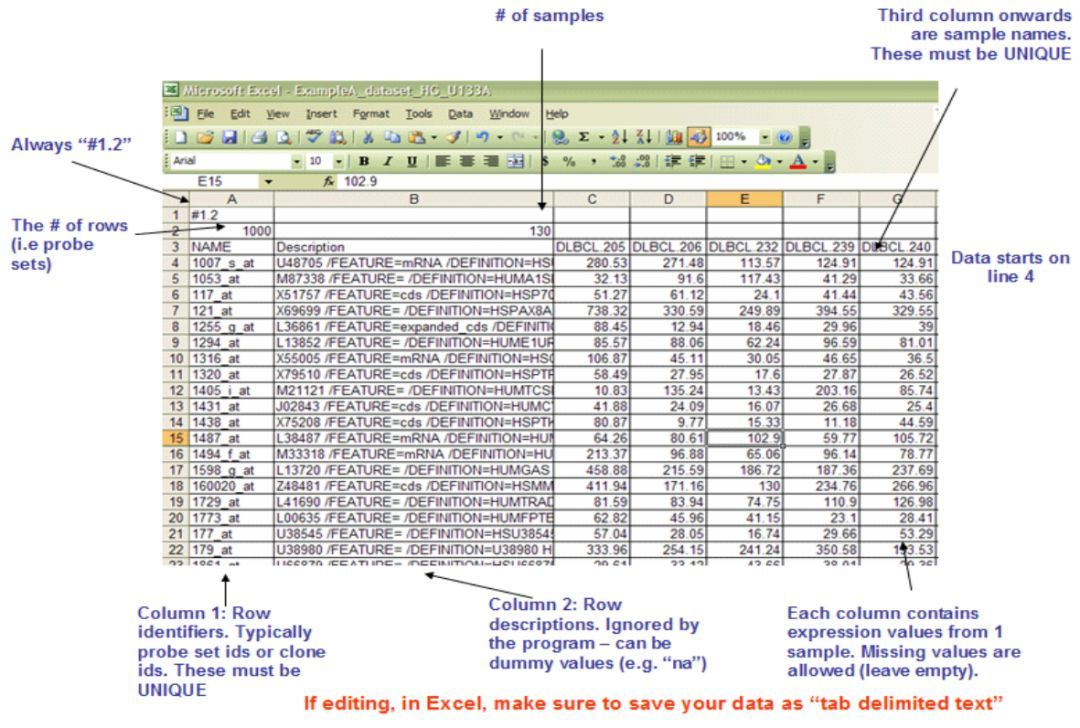
1. 样品表达量文件(res, gct, pcl, or txt)——必需文件
通常用.gct为后缀。文件第一行以“#1.2”开头;文件第二行的第一列为基因个数、第二列为样品个数;文件的第三行为表达谱的矩阵的title信息,第一列为基因symbol/探针号,第二列为基因/探针的描述信息,第三列以后为样品id。接下来的行对应每个基因/探针在每个样品中的表达信息。文件以tab作为分隔符。
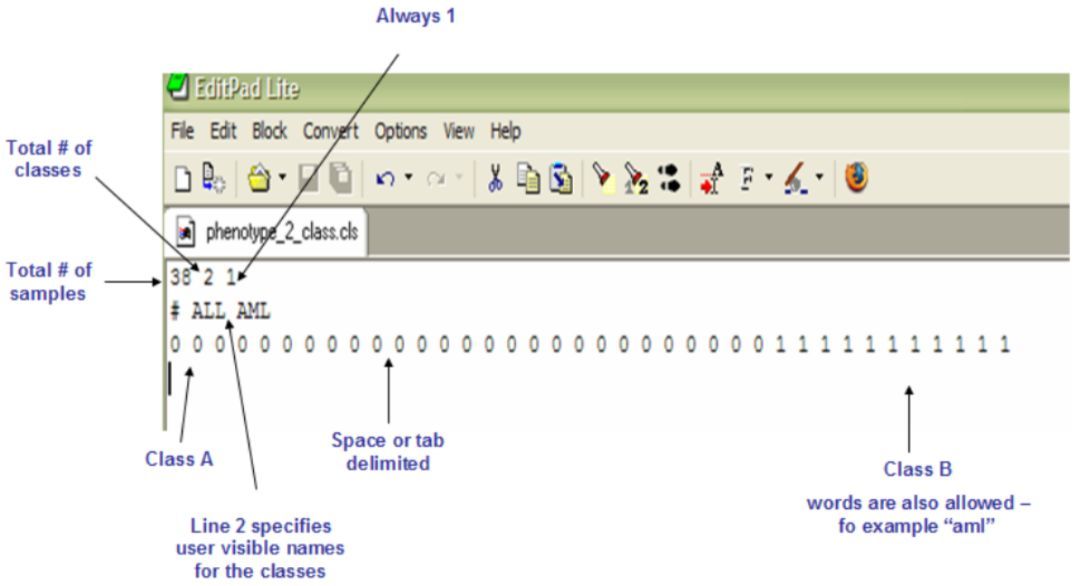
2. 样品表型分类文件(cls)——必需文件
样品表型分类文件需以.cls为后缀。文件第一行为三个数字,第一个是样品的总数,第二个是样品分为几类,第三个数字通常为1。第二行也通常三个字符串,第一个为#,第二个为分类1的名称,第三个位分类2的名称。第三行为每个样品的分类信息,0代表分类1,1则代表分类2。文件以空格或者tab分割。
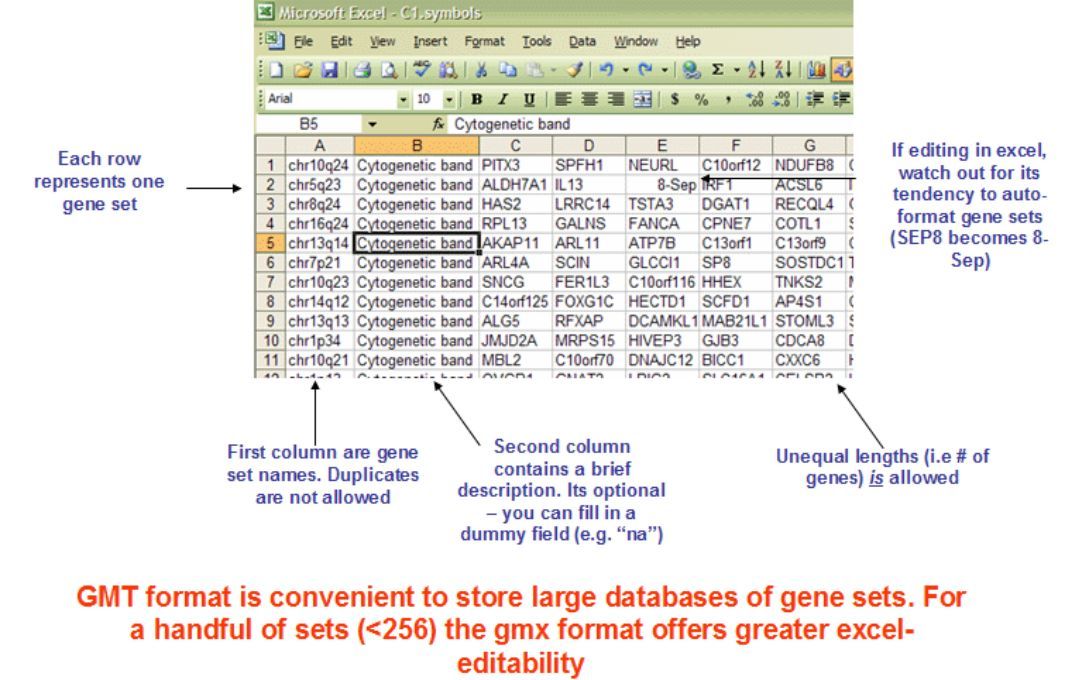
3. 预定义基因集(gmx or gmt)——非必需文件
通常用.gmt作为后缀。若采用GSEA预定义的MSigDB数据库中的功能基因集分析,则无需自己定义该文件。每一行为一个功能基因集,第一列为基因集的名称,第二列为简单描述,第三列及以后列为该功能基因集所包含的基因symbol。基因集包含多少个基因,就列出多少个基因。文件以tab作为分隔符。
开始分析
1.上传文件
首先点击左侧面板的Load data,其次在右侧面板点击Browse for files。弹出下述文件上传框,可以选择上面准备好的gct, cls等文件。
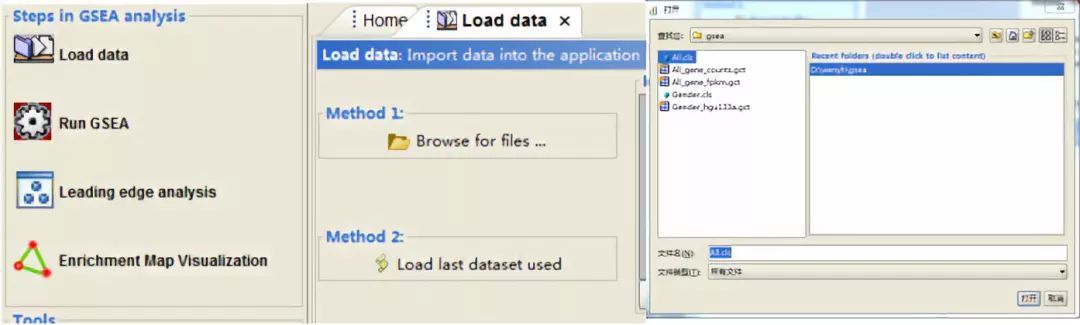
2.参数选择
点击左侧Run GSEA,接下来右侧面板会弹出参数选择框。上方的为必需的参数设置,下方的为非必需的。此处,主要讲解一下必需的参数设置。
- Expression dataset(表达文件): 选择上一步上传的表达gct文件
- Gene sets database (功能基因集数据库):GSEA包含了MSigDB数据库中的功能基因集,可以从中选择感兴趣的通路、癌症标记、转录因子数据库等。
- Number of permutations(扰动/随机次数):通常设置1000,此参数不可过小。
- Phenotypes labels(样品表型分类文件):选择上一步上传的表型cls文件
- Collapse dataset to gene symbols:通常true
- Permutation type(扰动类型): 通常选择phenotype,如果样品数目较少可以选择gene_set。
- Chip platform(芯片类型):如果表达gct文件的第一列为芯片探针id则此处需要选择对应的芯片平台,如果是基因symbol则无需选择。
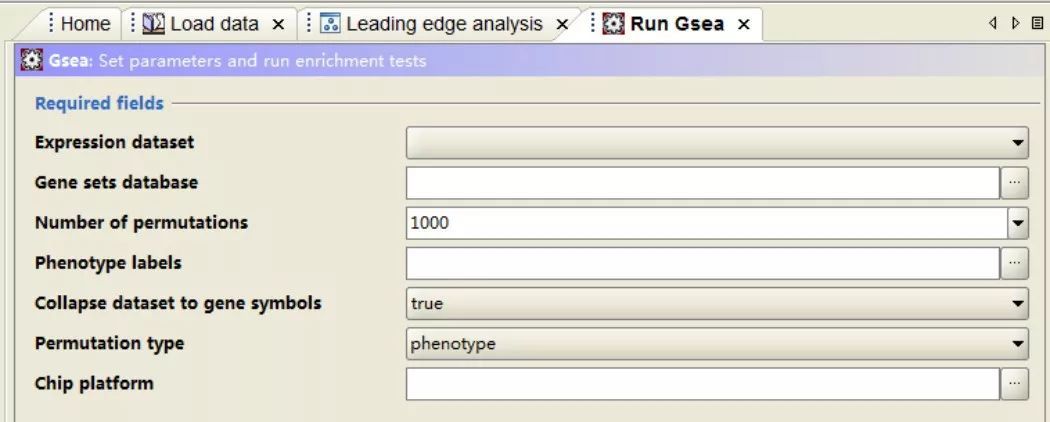
3. 运行
参数选择完毕点击右下角的Run运行GSEA。点击完成后在左下方面板会出现Gsea running字样。如果分析完成,Running状态会更改为Success状态。点击Success则可查看分析结果。这个运行时间与所选的功能基因集、扰动的次数有关。
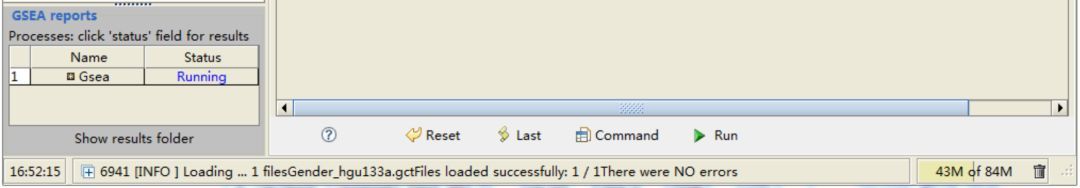
结果篇
GSEA的结果最经典的就是这张图了,那么结果怎么理解呢!
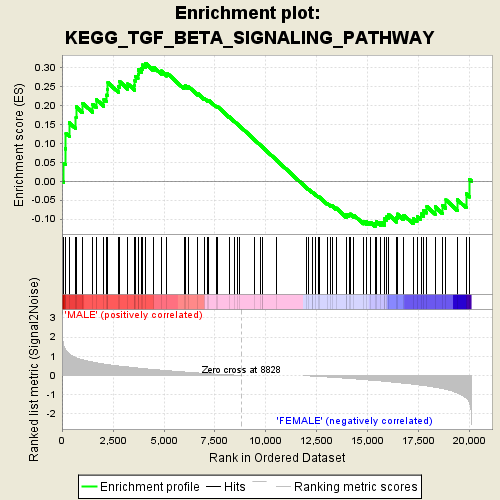
上方标题:代表该图展示的为所研究的表型在标题所示的的通路/GO条目中的富集情况。
横坐标:是从0-20000变化的,是指排序好的所有基因集。根据表达信息与表型的关联进行排序,靠近0的基因代表表达量与表型呈现正相关,靠近20000的为负相关基因。
纵坐标:上方的纵坐标是富集打分ES,这个富集打分是一个动态的值,通常用偏离0最远的值作为富集打分。下方的纵坐标代表基因表达与表型的关联,绝对值越大代表关联越强,数值大于0代表正相关,小于0则代表负相关。中间的竖线为感兴趣基因集所处的位置。
通常我们会根据pvalue或者FDR选择显著富集的这些功能基因集进行深入挖掘~~
小结
不要以为GSEA只能研究人,其他物种也是可以做哒,表达谱gct和表型cls文件都是一样的,只需要额外构建一个对应物种的功能基因集gmt文件就可以啦,上传文件的时候一并上传,在 Gene sets database 这个参数选择时选择自己上传的库信息就可以啦。只要表达谱的gene 和上传的基因集保持一致就行,无论你用symbol还是id都可以~~
上述操作是GSEA最基础的操作,关于GSEA你能做的还有很多:
- lncRNA的功能研究
- 药物敏感性与疾病的研究
。。。。

你们可爱的我,啊不,是GSEA它都能做!想知道怎么做?留言举高高小编,超过10个人留言,小编就写一个GSEA的进阶篇~~哈哈哈
转自:百迈克- 本文固定链接: https://www.maimengkong.com/zu/1252.html
- 转载请注明: : 萌小白 2022年10月6日 于 卖萌控的博客 发表
- 百度已收录
