各位医学方的朋友,大家好,我们你们的老朋友Flyman,时隔许久,今天我们又和大家见面了。今天我们和大家分享一下大家司空见惯的话题基因通路富集分析。谈到富集分析,大家首先会想到Y叔叔写的神包clusterprofiler,几行代码轻松搞定,目前主流的富集分析的算法寻找相关通路本质就是一个超几何分布,那我们今天剖析的GSEA算法则和超几何分布是略有不同。
GSEA全称是 gene-set enrichment analysis,即基因集富集分析,而不是简单地基因富集分析。上次我们和大家已经简述了GSEA的实现过程,那我们这次更深度的解析一下GSEA的算法本质,并更详细的实现相应的操作。
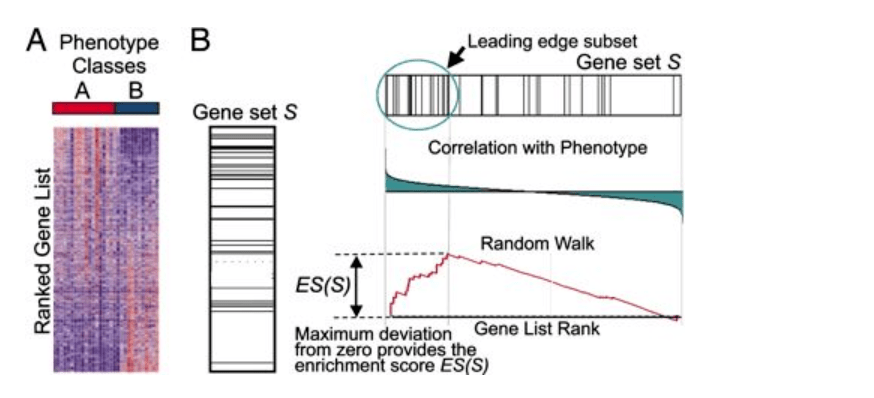
首先我们看一下上面的这张原理图,第一步我们需要准备一个基因表达量矩阵作为GSEA输入,在上图我们看到样本是可以分为A和B两组,类似我们常规的给药组和模型组以及模型组与正常组,接着对所有基因进行排序,这一步也是最关键的一步,对基因排序,一般我们是按照foldchange值从大到小排序,注意这里不是绝对值之后的排序,而是原始值的排序,用来表示基因在两组间表达量的变化趋势。比如AvsB排序之后的基因列表,大于0的基因列表表示在A组高表达,B组低表达,小于0的基因代表在A低表达,B高表达,那对于这样的基因列表,其顶部的基因可以看做是上调的差异基因,其底部是下调的差异基因。
GSEA分析的是一个基因集下的所有基因是否在这个排序列表的顶部或者底部富集,如果在顶部富集,我们可以说,从总体上看,该基因集是上调趋势,反之,如果在底部富集,则是下调趋势。
下面进入正题,基于R语言操作这里使用的是Y叔的R包clusterProfiler:
1
安装并加载包,关于包的安装,已经讲过多次,直接上代码:
2
构建基因排序信息,这里我们纳入采用内置的数据集geneList对象,该对象是包含每个基因的logFC值大小,由于该对象存在于DOSE中,所以大家也要安装好DOSE,另外该数据集如下:

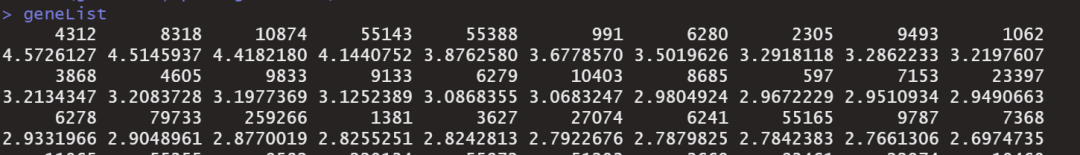
每个数值的名字属性,代表该基因的Entrz ID。数据准备好之后,我们便可以完成GSEA分析了。
3
针对KEGG的GSEA分析,如下,直接借助gseKEGG函数实现。

4
结果解读,生成的结果如下:
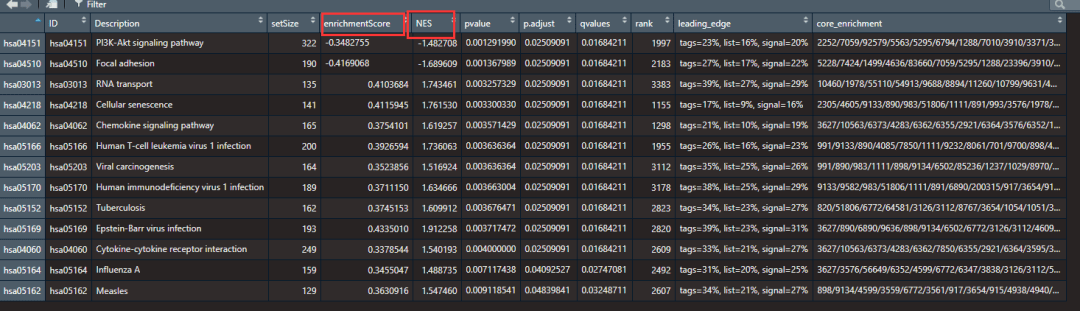
我们需要关注两列,enrichmentScore和NES列,一般这两列方向具有同号性,一般大于0可以认为该通路被激活或者高表达,小于0认为被抑制或者低表达。
5
首先结果可视化一下一条通路:
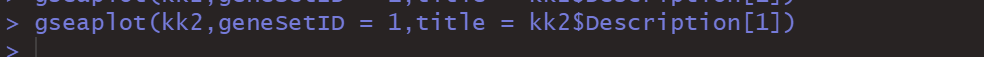
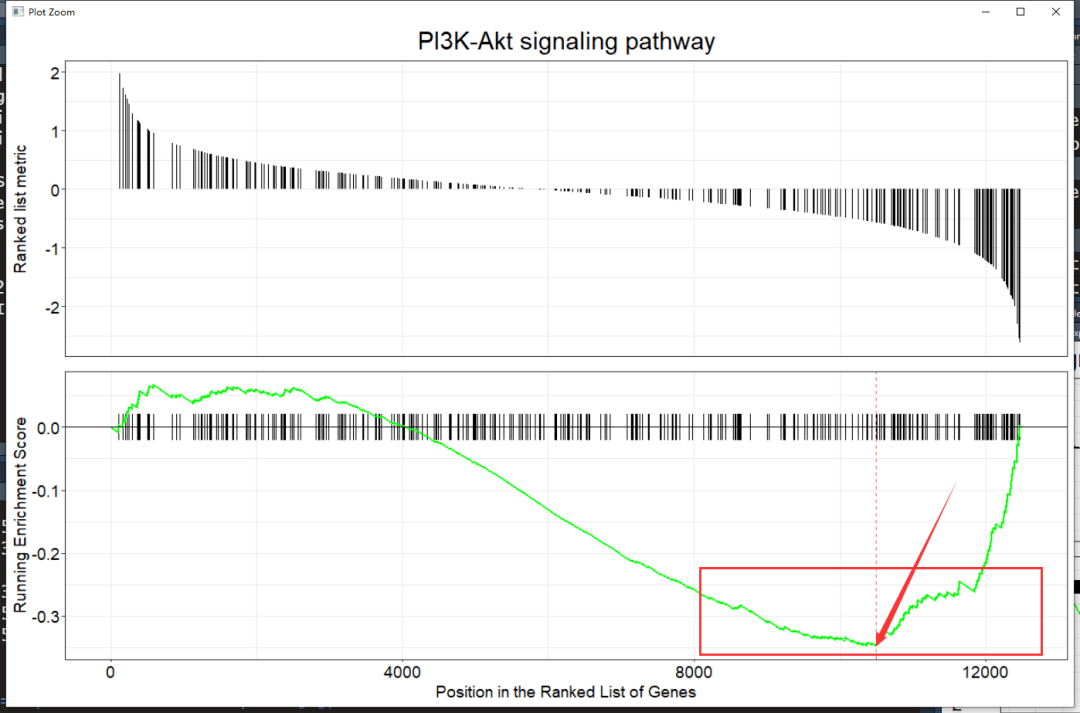
很明显当NES<0,其主要在底部富集,当NES>0,主要在顶部富集,如下:

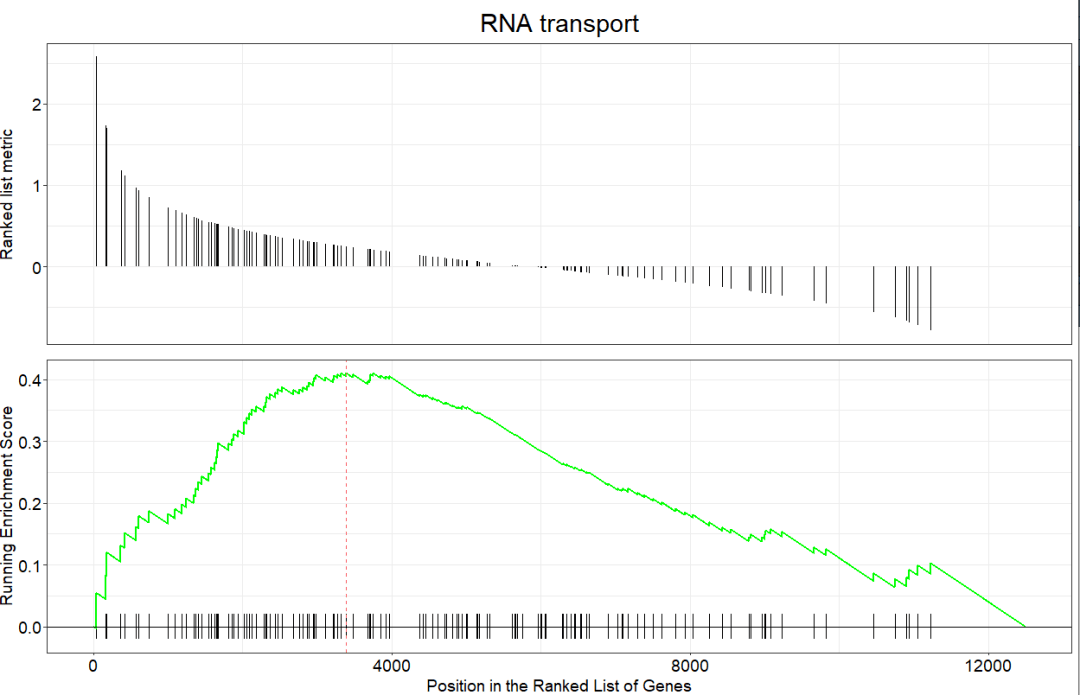
当然我们还可以进行更好看一些的颜色搭配,采用的是gseaplot2如下:

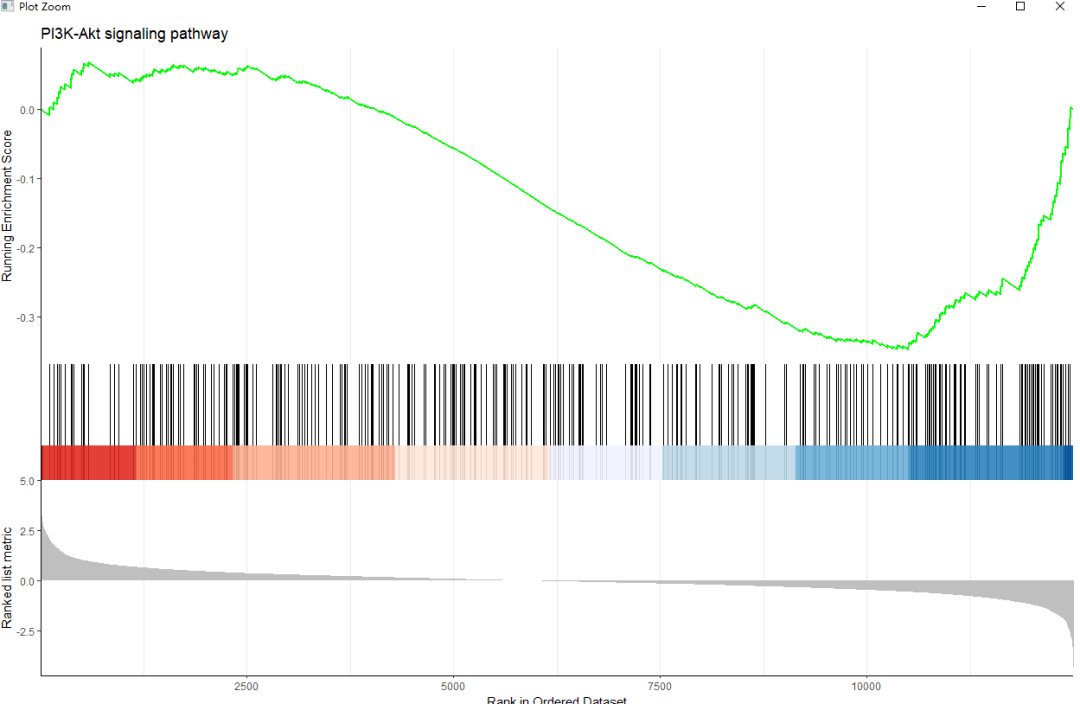
6
多条通路同时展示,如下:
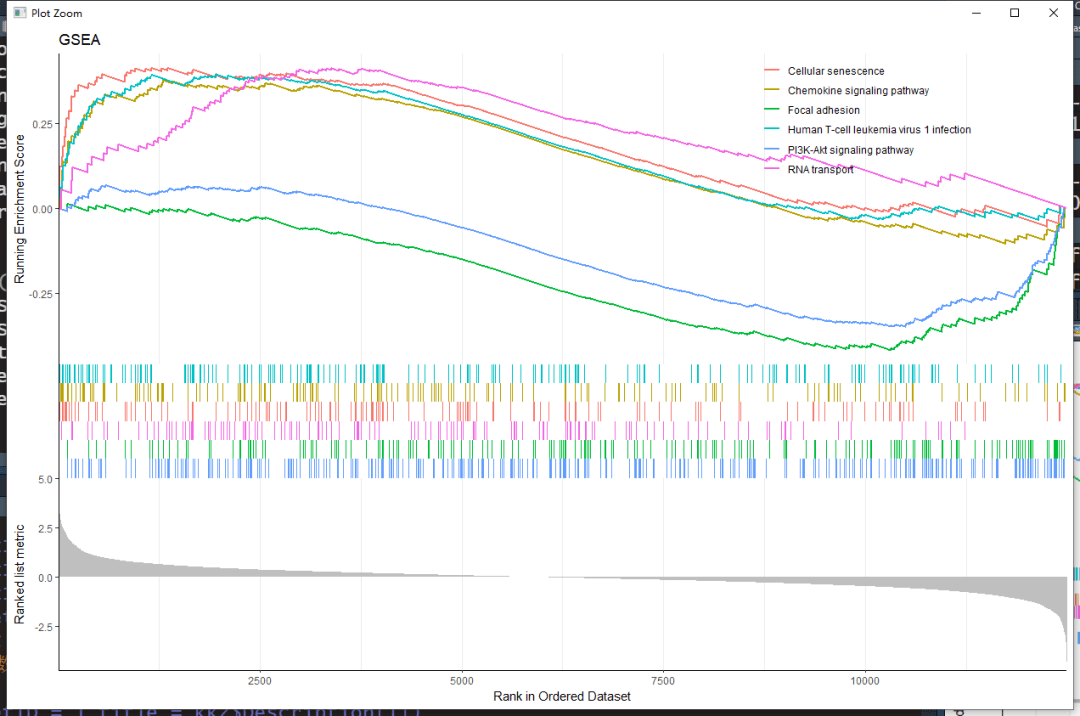

Ok,今天的推文就到这,我们分享了什么是GSEA以及如何使用clusterProfiler工具进行GSEA分析,希望能对大家有所帮助,最后,欢迎大家留言,有不正确的地方,也请大家留言指正。
END
- 本文固定链接: https://www.maimengkong.com/zu/1250.html
- 转载请注明: : 萌小白 2022年10月6日 于 卖萌控的博客 发表
- 百度已收录
