您的小可爱上线啦!“长链非编码RNA研究进展周刊”的专题连载如期更新,小编今日为大家更新第15期lncRNA研究进展周刊,走过路过不容错过!
公告通知
非编码RNA(ncRNA)是一组缺乏蛋白编码的内源RNA,分为小RNA和长链非编码RNA(lncRNA)。之前的研究多关注蛋白编码基因,对ncRNA功能知之甚少,甚至称其为转录“噪音”。然而,近年来的研究发现,ncRNA在多个生物学过程中起着重要的作用。自ncRNA概念提出以来,该领域就成为研究热点,而关于长链非编码RNA(lncRNA)的研究则是ncRNA研究的重点,相关的研究文章也在近年呈现爆发性的增长。一起浏览一下近期相关的报道吧~
1
中文标题:鉴定转录蛋白编码序列在lincRNAs内的残留
英文标题:Identification of transcribed protein coding sequence remnants within lincRNAs
期刊:NUCLEIC ACIDS RES
影响因子:11.561

长基因间非编码RNA(lincRNA)是长度>200个核苷酸的非编码转录物,其不与蛋白质编码序列重叠。重要的是,已知这些元件是组织特异性表达的,并且在数千个基因组基因座的基因调控中发挥广泛的作用。然而,对这些RNA元件的进化生物发生的机制知之甚少,特别是鉴于它们在物种间的保守性差。已经提出lincRNA可能来自假基因。
为了系统地测试这一点,美因茨约翰内斯古腾堡大学的研究人员开发了一种新方法,可以在lincRNA转录本中搜索蛋白质编码序列的残余;假设是我们可以追溯蛋白质编码基因或后转座子/反转录转座子插入的生物发生。应用该方法,他们发现了203个人类lincRNA基因,其区域与蛋白质编码序列显著相似。该方法提供了可视化工具,通过序列差异追踪lincRNA相对于蛋白质编码基因的进化生物发生。随后,研究人员利用公共RNA-seq信息库显示了lincRNA与其鉴定的亲本蛋白编码基因之间的表达相关性,暗示了新的基因调控关系。总之,他们开发了一种新的计算方法来研究非编码基因序列,可用于鉴定lincRNA的进化生物发生和功能。
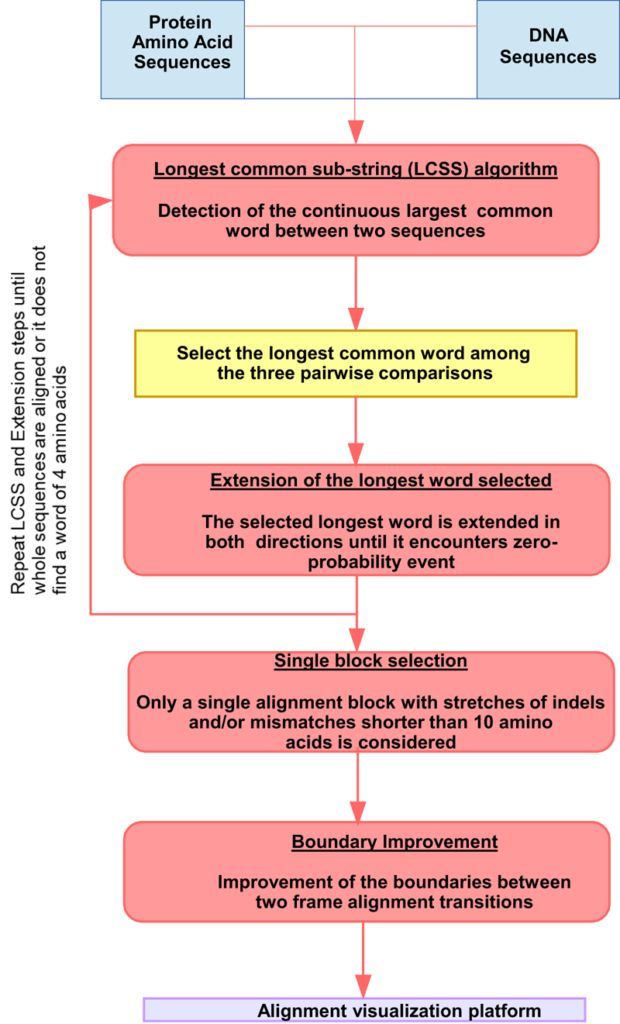
图 在lincRNA中发现蛋白质编码序列残余的方法
2
中文标题:LncPipe——用于从RNA-Seq数据中鉴定和分析长链非编码RNA的新一代流程
英文标题:LncPipe – A Nextflow-based pipeline for identification and analysis of long non-coding RNAs from RNA-Seq data
期刊:J GENET GENOMICS
影响因子:4.066

最近,长链非编码RNA分子(lncRNA)因其在多种生物过程中的关键作用以及对多种人类疾病和癌症的重要影响而受到广泛关注。鉴定和分析lncRNA是促进我们对其功能和调节机制的了解的基本步骤。然而,由于复杂的操作和实验过程,基于RNA测序的lncRNA发现受到限制。
中山大学癌症中心的研究人员开发了一种名为LncPipe的一站式流程,专注于从原始转录组测序数据中表征lncRNA。该流程是基于流行的工作流程框架Nextflow开发的,由四个核心程序组成,包括读取对齐,装配,识别和量化。它包含各种独特的功能,如精心设计的lncRNAs注释策略,优化的计算效率,多样化的分类和交互式分析报告。此外,LncPipe允许用户取消流程,从命令重置参数或直接修改主脚本并从继续检查点恢复分析。
3
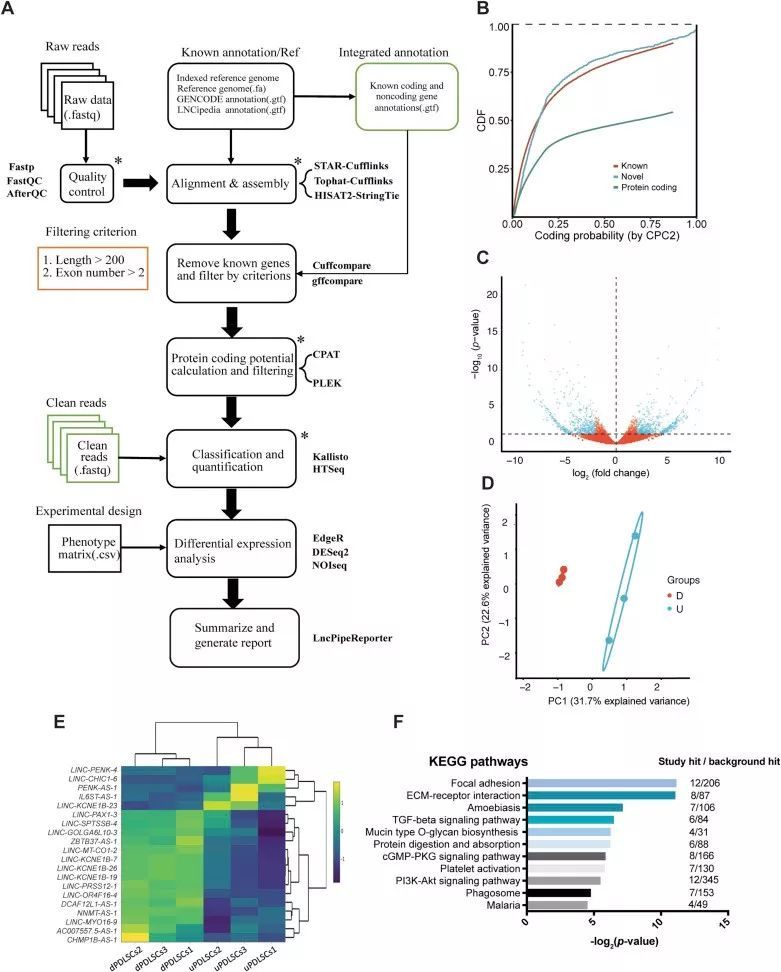
中文标题:CHO细胞中长链非编码RNA转录组全貌
英文标题:The long non-coding RNA tranome landscape in CHO cells
期刊:Biotechnol J
影响因子:3.507

近年来,非编码RNA在确定中国仓鼠卵巢(CHO)细胞的生长,生产力和重组产品质量属性中的作用受到了很多关注,特别是对miRNA的研究。然而,其他类型的非编码RNA受到的关注较少。本研究对这样一类非编码RNA进行了研究——lncRNA。
肯特大学的研究人员使用芯片进行了CHO中lncRNA转录组的第一次分析,该芯片也提供了编码转录组的监测。他们报告了在两个不同日期的批次和补料分批条件下存在于宿主CHO细胞系中的那些lncRNA,并且将不同lncRNA的表达相互关联。研究人员证明,小鼠芯片适用于检测和分析数千种CHO lncRNA,并通过qRT-PCR验证了其中的一些。然后,他们进一步分析数据,以确定那些在培养不同阶段或在批次和补料分批培养之间表达变化最大的lncRNA,以确定潜在的lncRNA靶标,用于进一步的功能研究,以及它们在控制CHO细胞生长中的作用。
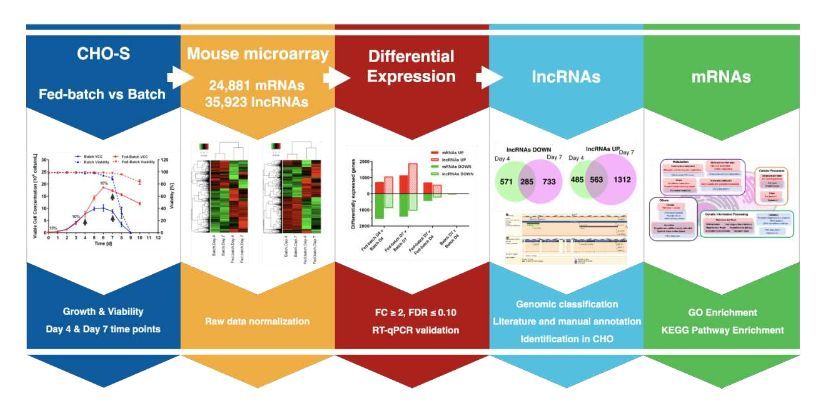
图 实验工作流程摘要
4
中文标题:使用核糖体谱数据鉴定和分析核糖体相关的lncRNA
英文标题:Identification and analysis of ribosome-associated lncRNAs using ribosome profiling data
期刊:BMC Genomics
影响因子:3.730

尽管已发现的长链非编码RNA(lncRNA)数量急剧增加,但其生物学作用尚未确定。最近的许多研究使用核糖体谱数据来评估lncRNA的蛋白质编码能力。然而,很少有人确定核糖体相关的lncRNA,这里定义为与蛋白质合成相关的核糖体相互作用的lncRNA以及其他不明确的生物学功能。
在这项研究中,来自早稻田大学的Zeng及其同事对大量已发表的核糖体谱数据集进行了广泛的分析。平均而言,观察到39.17%的表达的lncRNA与人体中的核糖体和48.16%的小鼠相互作用。作者开发了核糖体相关指数(RAI),其量化了lncRNA在各种组织和细胞类型上的核糖体相关性的证据,以分别编目与人和小鼠中的核糖体强烈相关的691和409个lncRNA。此外,研究人员分别鉴定了在人和小鼠中编码肽的高概率的78和42个lncRNA。与不含核糖体的lncRNA相比,观察到核糖体相关的lncRNA更可能位于细胞质中,并且对无义介导的衰变更敏感。结果表明,RAI可用作区分核糖体相关和游离lncRNA的整合和循证工具,为研究lncRNA功能提供了宝贵的资源。
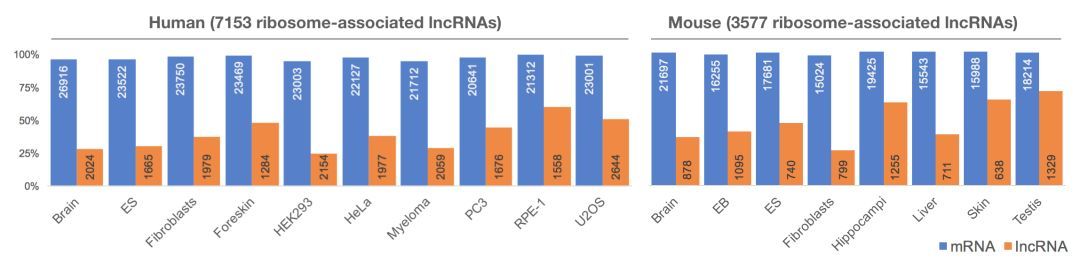
图 表达的mRNA(蓝色)和lncRNA(橙色)的百分比与多种组织或细胞类型的核糖体相关
5
中文标题:基于长链非编码RNA关联的功能富集分析
英文标题:Functional enrichment analysis based on long noncoding RNA associations
期刊:BMC Syst Biol
影响因子:2.050

使用RNA-seq数据进行差异基因表达分析是在某些环境条件下发现特定调节机制的常用方法。GO和KEGG途径富集分析都是研究参与共同生物反应或具有相关功能的基因组的主要方法。然而,基于差异表达基因的传统方法仅检测一些重要的GO term和途径,这通常不足以解释全部基因调控机制。
来自国立台湾海洋大学的研究人员对survivin(birc5)基因敲除实验和野生型对照斑马鱼胚胎的转录组进行了测序和组装,获得了差异表达(DE)基因列表,用于传统的功能富集分析。除了包含具有显著倍数变化水平的DE基因外,研究人员还考虑了与差异表达的长链非编码RNA(DE lncRNA)接近或重叠的其他相关基因,这些基因可直接或间接激活或抑制靶基因,并在调控网络中发挥重要作用。将原始DE基因列表和额外的DE lncRNA相关基因组合以进行全面的过表达分析。
在这项研究中,共有638个DE基因和616个DE lncRNA相关基因(lncGenes)同时用于寻找重要的GO term和KEGG途径。与仅使用差异表达基因列表的传统方法相比,所提出的使用DE lncRNA相关基因的方法鉴定了几种其他重要的GO term和KEGG途径。在GO富集分析中,获得了60%以上的GO term,并且检索了几个神经元发育功能term作为完整注释。研究人员还观察到其他重要途径,如FoxO和MAPK信号通路,其在以前的报告中已有报道,在由survivin基因调控的细胞凋亡和神经元发育功能中发挥重要作用。
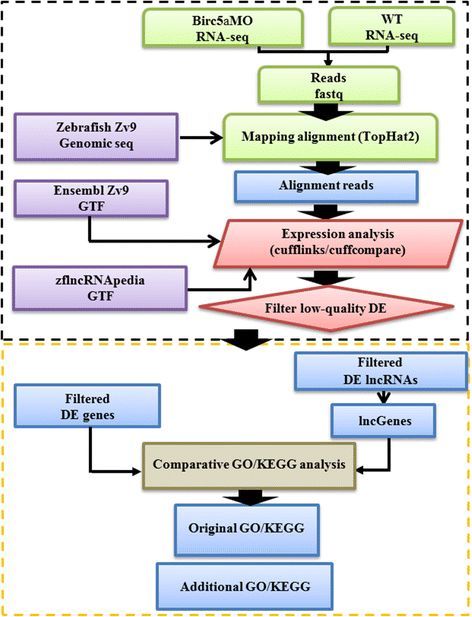
图 RNA-seq分析流程图
1. Talyan S, Andrade-Navarro MA, Muro EM. (2018) Identification of transcribed protein coding sequence remnants within lincRNAs. Nucleic Acids Res[Epub ahead of print].
2. Zhao Q, Sun Y, Wang D, Zhang H, Yu K, Zheng J, Zuo Z. (2018) LncPipe: A Nextflow-based pipeline for identification and analysis of long non-coding RNAs from RNA-Seq data. J Genet Genomics[Epub ahead of print].
3.Vito D, Smales CM. (2018) The Long Non-Coding RNA Tranome Landscape in CHO Cells Under Batch and Fed-Batch Conditions. Biotechnol J[Epub ahead of print].
4.Chao Zeng, Tsukasa Fukunaga and Michiaki Hamada, (2018) Identification and analysis of ribosome-associated lncRNAs using ribosome profiling data. BMC Genomics19:414.
5.Hung KS, Hsiao CC, Pai TW, Hu CH, Tzou WS, Wang WD, Chen YR. (2018) Functional enrichment analysis based on long noncoding RNA associations.BMC Syst Biol12(Suppl 4):45.
本周lncRNA相关报道就讲到这里了,下期见~
- 本文固定链接: https://www.maimengkong.com/moreshare/1111.html
- 转载请注明: : 萌小白 2022年7月9日 于 卖萌控的博客 发表
- 百度已收录
