之前使用clusterProfiler包进行GSEA富集,得到的标准的GSEA富集结果图中只展示基因集的ES排序,但在图中不能标注基因,一直令我比较头秃。
但最近看到一个R包GseaVis,刚好可以和clusterProfiler互补,使之能轻松在GSEA标准图上标注候选基因,同时也支持点阵图样式,非常简单!今天就来用用看!
#部分所需R包安装与载入:
devtools::install_github( "junjunlab/GseaVis")
library(stringr)
library(org.Hs.eg.db)
library(clusterProfiler)
library(GseaVis)
########
#1.先使用clusterProfiler包完成GSEA富集
#DESeq2差异分析结果表导入:
load( "DESeq2_homo.Rdata")
head(DESeq2) #使用所有基因进行GSEA,需要log2FC值以及symbol
#symbol转entrez ID:
symbol<- rownames(DESeq2)
entrez<- bitr(symbol,
fromType= "SYMBOL", #现有的ID类型
toType= "ENTREZID", #需转换的ID类型
OrgDb= "org.Hs.eg.db")
head(entrez)
#准备genelist文件(entrez+log2FC):
genelist<- DESeq2$log2FoldChange
names(genelist) <- rownames(DESeq2)
#过滤掉ID转换中缺失部分基因:
genelist<- genelist[names(genelist) %in% entrez[,1]]
head(genelist)
names(genelist) <- entrez[match(names(genelist),entrez[,1]),2]
head(genelist)
#按照log2FC从高到低排序:
genelist<- sort(genelist,decreasing = T)
head(genelist)
#GSEA_KEGG富集分析:
R.utils::setOption( "clusterProfiler.download.method", "auto") ##如果富集时报错就加上这句代码
KEGG_ges<- gseKEGG(
geneList= genelist,
organism= "hsa",
minGSSize= 10,
maxGSSize= 500,
pvalueCutoff= 0.05,
pAdjustMethod= "BH",
verbose= FALSE,
eps= 0
)
#将entrez重新转换为symbol:
KEGG_ges2<- setReadable(KEGG_ges,
OrgDb= org.Hs.eg.db,
keyType= "ENTREZID")
#转换前:
KEGG_ges@result$core_enrichment[1]
#转换后:
KEGG_ges2@result$core_enrichment[1]
save(KEGG_ges,KEGG_ges2,file = c('GSEA_KEGG.Rdata'))
rm(list=ls)
########
#2.使用GseaVis包完成GSEA结果可视化:
load('GSEA_KEGG.Rdata')
result<- KEGG_ges2@result
View(result) #查看GSEA结果表,选择想展示的基因集/通路绘图
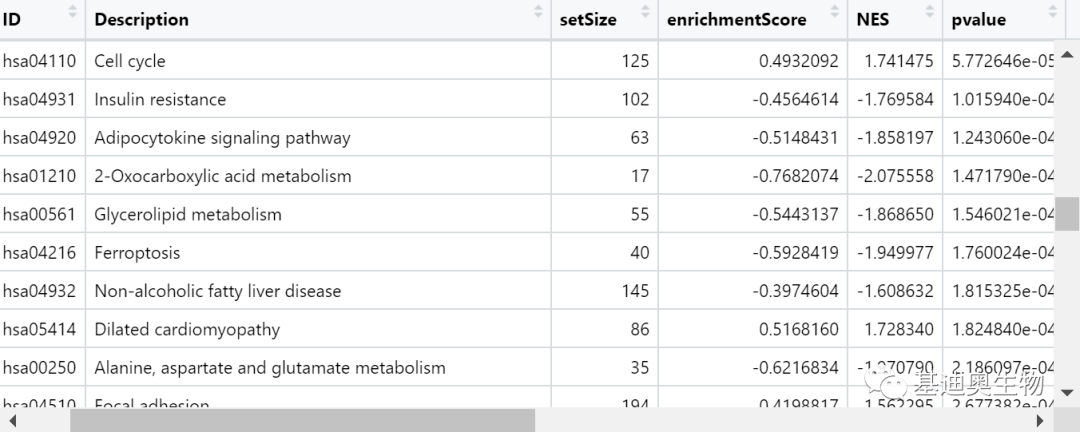
colnames(result)
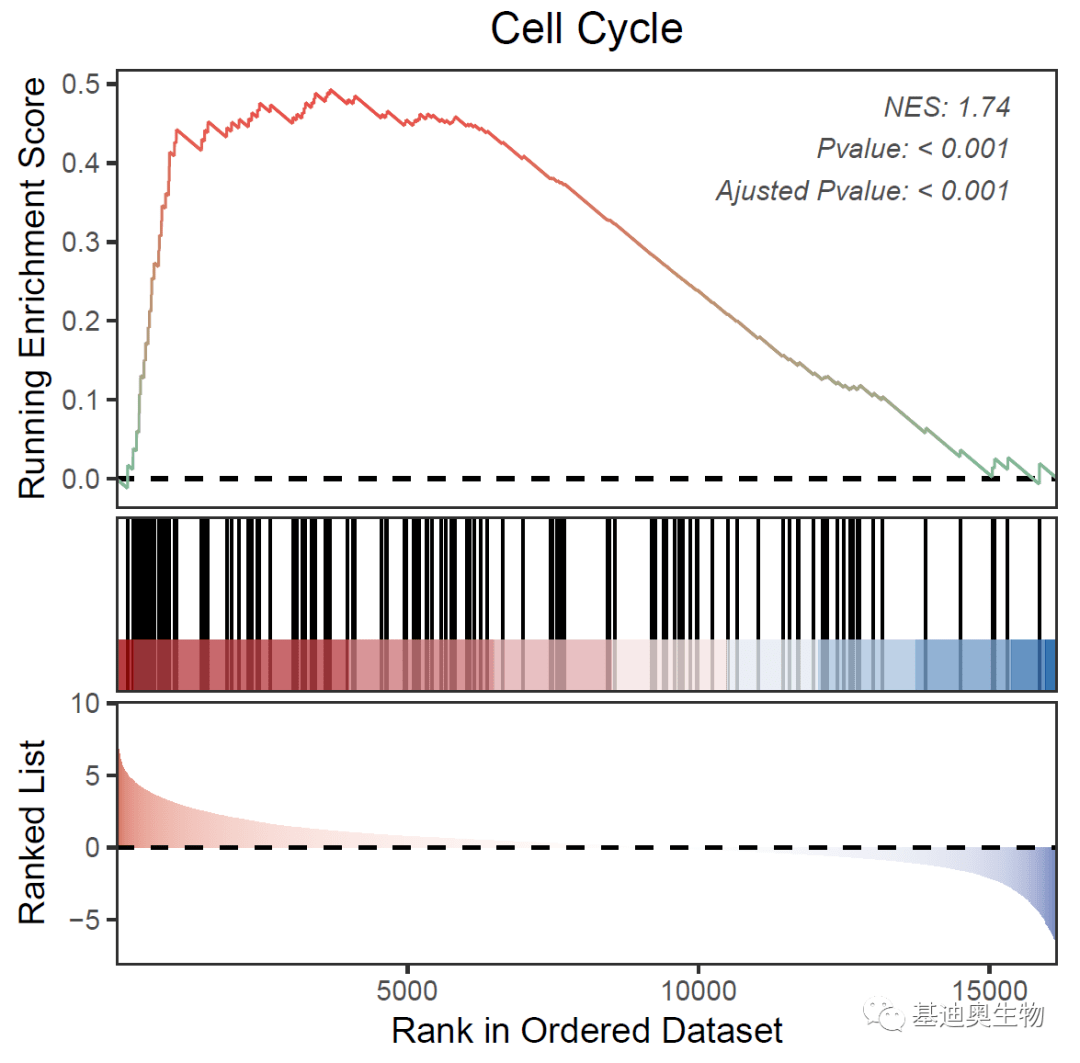
#2.1——标准GSEA富集分析结果图:
gseaNb(
object= KEGG_ges2,
geneSetID= KEGG_ges2@result$ID[38], #绘制结果表中第38个pathway(自行选择即可)
subPlot= 3, #常规为3图组合,如果不需要条码图或rank图可以设置为1 or 2
addPval= T, #图中是否显示P值和NES标签
pvalX= 0.95,
pvalY= 0.8 #调整标签X/Y坐标控制位置
)
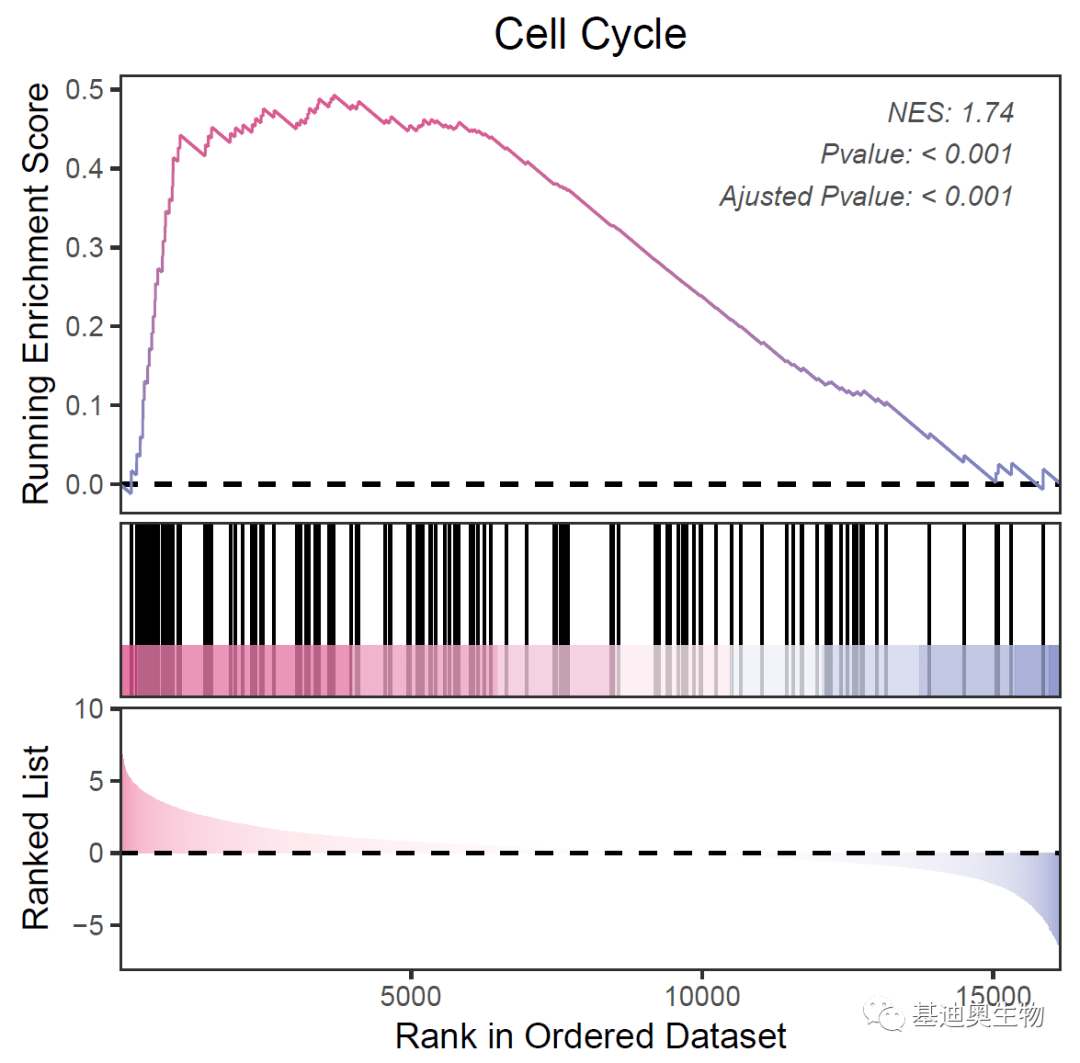
#更改配色:
gseaNb(
object= KEGG_ges2,
geneSetID= KEGG_ges2@result$ID[38],
subPlot= 3,
addPval= T,
pvalX= 0.95,
pvalY= 0.8,
curveCol= c('#7582c1', '#dd568d'), #ES折线图颜色更改
htCol= c( "#7582c1", "#dd568d"), #热图条颜色更改
rankCol= c( "#7582c1", "white", "#dd568d") #rank分布图颜色更改
)
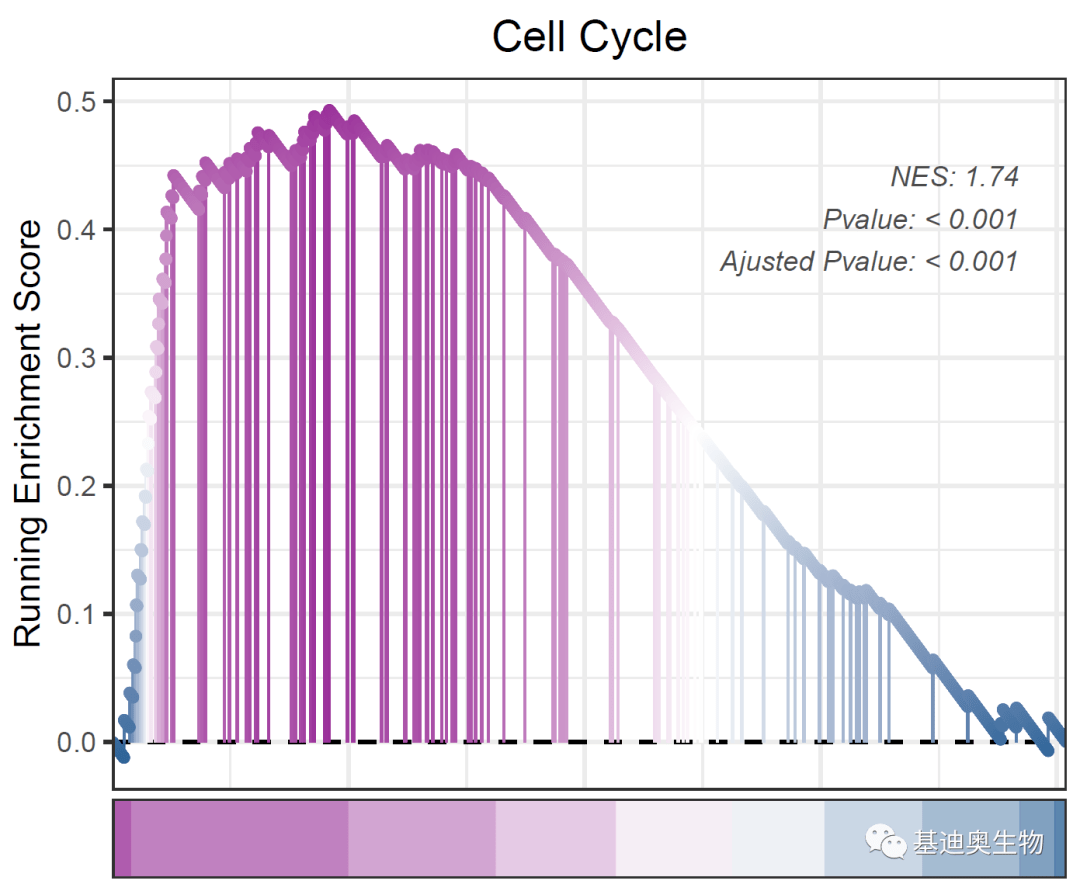
#2.2——GSEA富集分析点阵图:
gseaNb(
object= KEGG_ges2,
geneSetID= KEGG_ges2@result$ID[38],
addPval= T,
pvalX= 0.95,
pvalY= 0.8,
newGsea= T, #选择点阵图样式
)
#去掉散点和热图条:
gseaNb(
object= KEGG_ges2,
geneSetID= KEGG_ges2@result$ID[38],
addPval= T,
pvalX= 0.95,
pvalY= 0.8,
newGsea= T,
addPoint= F, #是否添加散点
rmHt= T #是否移除底部热图条
)
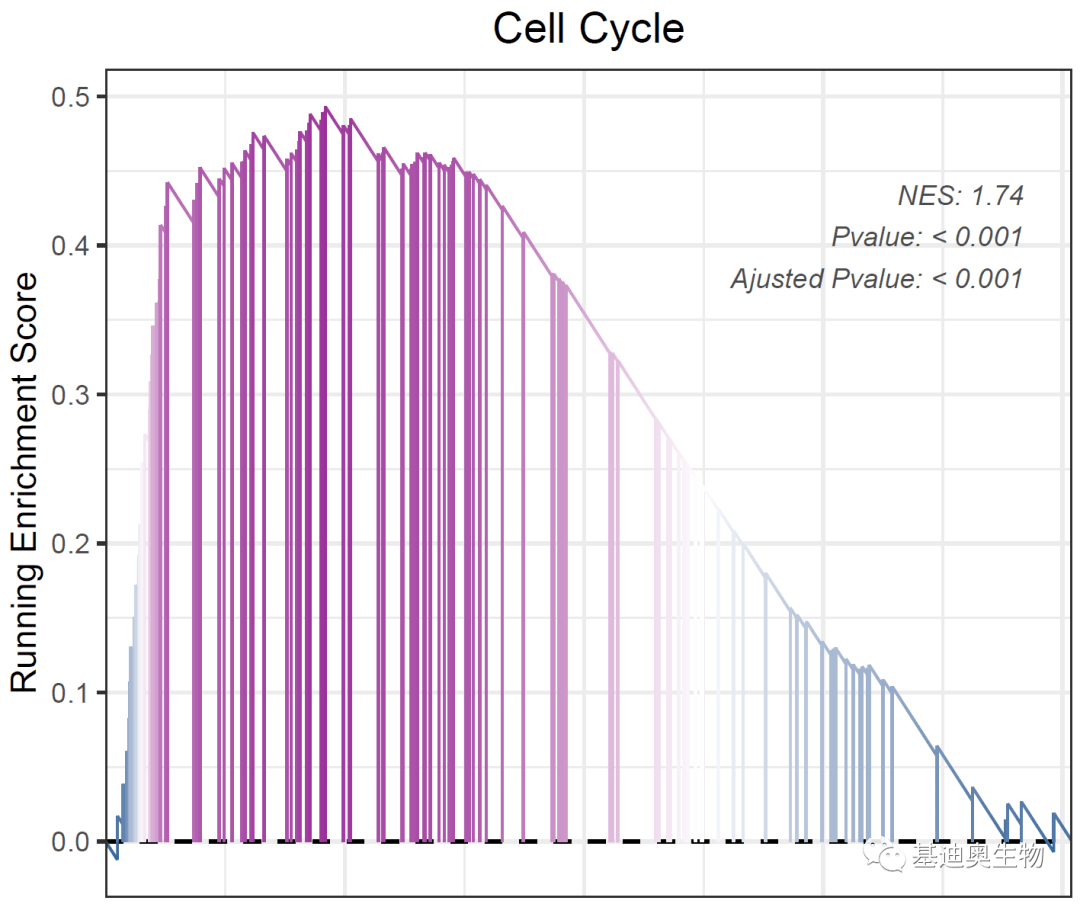
#更改配色:
gseaNb(
object= KEGG_ges2,
geneSetID= KEGG_ges2@result$ID[38],
addPval= T,
pvalX= 0.95,
pvalY= 0.8,
newGsea= T,
addPoint= F,
newCurveCol= c( "#001871", "#b9b4ad", "#f99f1c"), #ES点阵图颜色更改
newHtCol= c( "#001871", "white", "#f99f1c") #热图条颜色更改
)
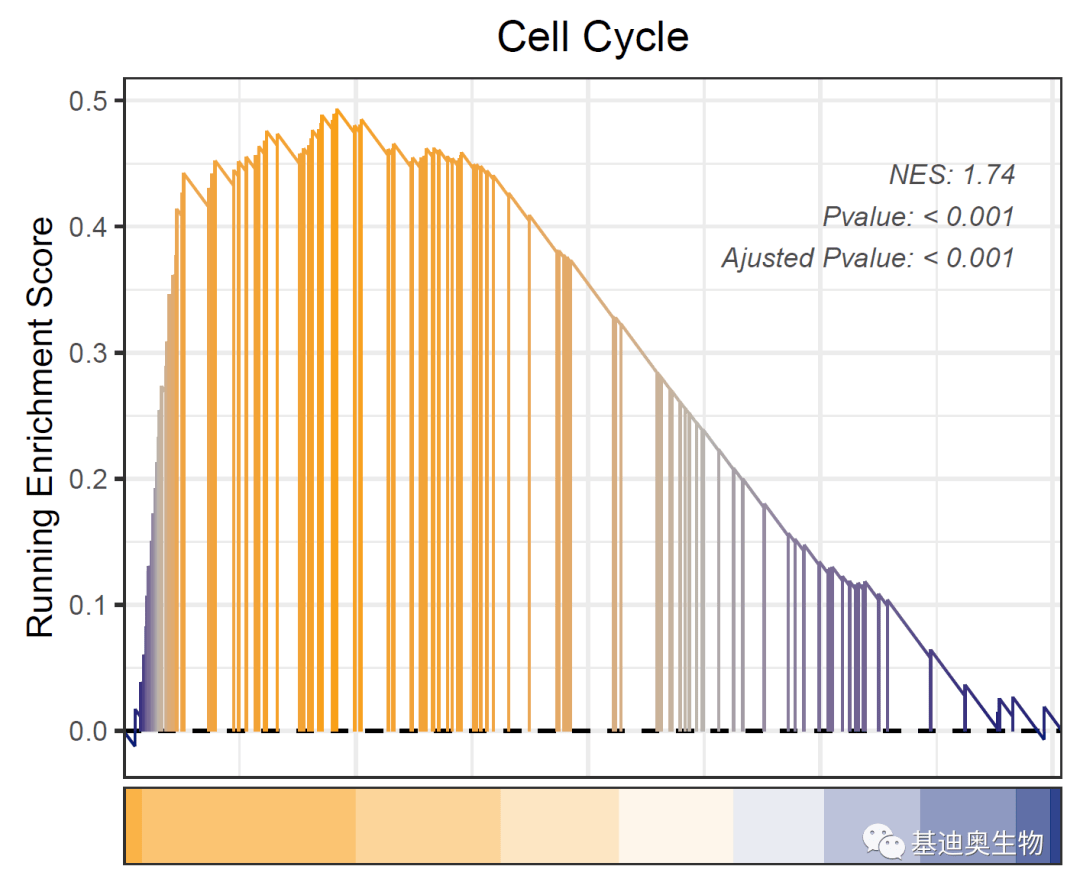
#标注Top rank基因:
gseaNb(
object= KEGG_ges2,
geneSetID= KEGG_ges2@result$ID[38],
addPval= T,
pvalX= 0.95,
pvalY= 0.8,
newGsea= T,
addPoint= F,
newCurveCol= c( "#001871", "#b9b4ad", "#f99f1c"),
newHtCol= c( "#001871", "white", "#f99f1c"),
addGene= T, #是否添加基因
markTopgene= T, #是否标注Top基因
topGeneN= 25, #标注前多少个gene
kegg= T, #是否将entrez转symbol
geneCol= ' #4d4d4d' #基因名标签颜色更改
)
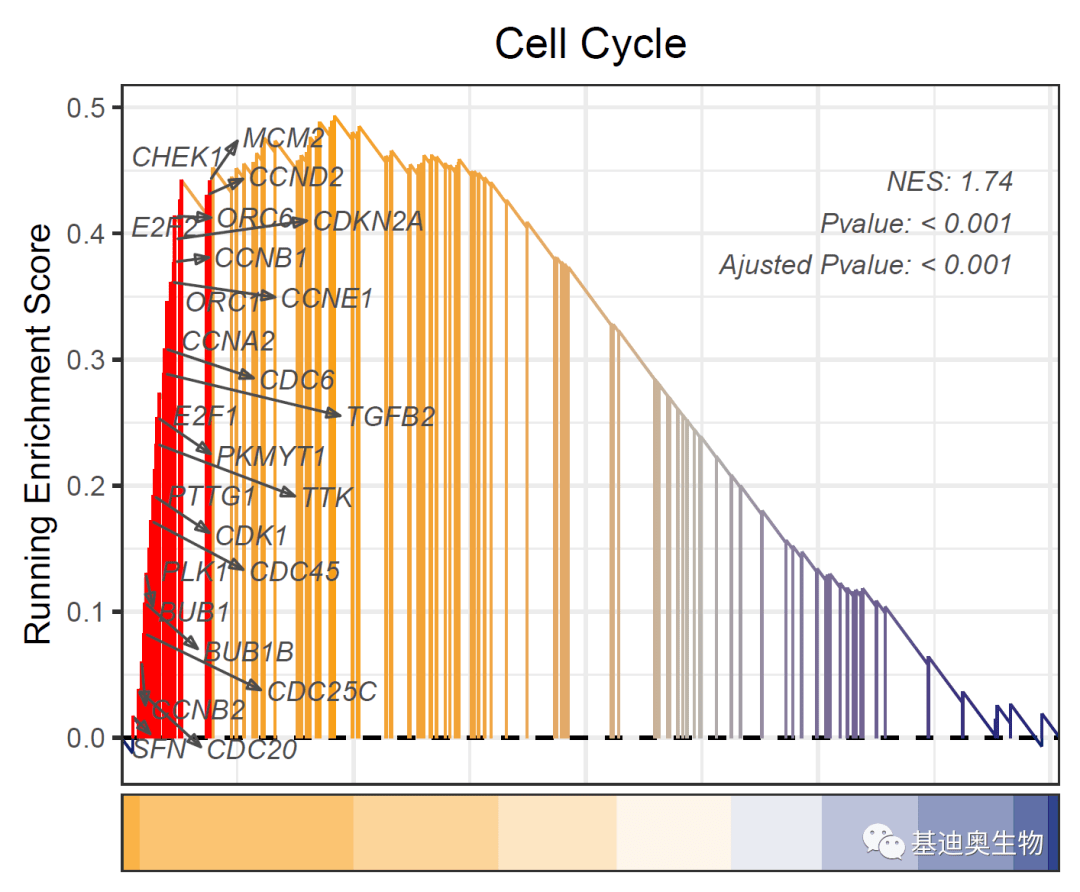
#显示该基因集全部核心基因:
##当然,也可以根据背景知识自行构建候选基因集进行标注:
core<- KEGG_ges2@result$core_enrichment[38]
length(core) #所有核心基因组成的1个向量,'/'分割;
core
#需要将core拆成单个基因组成的向量集:
core_genes<- str_split(core ,'/')[[1]]
core_genes
gseaNb(
object= KEGG_ges2,
geneSetID= KEGG_ges2@result$ID[38],
addPval= T,
pvalX= 0.95,
pvalY= 0.8,
newGsea= T,
addPoint= F,
newCurveCol= c( "#001871", "#b9b4ad", "#f99f1c"),
newHtCol= c( "#001871", "white", "#f99f1c"),
addGene= core_genes, #选择添加我们定义的基因集
kegg= T,
geneCol= '#4d4d4d'
)
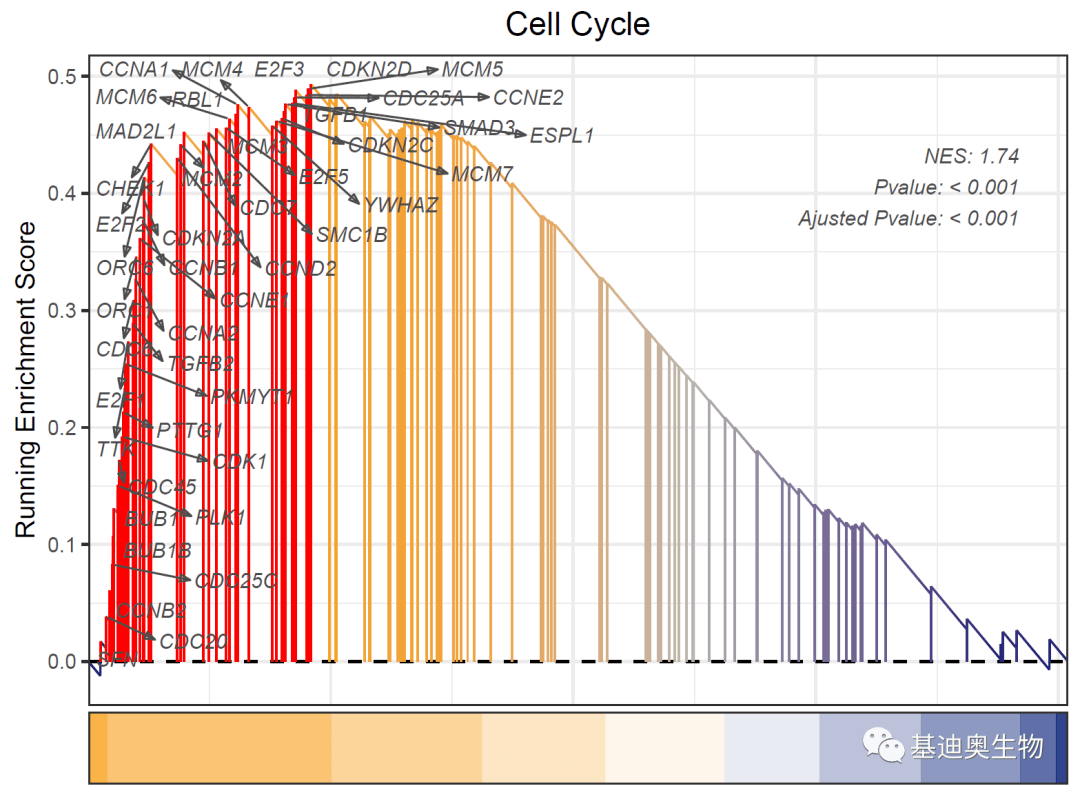
#不显示红线:
gseaNb(
object= KEGG_ges2,
geneSetID= KEGG_ges2@result$ID[38],
addPval= T,
pvalX= 0.95,
pvalY= 0.8,
newGsea= T,
addPoint= F,
newCurveCol= c( "#001871", "#b9b4ad", "#f99f1c"),
newHtCol= c( "#001871", "white", "#f99f1c"),
addGene= core_genes, #选择添加我们定义的基因集
kegg= T,
geneCol= '#4d4d4d',
rmSegment= T #是否移除红线
)
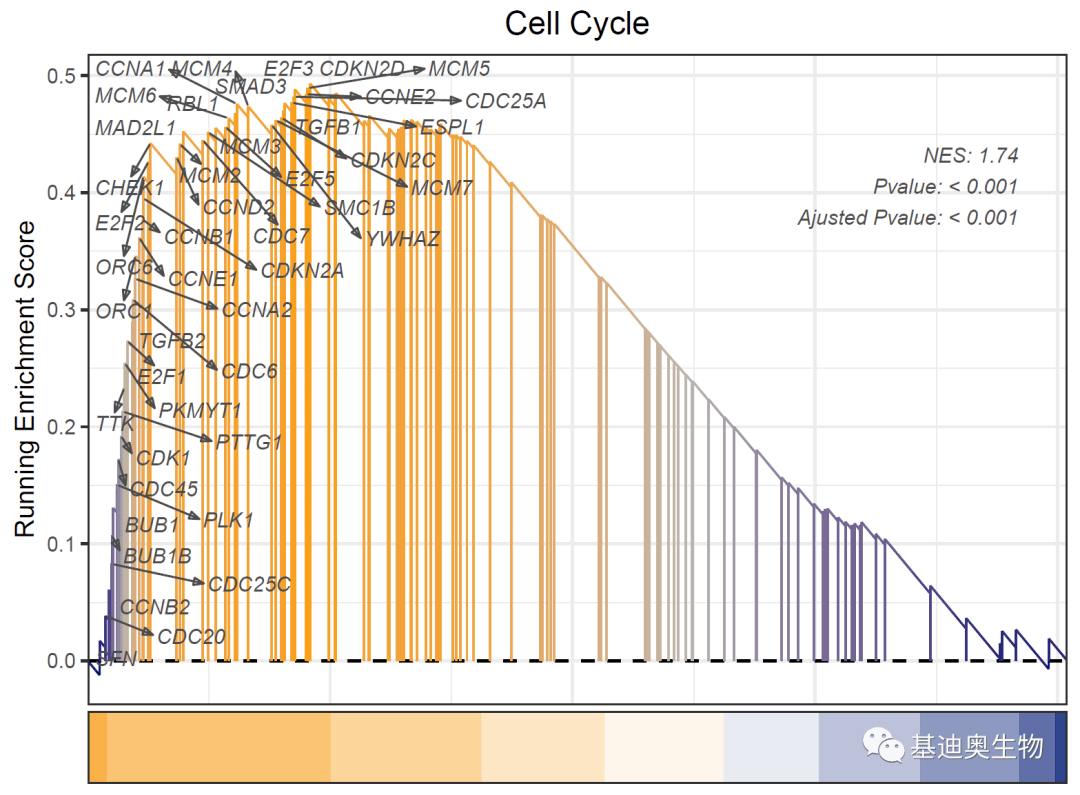
#2.3——在标准的GSEA富集结果图中标注关注的候选基因:
genes<- core_genes[sample(1:45,15)] #随机抽选15个基因仅供范例
gseaNb(
object= KEGG_ges2,
geneSetID= KEGG_ges2@result$ID[38],
subPlot= 3,
addPval= T,
pvalX= 0.95,
pvalY= 0.8,
curveCol= c('#7582c1', '#dd568d'),
htCol= c( "#7582c1", "#dd568d"),
rankCol= c( "#7582c1", "white", "#dd568d"),
addGene= genes,
kegg= T,
geneCol= '#4b4949',
rmSegment= T #移除红线
)
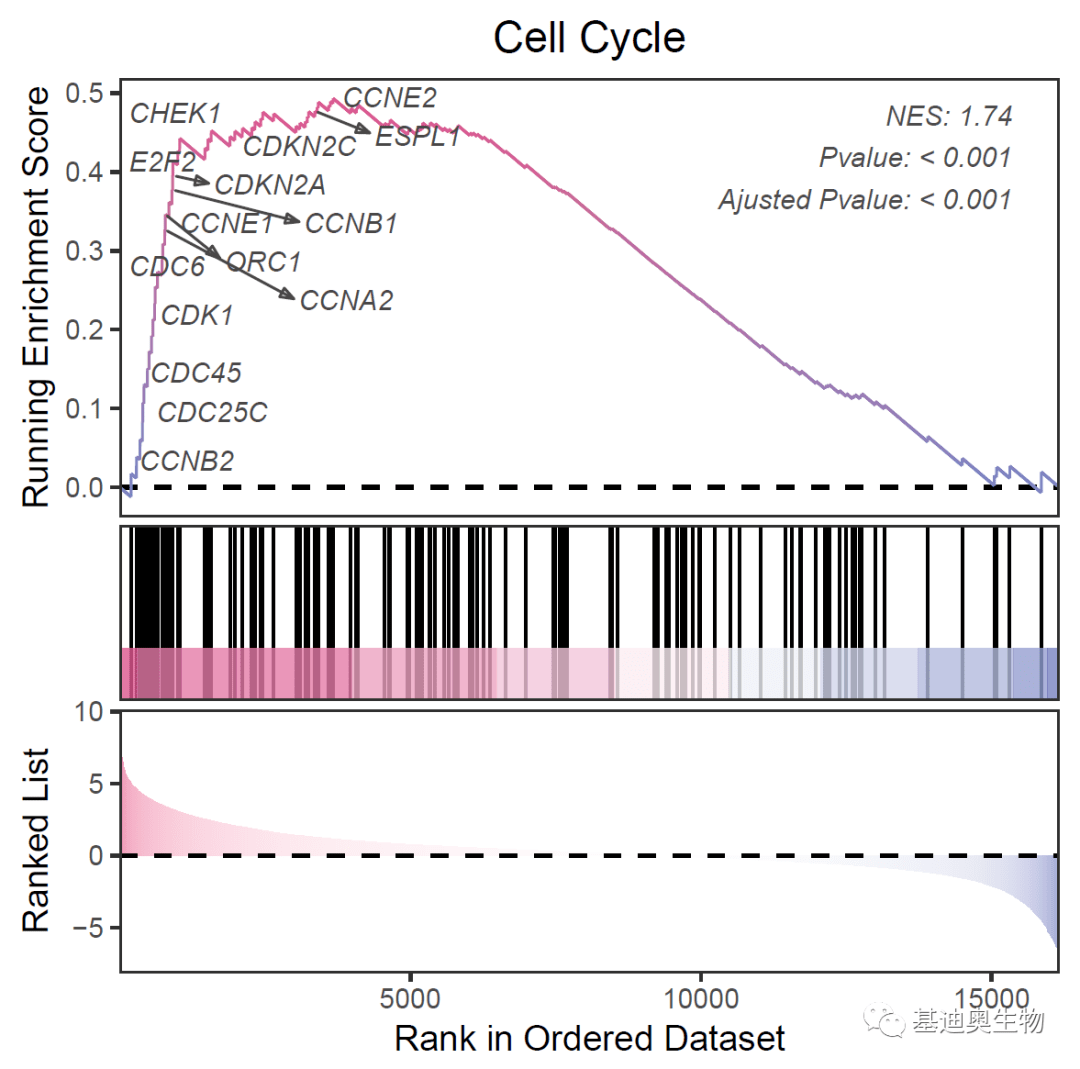
########
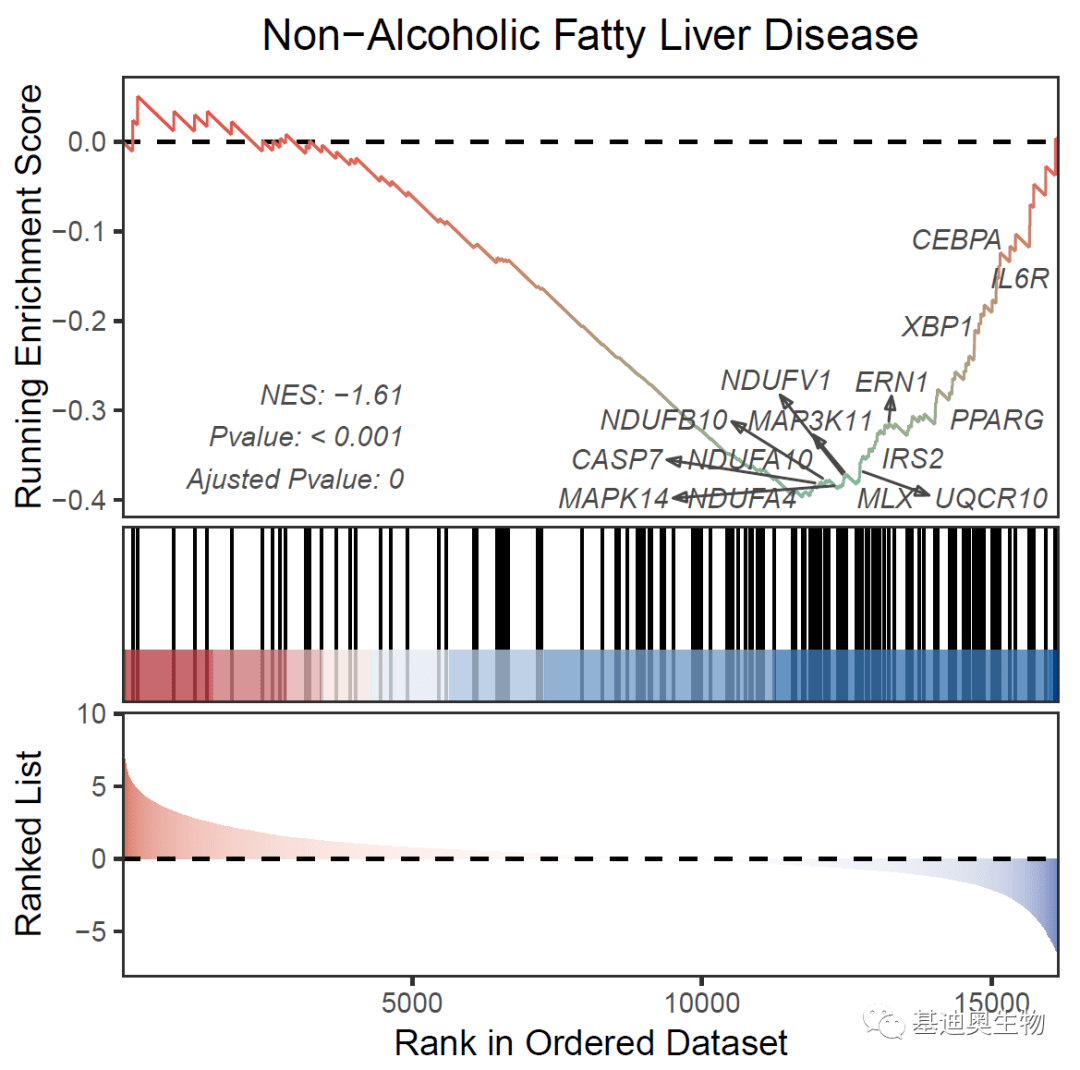
#最后再换一个核心基因下调的pathway简单瞅瞅:
#标注候选基因:
core2<- KEGG_ges2@result$core_enrichment[44]
core_genes2<- str_split(core2 ,'/')[[1]]
genes2<- core_genes2[sample(1:77,15)]
#标准:
gseaNb(
object= KEGG_ges2,
geneSetID= KEGG_ges2@result$ID[44],
subPlot= 3,
addPval= T,
pvalX= 0.3,
pvalY= 0.1,
addGene= genes2,
kegg= T,
geneCol= '#4b4949',
rmSegment= T
)
#点阵:
gseaNb(
object= KEGG_ges2,
geneSetID= KEGG_ges2@result$ID[44],
addPval= T,
pvalX= 0.2,
pvalY= 0,
newGsea= T,
addPoint= F,
addGene= genes2, #选择添加我们定义的基因集
kegg= T,
geneCol= '#4d4d4d'
)
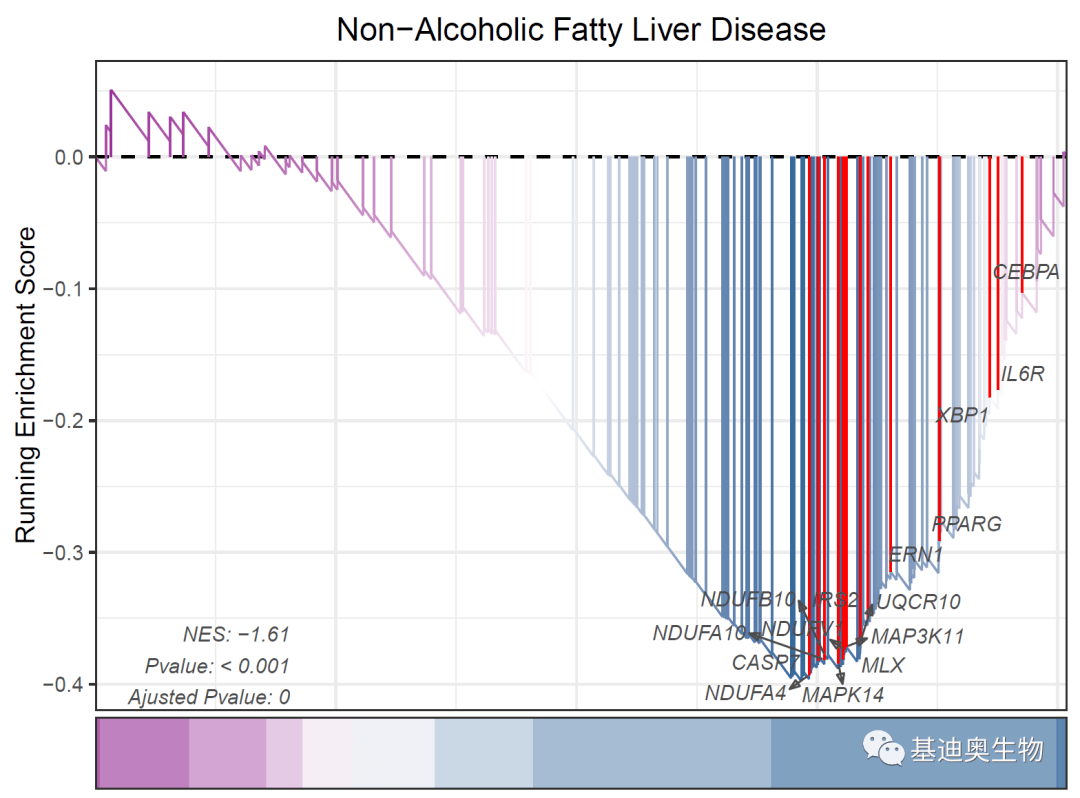
能够自定义标注基因,舒服了!
更多使用方法可查看该R包的说明文档,好啦,今天的分享就到这里!
参考资料
https://github.com/junjunlab/GseaVis/wiki
不会吧不会吧!!竟然还有人不知道我们有绘图公众号???
想获取更多高分期刊科研图表美化技巧么?想学会更多生信图表的实用绘制干货么?
请务必关注基迪奥旗下绘图公众号SCIPainter!什么叫大隐隐于市,什么叫酒香巷子深,什么叫少林寺的扫地僧!怀揣绘图绝技,深藏功名的就是它!SCIPainter!!!之后更多与科研图表绘制相关内容,我们会在这个账号中持续进行分享和发布。
能够坚持不跳出并看到文末的各位朋友,一定冰雪聪明机智过人精明能干七窍玲珑,慧眼识珠的你就差一个SCIPainter的关注!!!
*未经许可,不得以任何方式复制或抄袭本篇文章之部分或全部内容。版权所有,侵权必究。
基迪奥生物
- 本文固定链接: https://www.maimengkong.com/image/1247.html
- 转载请注明: : 萌小白 2022年10月5日 于 卖萌控的博客 发表
- 百度已收录
