学会这3张图,搞定数据质控So easy
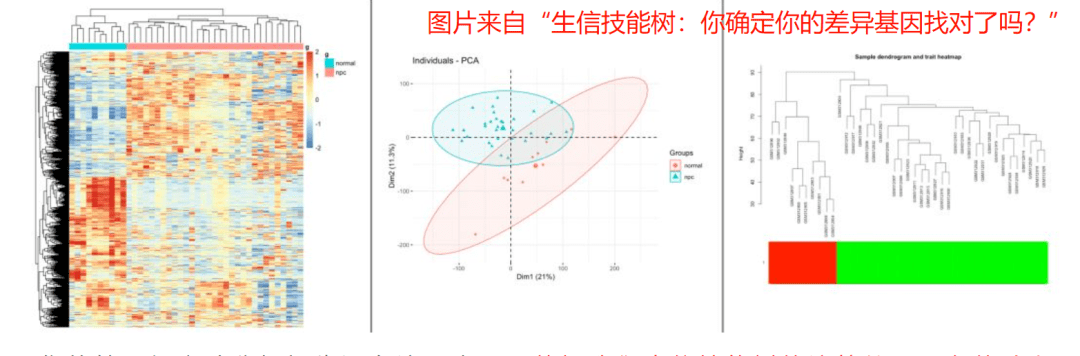
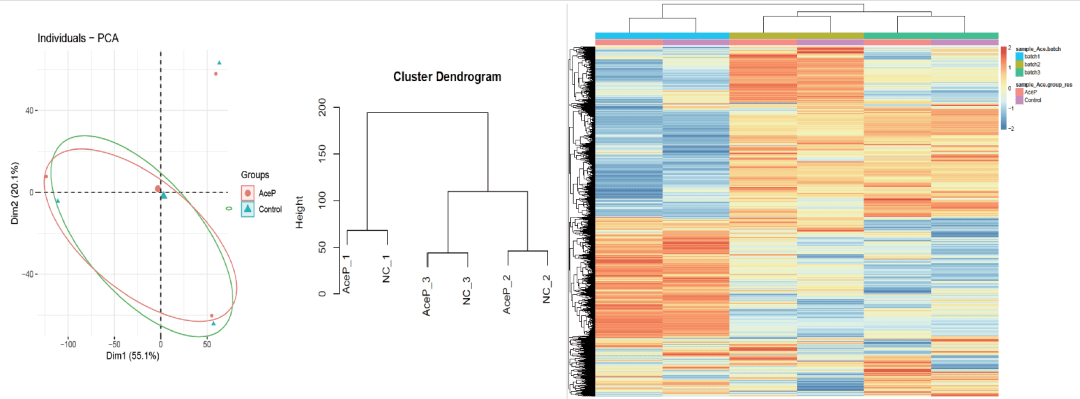
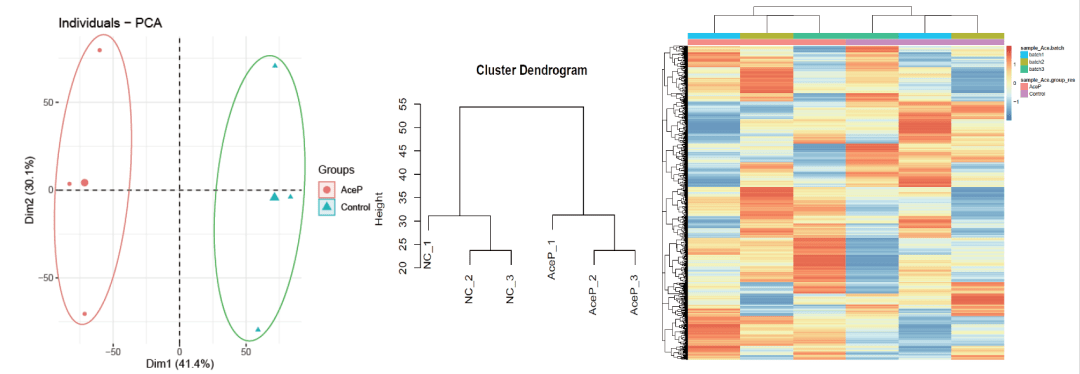
嗨,小伙伴们大家好~!上期我们讲到数据质控的重要性,即在表达差异分析之前需查验数据质量是否合格,如下面3张图所示,热图、PCA图和聚类树的结果均表示实验组和对照组分开的比较好,数据质量可。那么这期我们就接着这个话题,来看看如何绘制这3张图,搞定数据质控问题So easy!
▌加载示例数据
加载基因表达谱和样本信息文件,gene exp res是已完成基因注释和标准化处理的基因表达谱,sample_info是样本信息,其中包含1个干预组(AceP)和1个对照组(NC),每组包含3个样本(AceP1~3;NC1~3),每个样本是一个批次(AceP1/NC1;AceP1/NC1;AceP1/NC1),添加batch信息。
library(tidyverse) # 加载转录组数据 load( file= "All_data.Rdata") head(gene_exp_Ace) head(sample_Ace) # > head(gene_exp_Ace) # AceP_1 AceP_2 AceP_3 NC_1 NC_2 NC_3 # A1cf -2.3306531 0.1409379 -0.5429896 -1.9151062 0.5139516 -1.5410614 # A2m 1.7365109 -0.3501195 -0.5429896 1.4106091 -0.2948892 -0.5298033 # A3galt2 4.4778232 6.3508171 6.7475678 4.0037998 6.0675507 6.7629943 # A4galt 0.8584442 1.9777004 2.0243587 1.5603305 1.6145429 1.9387746 # AA926063 -0.3585691 -0.1598669 0.3085609 -0.2271234 -0.2948892 0.8555929 # Aaas 5.2688190 5.1461509 4.9110674 5.1650302 5.1788173 4.8904967 # > head(sample_Ace) # sample_id group_res # AceP_1 AceP_1 AceP # AceP_2 AceP_2 AceP # AceP_3 AceP_3 AceP # NC_1 NC_1 Control # NC_2 NC_2 Control # NC_3 NC_3 Control # 样本添加batch信息 sample_Ace <- sample_Ace %>% mutate(batch = paste0( "batch",c( 1, 2, 3, 1, 2, 3))) sample_Ace # sample_id group_res batch # AceP_1 AceP_1 AceP batch1 # AceP_2 AceP_2 AceP batch2 # AceP_3 AceP_3 AceP batch3 # NC_1 NC_1 Control batch1 # NC_2 NC_2 Control batch2▌数据质量评价
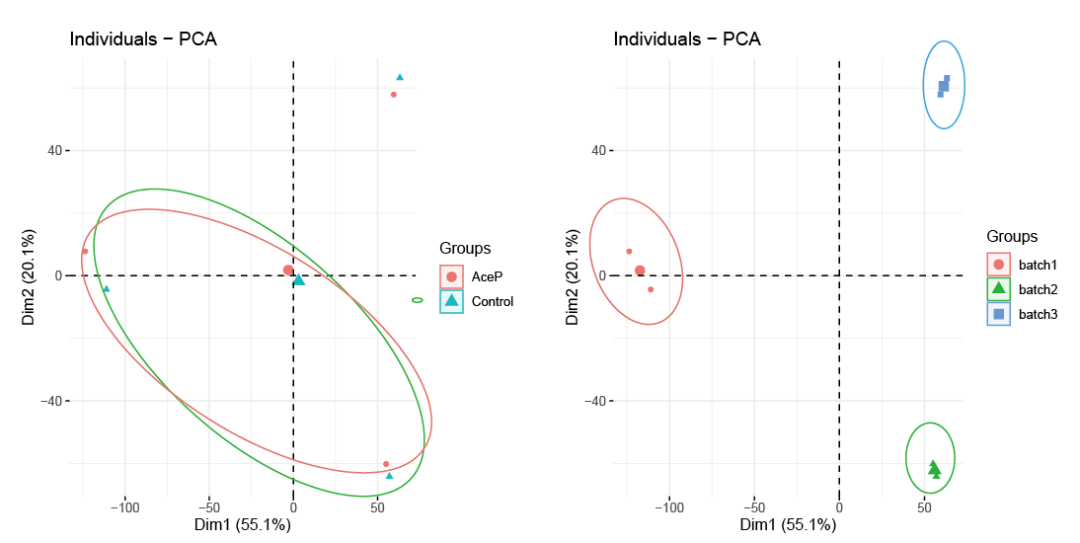
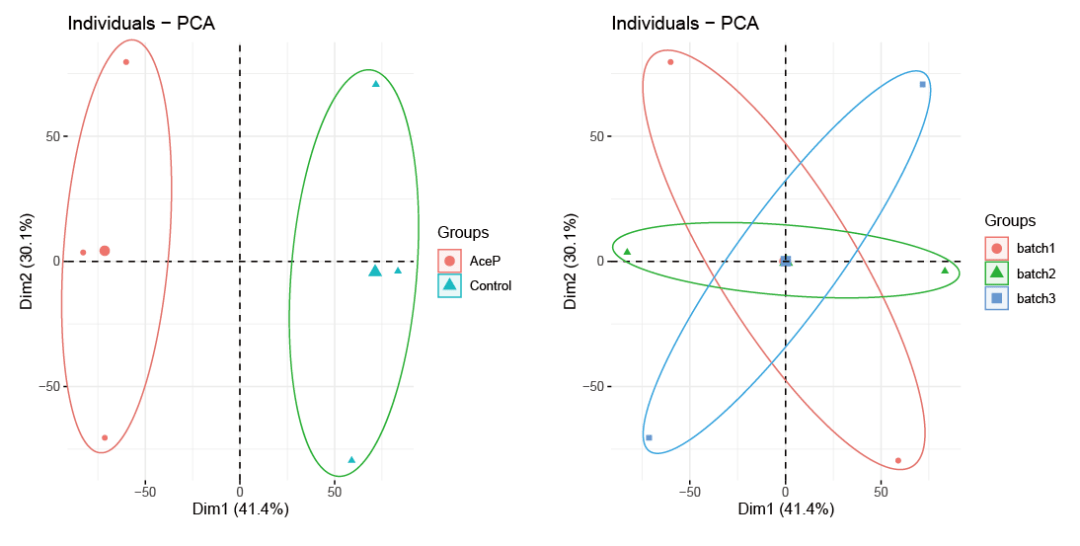
第一张图:PCA图,使用fviz pca ind函数。PCA直观可以看到干预组和对照组完全没有分开,样本是按照3个批次来聚类的,数据存在很明显的批次效应。
# 第一张图:PCA图 fviz_pca_ind函数 expr_pca<- t(gene_exp_Ace) pca_data<- cbind(expr_pca,sample_Ace) expr_pca<- prcomp(pca_data[,1:15061], scale= TRUE) expr_pca library(factoextra) # 按分组标注 fviz_pca_ind(expr_pca, label= "none", habillage= pca_data$group_res, # 按分组标注 addEllipses= TRUE, ellipse.level= 0.95) # 按批次标注 fviz_pca_ind(expr_pca, label= "none", habillage= pca_data$batch, # 按批次标注 addEllipses= TRUE, ellipse.level= 0.95)
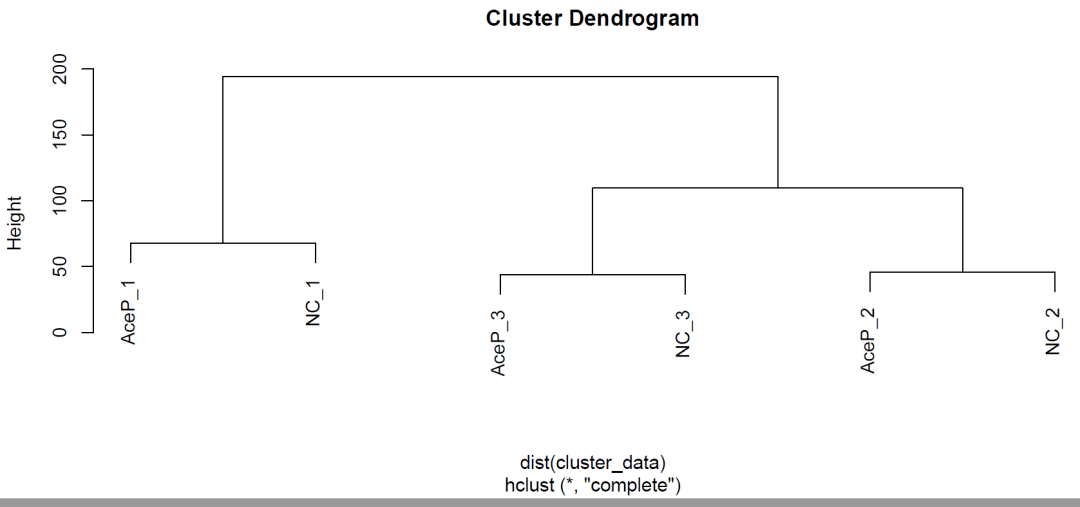
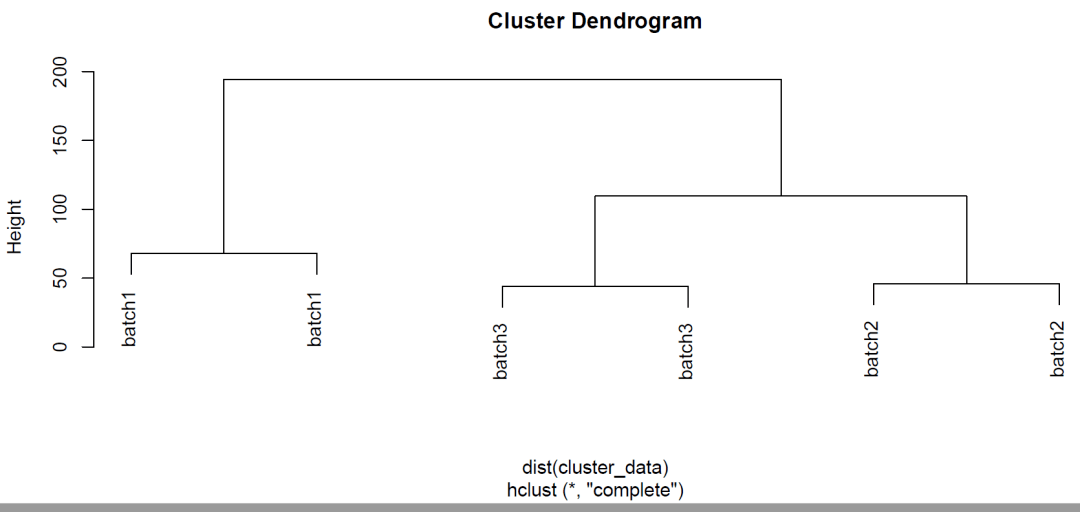
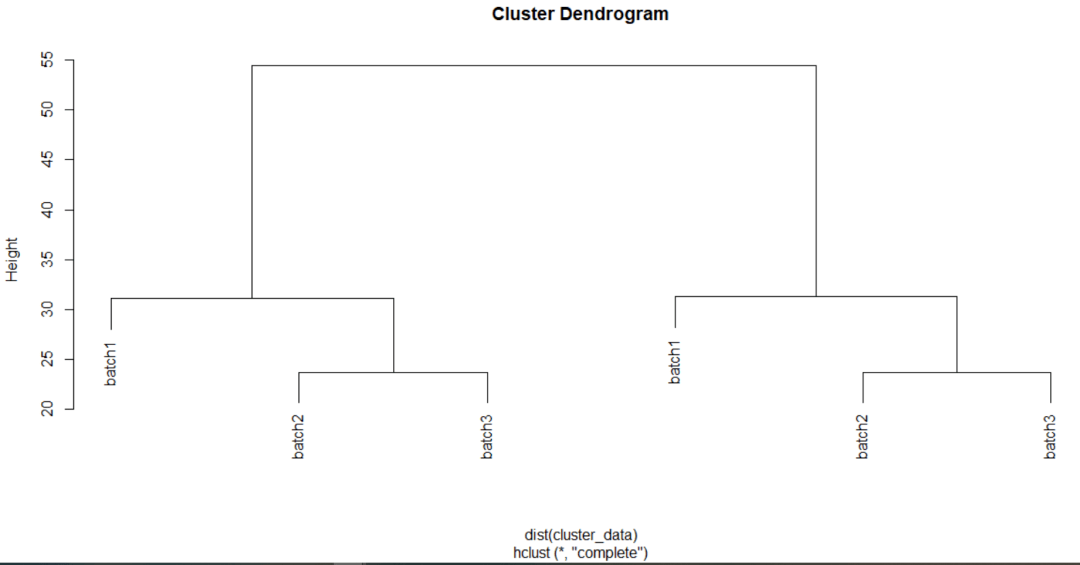
第二张图:聚类树图,使用hclust函数完成聚类分析,plot进行简单绘图。聚类树图同样直观可见干预组和对照组样本是按照3个批次来聚类的,并没有按组区分开。
# 第二张图:聚类树图 # 按样本聚类 cluster_data <- t(gene_exp_Ace) hc = hclust(dist(cluster_data)) plot(hc) # 按batch聚类 cluster_data <- t(gene_exp_Ace) rownames(cluster_data) = sample_Ace$batch hc = hclust(dist(cluster_data)) plot(hc)
第三张图:聚类热图,pheatmap函数实现。聚类热图也是同样的结果,直观可见干预组和对照组的样本是按照3个批次来聚类的。
# 第三张图:聚类热图 # 列注释信息 annotation_col<- data.frame(sample_Ace$group_res,sample_Ace$batch) rownames(annotation_col) <- colnames(gene_exp_Ace) # 绘制热图 library(pheatmap) pheatmap(gene_exp_Ace, scale= "row", show_colnames= F, show_rownames = F, border_color= NA, annotation_col= annotation_col, annotation_names_col= F)
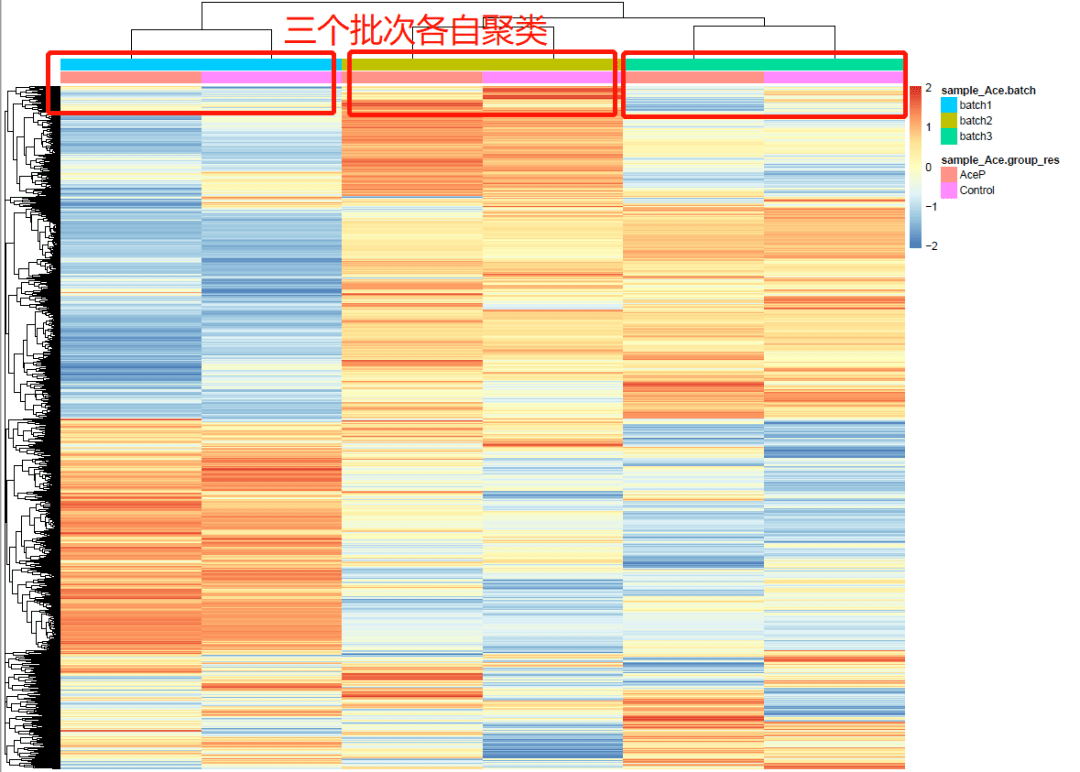
▌去除批次效应
既然数据存在很明显的批次效应,不放去以ComBat除批次效应试试看,随后再分别绘制PCA图、聚类树图和聚类热图,结果均显示样本在组间分开较好,而不再是按批次聚类。
# 去除批次效应 ComBat # ??ComBat library(sva) mod<- model.matrix(~sample_Ace$group_res) exprAce_batch<- ComBat(dat = gene_exp_Ace, batch= sample_Ace$batch, mod= mod) # Found3batches # Adjusting for1covariate(s) or covariate level(s) # Standardizing Data across genes # Fitting L/S model and finding priors # Finding parametric adjustments # Adjusting the Data # 第一张图:PCA图 fviz_pca_ind函数 expr_pca<- t(exprAce_batch) pca_data<- cbind(expr_pca,sample_Ace) expr_pca<- prcomp(pca_data[,1:15061], scale= TRUE) expr_pca library(factoextra) # 按分组标注 fviz_pca_ind(expr_pca, label= "none", habillage= pca_data$group_res, # 按分组标注 addEllipses= TRUE, ellipse.level= 0.95) # 按批次标注 fviz_pca_ind(expr_pca, label= "none", habillage= pca_data$batch, # 按批次标注 addEllipses= TRUE, ellipse.level= 0.95) # 第二张图:聚类树图 # 按样本聚类 cluster_data<- t(exprAce_batch) hc= hclust(dist(cluster_data)) plot(hc) # 按batch聚类 cluster_data<- t(exprAce_batch) rownames(cluster_data)= sample_Ace$batch hc= hclust(dist(cluster_data)) plot(hc) # 第三张图:聚类热图 # 列注释信息 annotation_col<- data.frame(sample_Ace$group_res,sample_Ace$batch) rownames(annotation_col) <- colnames(exprAce_batch) # 绘制热图 library(pheatmap) pheatmap(exprAce_batch, scale= "row", show_colnames= F, show_rownames = F, border_color= NA, annotation_col= annotation_col, annotation_names_col= F)

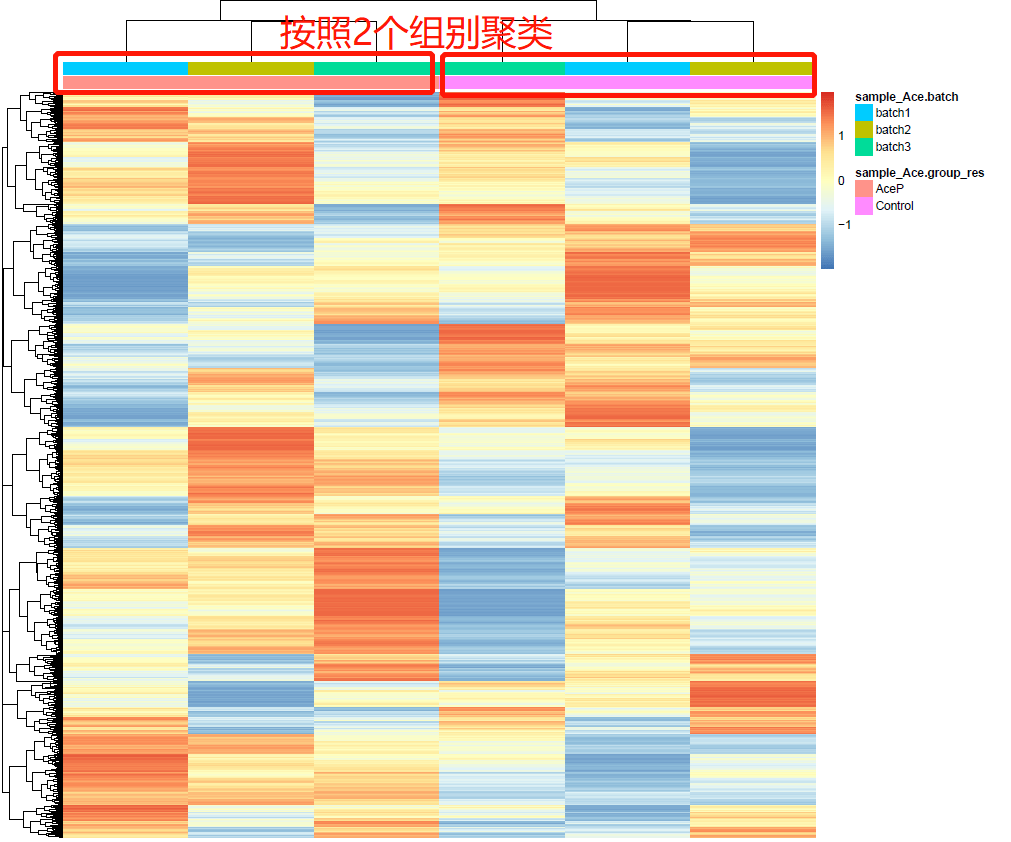
总结
汇总一下去除批次效应前后的3张图,大家细品~!
- 本文固定链接: https://www.maimengkong.com/image/1034.html
- 转载请注明: : 萌小白 2022年6月24日 于 卖萌控的博客 发表
- 百度已收录
