文章信息
发表期刊:Plant Biotechnology Journal
影响因子:6.84
发表时间:2019年9月24日
合作单位:中国农业科学院郑州果树研究所
1.
背景介绍
石榴是中亚地区重要的果树品种,以其果实的功能和营养特性备受欢迎。同时,石榴的果汁、种子及提取物可以用治疗心血管疾病、糖尿病、前列腺癌等病症。石榴有硬籽和软籽品种之分,两种石榴各有其特点,硬籽石榴耐低温,而软籽品种更易吞咽。揭示硬籽和软籽品种之间性状的变异对于石榴的分子标记辅助育种意义重大。
不同石榴品种硬籽和软籽表型的差异是多方面影响的结果,如复杂的地理环境、自然选择、杂交、进化压力等,可以通过比较基因组和分析群体多样性来解析造成表型差异的原因。泰山红和大笨籽两个硬籽石榴的代表性品种,2017 年Qin等通过二代短reads组装的大笨籽基因组发表,同年,泰山红的基因组也被Yuan等学者破译并发表。但是这两版基因组组装片段较多,而且较多contigs和scaffolds的排序和定向存在错误,这将阻碍基因图位克隆以及重要性状的遗传调控区域挖掘研究工作的进展。
前人关于石榴硬籽和软籽研究的重点主要集中在种子硬度上,比如分析木质素和纤维素合成基因(CAD, CelSy, SuSy, CCoA-OMT, MYB等)在硬籽和软籽石榴不同发育阶段的表达情况。再比如通过蛋白定量分析以及microRNA测序发现可以改变细胞壁结构的基因对于硬籽和软籽形成做出贡献。基因QTL定位研究显示,前人定位到4个与籽粒硬度相关的QTL,解释的表型贡献率介于15%-30%之间。然而,如果想深度揭示石榴籽粒硬度的遗传机制,仍需大量的工作。
本研究中,作者借助单分子实时测序技术以及Hi-C技术组装了软籽石榴Tunisia的高质量基因组,并对26个不同籽粒硬度的石榴品种进行了重测序分析。通过比较基因组和群体遗传学分析,作者进一步揭示了石榴的遗传多样性和群体结构,尤其是硬籽和软籽石榴之间的遗传多样性。
2.
选材
11年生的软籽石榴Tunisia用于三代测序,26个不同籽粒硬度的石榴品种进行Illumina HiSeq2500测序。
流式细胞分析估计基因组大小
提取Tunisia的DNA,使用MoFlo XDP Cell Sorter进行定量分析,以玉米做内参。
文库构建及测序
CTAB法提取全基因组DNA,按protocol构建Illumina文库。使用PacBio DNA试剂盒构建三代测序文库,共测4个SMRT cell,产生20Gb的数据。
基因组组装及染色体水平挂载
PacBio数据经过滤后,使用Canu进行从头组装。使用Pilon用二代数据进行基因组纠错。
取Tunisia的根、茎、叶、花、果皮和种子样品DNA进行Hi-C文库构建,使用LACHESIS进行contig的挂载。
取Tunisia的根、茎、叶、花、果皮和种子样品RNA,构建1–2 kb, 2–3 kb和> 3 kb的文库,进行全长转录组的的测序。
利用163个F1子代(Tunisia与硬籽品种Sanbai杂交得到),结合SLAF-seq的方法,构建了石榴的高密度遗传图谱,利用该遗传图谱和ALLMAPS软件进行contig的挂载。
重复序列预测
重复序列预测使用从头预测和基于同源检索的方法,从头预测使用MITE-Hunter,LTR-FINDER , RepeatScout和PILER-DF等软件,PASTEClassifier和RepeatMasker被用来进行重复序列的检测和分类。
基因预测及功能注释
基因预测使用从头预测、基于同源序列预测以及基于转录组预测的方法。使用TransDecoder进行开放阅读框预测,利用拟南芥、苹果、葡萄、巨桉进行同源预测,PASA用来进行基于cDNA的基因预测,使用EVM将三种方式的预测结果进行整合。通过与NCBI的非冗余蛋白库以及TrEMBL数据库进行比对确定基因功能,通过INTERPRO 、GO、KEGG、KOG等数据进行基因模式分析。
利用miRBase, Rfam和tRNAscan-SE数据库进行非编码RNA(microRNAs, rRNAs和tRNAs)预测,使用GenBlastA检测同源基因组序列,使用GeneWise进行假基因分析。
比较基因组分析
使用MUMmer软件分别对Tunisia和泰山红、大笨籽之间进行共线性和PAV检测。大笨籽二代数据比对Tunisia基因组使用BWA。
群体测序及结构分析
群体测序使用Xten平台,reads比对使用BWA软件,SNP calling使用GATK。MEGA 5用于无根NJ树的构建,Structure用于群体结构分析,EIGENSOFT用来进行PCA分析,PLINK 2用于LD分析。
遗传多样指数分析
Fst、群体核酸多态性π、Tajima’D等参数分析使用PopGenome进行分析。
3.
主要结果
(1)基因组测序及组装
使用Pacbio sequel平台共得到20.94Gb的数据,共组装得到石榴320.31Mb基因组(表1),包含661个contigs、473个scaffolds,contig N50和scaffolds N50分别为14.77Mb和55.56Mb,显著高于大笨籽(contig N50=66.97Kb)和泰山红(contig N50=97Kb)基因组组装指标。基因组组装质量评估使用三种策略,第一是使用二代数据进行回比,比对效率为96.76%,第二,BUSCO评估结果为93.33%,第三使用CEGMA评估,458个保守基因中组装得到其中454个,由此可见,基因组完整度较高。
Hi-C测序共得到17.62Gb(55x覆盖度)的数据,其中63.55%唯一比对的数据用于后续挂载,共将97.76%的序列挂载到染色体水平,从而得到石榴的8条染色体。利用161个F1群体构建得到含1725个SLAF标记的遗传图谱,平均图距仅为0.70 cM,借助该高密度图谱再次进行染色体水平挂载。
表1 组装结果
|
Assembly feature |
Statistic |
|
Estimate of genome size by flow cytometry |
313.18 Mb |
|
Total PacBio reads |
20.94 Gb |
|
Assembly length |
320.31 Mb |
|
Chromosome number(2n) |
2x8 |
|
Assembly % of chromosome |
97.76 |
|
Number of contigs |
661 |
|
Contig N50 |
4.49 Mb |
|
Longest contig |
14.77 Mb |
|
Number of scaffolds |
473 |
|
Scaffold N50 |
39.96 Mb |
|
Longest scaffold |
55.56 Mb |
(2)基因组注释
利用三种方式进行Tunisia基因预测,共得到33594个编码蛋白基因,其中28080个基因(表2)在多个数据库中进行功能注释。同时,检测得到52个miRNA、1468个rRNA、440个人tRNA和1388个假基因。
分析显示,Tunisia基因组中重复序列比例为50.93%,其中反转录转座子占比47.23%,Gypsy型占比17.33%,Copia型占7.2%。相较于泰山红和大笨籽,Tunisia基因组中的重复序列含量较高。
表2 基因组注释结果
|
Assembly feature |
Statistic |
|
Repeat region % of assembly |
50.93 |
|
Predicted gene models |
33,594 |
|
Assembly genes of chromosome |
32,538 |
|
Average gene length |
2,229 bp |
|
Average exon length |
263 bp |
|
Average CDS length |
1,048 bp |
|
GC content % |
40.38 |
(3)全基因组比较分析
通过Tunisia与泰山红基因组比较,发现83.74%的Tunisia基因组序列可以与泰山红97.90%的序列存在共线性,同样也发现Tunisia 85.63%的序列与83.58%的大笨籽序列存在共线性。另外,在Tunisia_大笨籽基因组间检测到7242个结构变异,在Tunisia_泰山红之间检测到1858个,随机挑选部分结构变异断点进行三代测序验证,验证率达80.56%。同时,发现Tunisia相较于泰山红基因组,存在46.01Mb的缺失,而与大笨籽相比,缺失量为41.13Mb。SNP和InDel分析发现,Tunisia与大笨籽之间存在496045个SNP和263228个InDel,Tunisia与泰山红之间存在326147个SNP和218687个InDel(图1)。硬籽品种Sanbai重测序分析与Tunisia基因组比对得到250,144个SNP和90,406217个InDel(图2)。
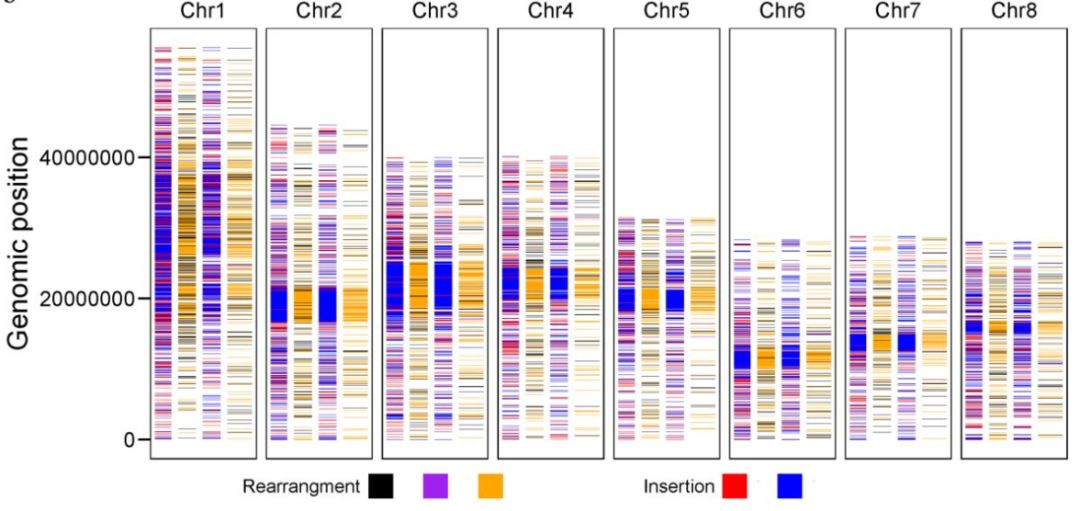
图1 Tunisia与泰山红、Tunisia与大笨籽之间全基因组变异比较
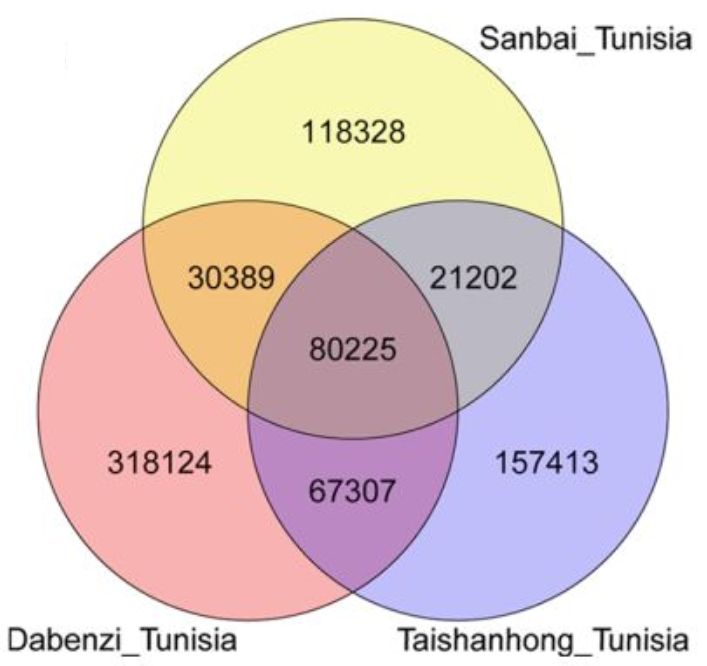
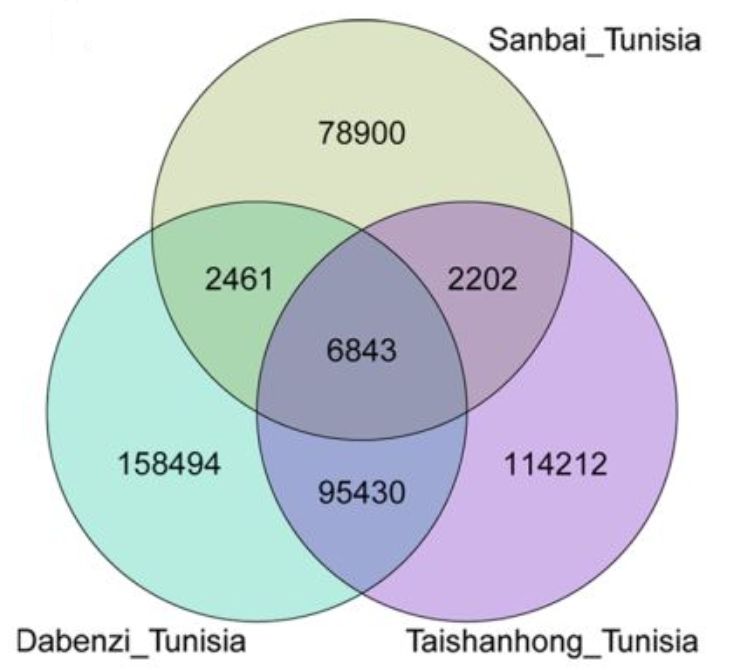
图2 SNP及InDel在Tunisia、泰山红、大笨籽之间的Veen图
(4)含SNP、IdeDel的基因
作者分别将Tunisia与三白、大笨籽和泰山红进行比较,得到共有的SNP、InDel。注释分析显示,这些变异共影响13034个基因,转录组分析发现,其中3492个基因在Sanbai和Tunisia的种子中存在差异表达,这些基因参与多个生物过程,如细胞分裂、信号转导等。另外,发现多个转录因子家族(如MYB、WRKY、AP2-like等)基因上存在SNP、InDel变异(图3),研究发现这些转录因子参与了石榴和山楂籽粒硬度的调控。
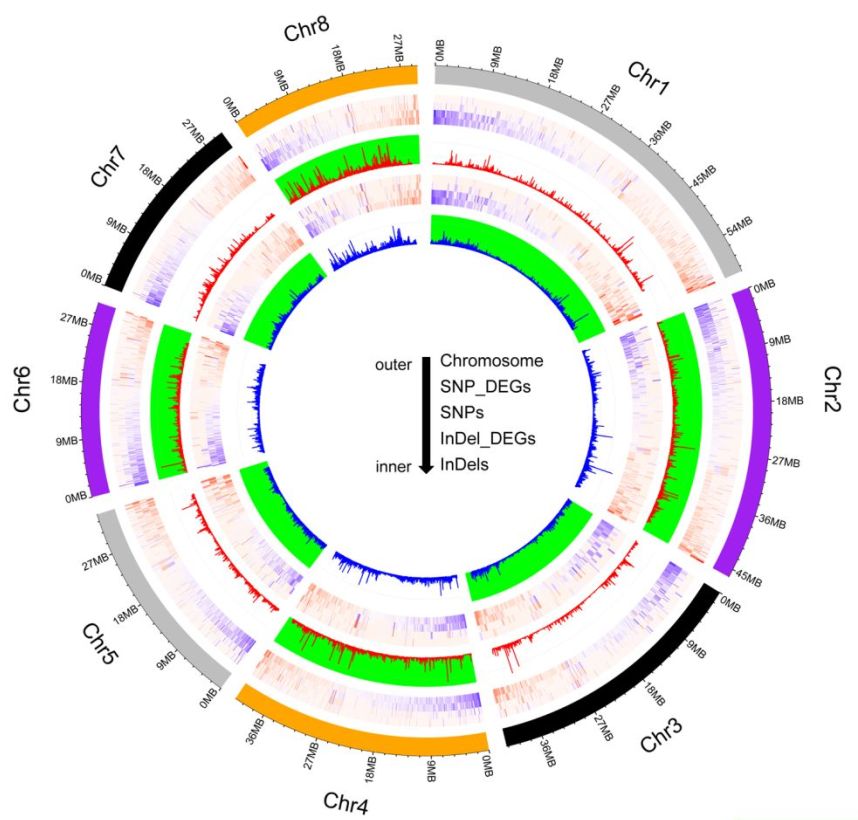
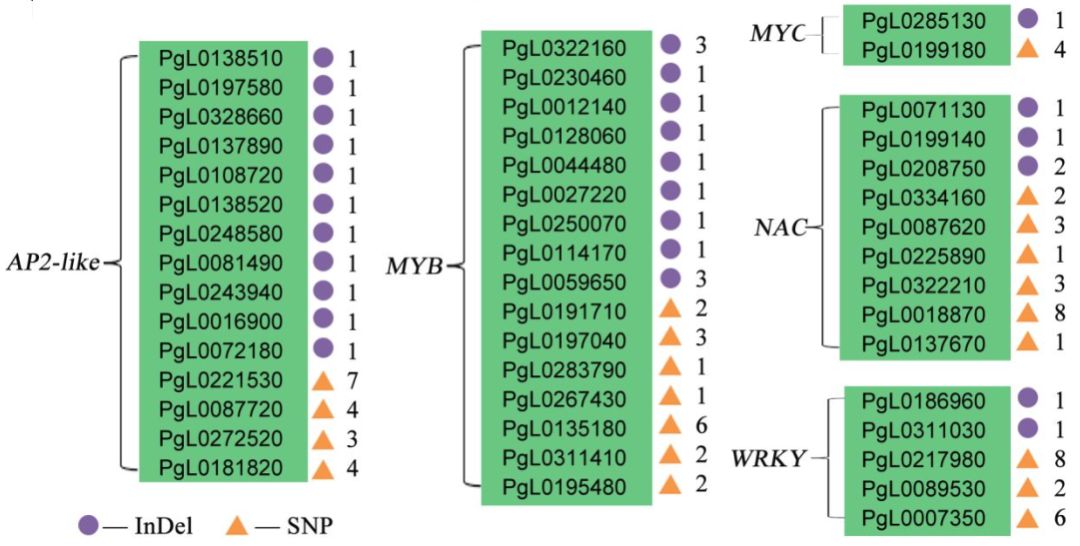
图3 SNP及InDel在Tunisia、泰山红、大笨籽之间分布及SNP、InDel在转录因子家族中的的分布
(5)遗传多样性及连锁不平衡分析
对26个不同品种的石榴资源进行重测序,平均每个样品测序深度17x,基因组覆盖度为97.91%。经过过滤,选取457525个SNP用于进化分析,进化树分析显示,硬籽品种独立分成一枝,而软籽、半软籽品种分到了一枝。群体结构分析发现,26个品种可以分为3个亚群,硬籽、软籽和半软籽品种分别划分到不同的亚群(图4)。PCA分析结果与群体结构分析结果一致。
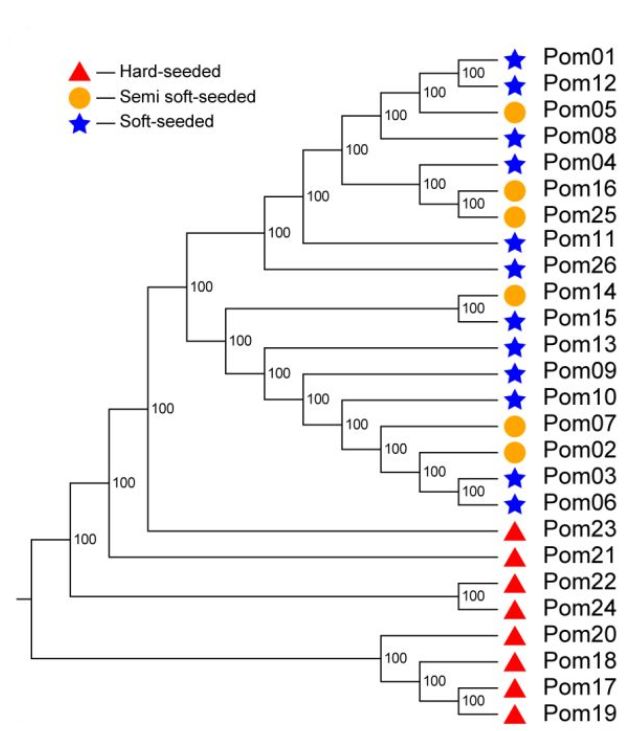
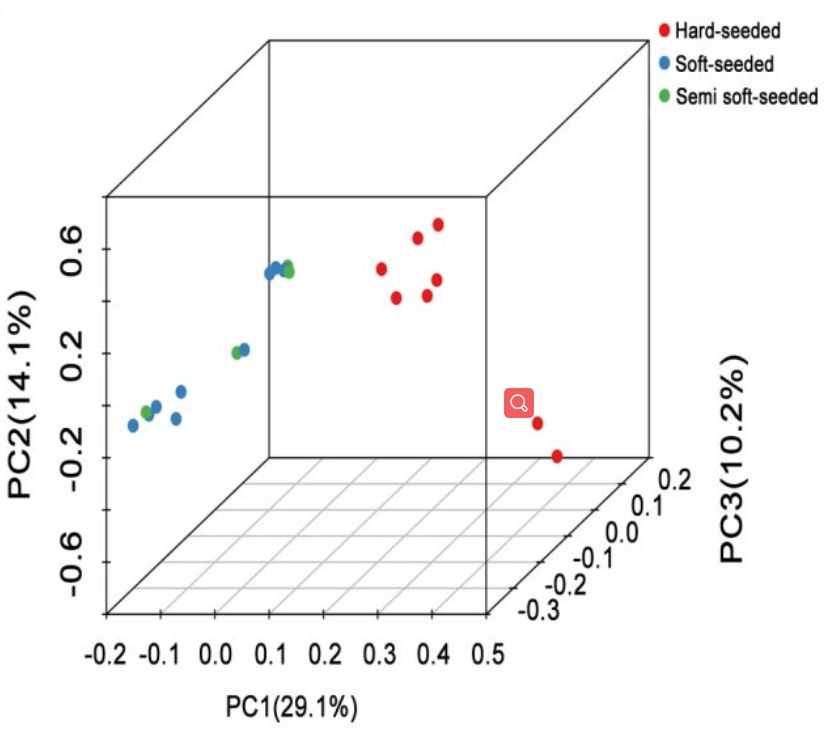

图4 群体多样性分析
LD分析显示,硬籽品种相较于软籽品种的LD衰减更快,种在相同的环境下栽培,而软籽品种更适宜在热带区域栽培,相比较而言硬籽品种有更广阔的栽培区域。由于软籽品种的LD较大,所以在进行标记辅助选择育种时只需要少量标记即可。
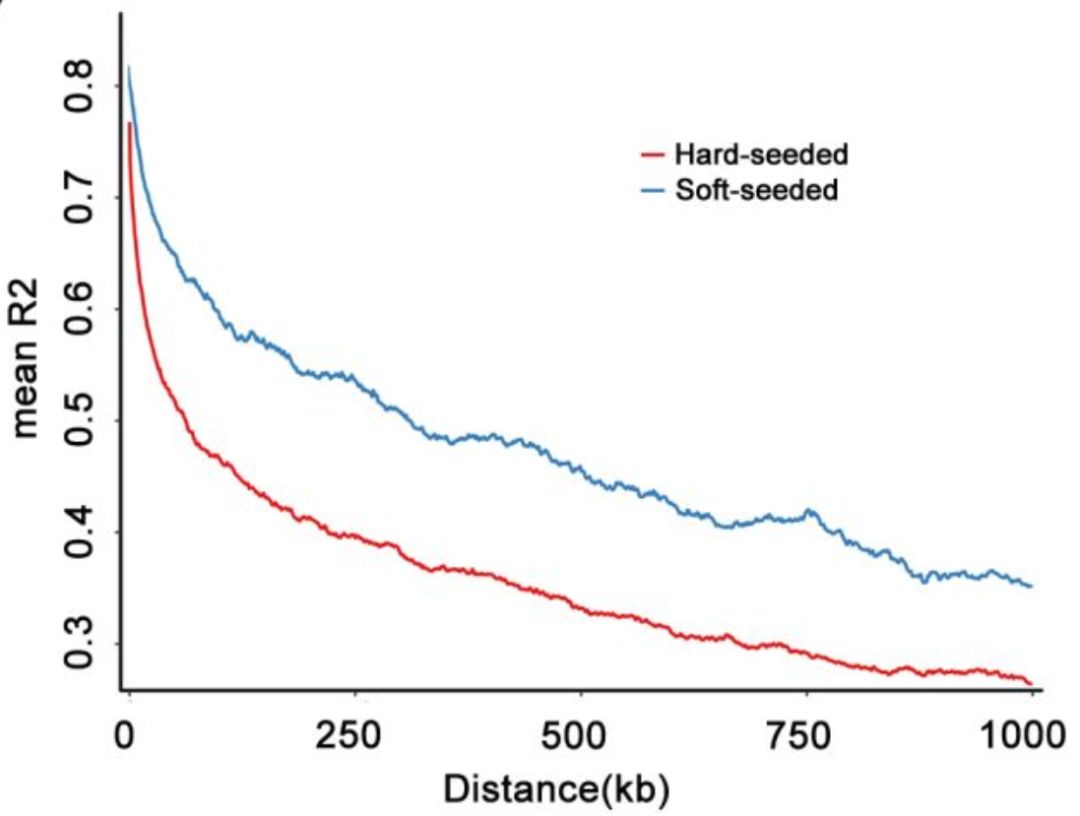
图5 硬籽与软籽石榴连锁不平衡分析
(6)选择性清除分析
为了比较软籽和硬籽品种之间等位基因的多样性,作者比较了8个硬籽品种和12个软籽品种之间的SNP。两个群体间Fst的平均值为0.26,共鉴定到131个区域的Fst值大于0.55。硬籽品种和软籽品种的π值分别为0.00047/kb和0.00038/kb,Tajima’D值分别为1.12和1.0。这些结果表明,硬籽品种比软籽品种的有更高的全基因组核苷酸多样性。
选择性清除区域往往包含与进化相关的基因,于是作者对π分析和Fst分析得到的共有区间进行筛选,得到14和10个受选择的区域,分别包含282个249个位点。
(7)受选择基因与蔗糖转运
前人研究表明,蔗糖的分配与玉米和油菜种子产量相关,蔗糖作为信号在草莓果实发育和成熟中发挥功能,而蔗糖的转运对于棉花纤维的伸长是必需的。作者在3号染色体受选择的区域中发现2个基因PgL0145770和PgL0145810(图6),这2个基因编码蔗糖转运蛋白SUC8-like和SUC6。对Sanbai和Tunisia的籽粒进行转录组分析发现,这2个基因在开花后60天的表达量显著高于120天的表达量,说明这2个基因可能与籽粒发育相关。同时发现,相较于Sanbai(硬籽品种),SUC8-like在Tunisia显著下调表达(图6),这说明该基因组在软籽和硬籽品种间存在差异表达。软籽石榴一般拥有茂盛的叶片、比较厚的果皮和果肉、种子不饱满等特征,而SUC8-like在软籽石榴的下调表达,暗示着光合产物不能高效转运到种子中,从而导致了软籽的表型。
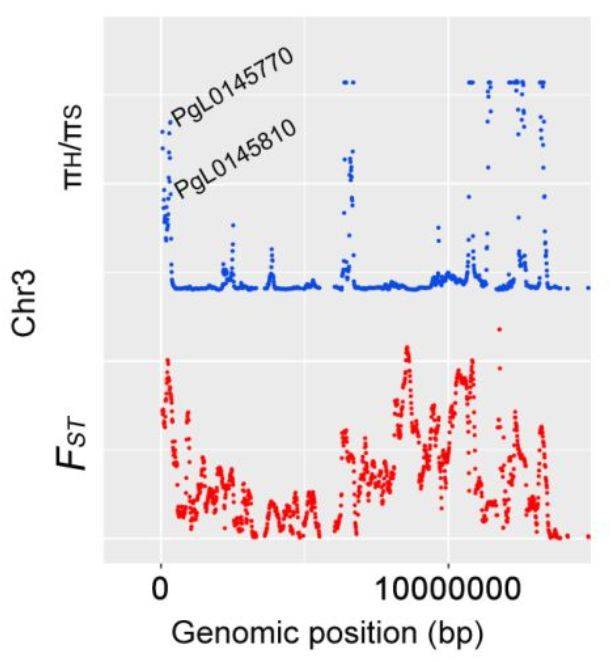
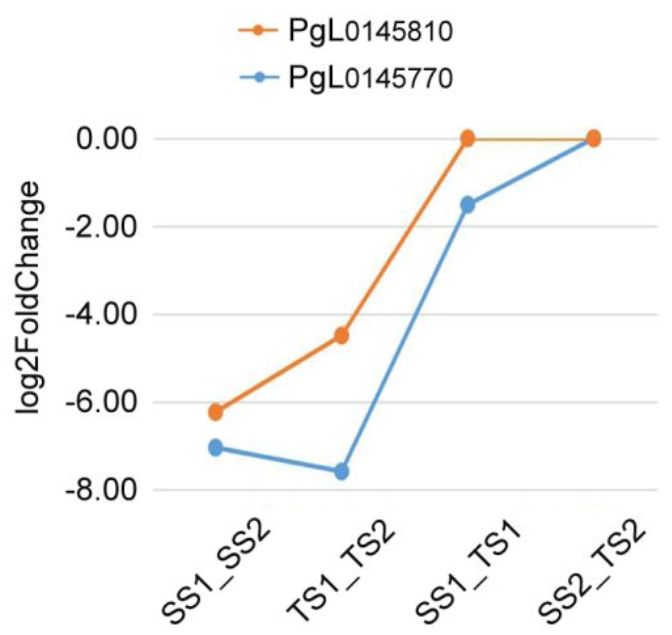
图6 受选择区域与蔗糖转运相关基因及2个基因的表达分析
(8)受选择基因与环境适应性
环境变化产生的选择压力将会促进适应环境变化表型的发展,而定位受环境选择基因将会揭示表型多样的遗传基础。在硬籽品种中,6个受选择的基因(PgL0314630, PgL0218430, PgL0044640, PgL0044700, PgL0044670和PgL0314990)富集到运输和信号转导通路(图7),如FoxO信号通路、MAPK通路。前人研究显示,果蝇、番茄中FoxO和MAPK通路能够响应冷环境。这就很好解释了为什么硬籽石榴比软籽石榴更耐冷环境。
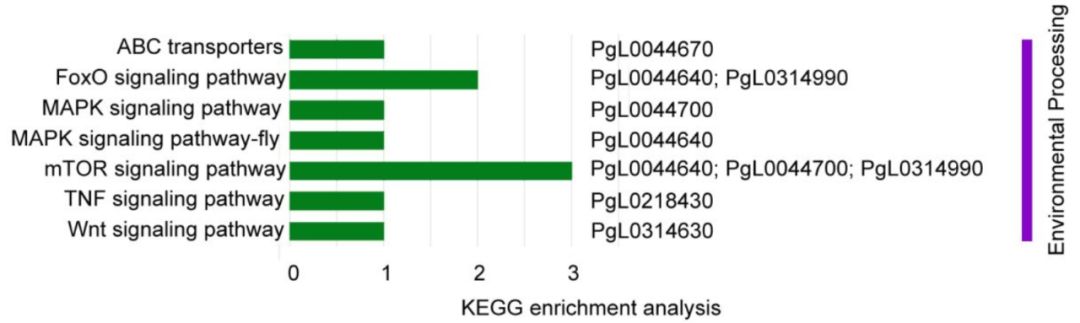
图7 受选择区域与环境适应性相关基因的注释分析
(9)1号染色体上大段受选择区域
本研究发现,硬籽群体中在1号染色体上有一段26.2Mb的受选择区域,这段区域具有较高的π、Tajima’s D(πS/πH > 8, 平均Tajima’s D = 2.08,)和较小的Fst值(Fst<0.3)。在硬籽群体中,该区域SNP的LD衰减速度较快,同时表现出较低的杂合度(图9)。该区域较低的重组率可能是因为存在大尺度的结构变异,也可能是因为该区域处在着丝粒附近。
这个大段的受选择区域可能是由于自然选择造成的,一是因为硬籽石榴群体来自中国,而软籽石榴则来源于美国、意大利、突尼斯或者其他国外品种,这种地理隔离阻碍了杂交的发生。另外,石榴具有一个短暂的育种和人工选择史,硬籽石榴和软籽石榴间并不存在杂交障碍。
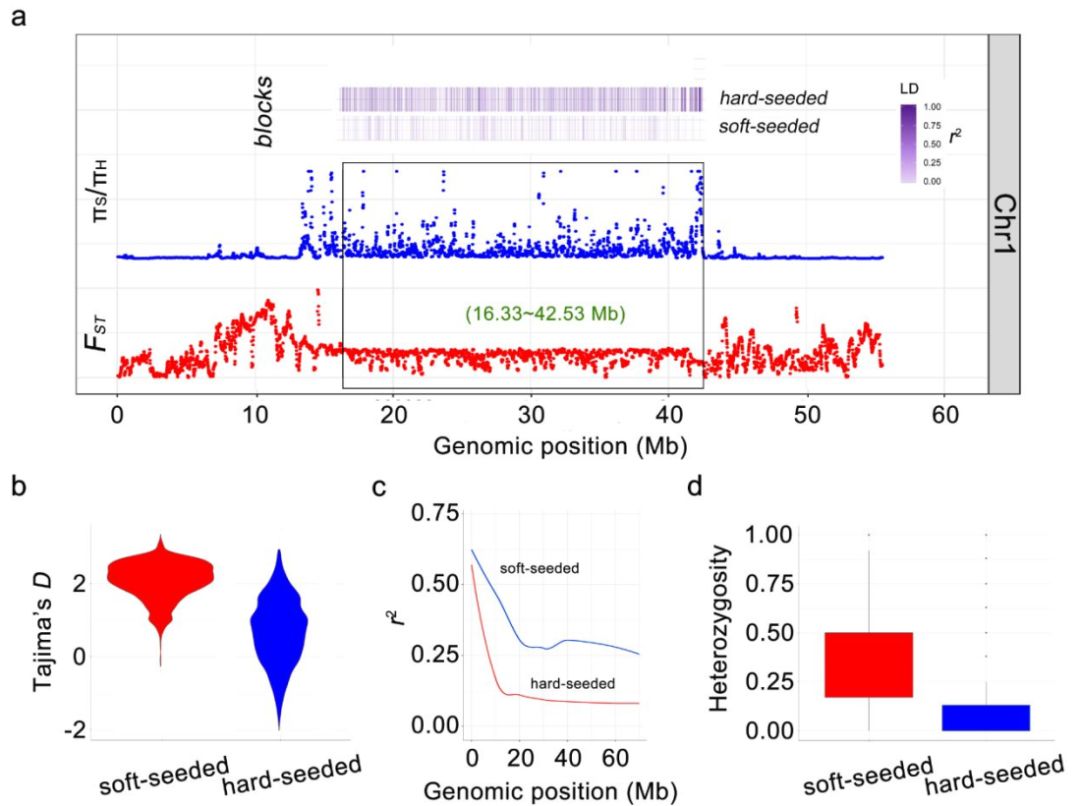
图8 1号染色体上大段受选择的遗传区域及该区间内选择信号、LD及杂合度分析
4.
总结
该研究借助三代Pacbio测序及Hi-C等最新技术,成功破译了软籽石榴栽培品种Tunisia的基因组信息,获得了高质量的石榴基因组,并对软籽和硬籽石榴进行了全基因组水平比较分析,揭示了硬籽和软粒品种之间的遗传差异。该研究成果将极大推动石榴的进化和功能研究,以及石榴品种的驯化和改良。
5.
参考文献
[1] Luo, X.,Li, H., Wu, Z., et al. (2019). The pomegranate (Punica granatum L.) draft genome dissects genetic divergence between soft‐ and hard‐seeded cultivars. Plant Biotechnology Journal.
[2] Yuan, Z. , Fang, Y. , Zhang, T. , et al. (2017). The pomegranate (punica granatum, L.) genome provides insights into fruit quality and ovule developmental biology. Plant Biotechnology Journal.
转自:百迈克- 本文固定链接: https://www.maimengkong.com/zixun/1189.html
- 转载请注明: : 萌小白 2022年9月30日 于 卖萌控的博客 发表
- 百度已收录
