细胞色素P450(cytochromeP450或CYP450),为一类亚铁血红素—硫醇盐蛋白的超家族,它参与内源性物质和包括药物、环境化合物在内的外源性物质的代谢。在真菌中,次级代谢产物的合成和外来化合物的代谢有着重要作用,许多化合物的生物转化是由CYP450介导的,在实际应用上,CYP450在农药降解中也起着重要作用。
虽然Nr数据库中有部分微生物的P450信息,总体来说,信息都不够全面。今天我们来介绍一个比较专业、全面的细胞色素P450数据库。
数据库网址:http://p450.riceblast.snu.ac.kr/index.php?a=view
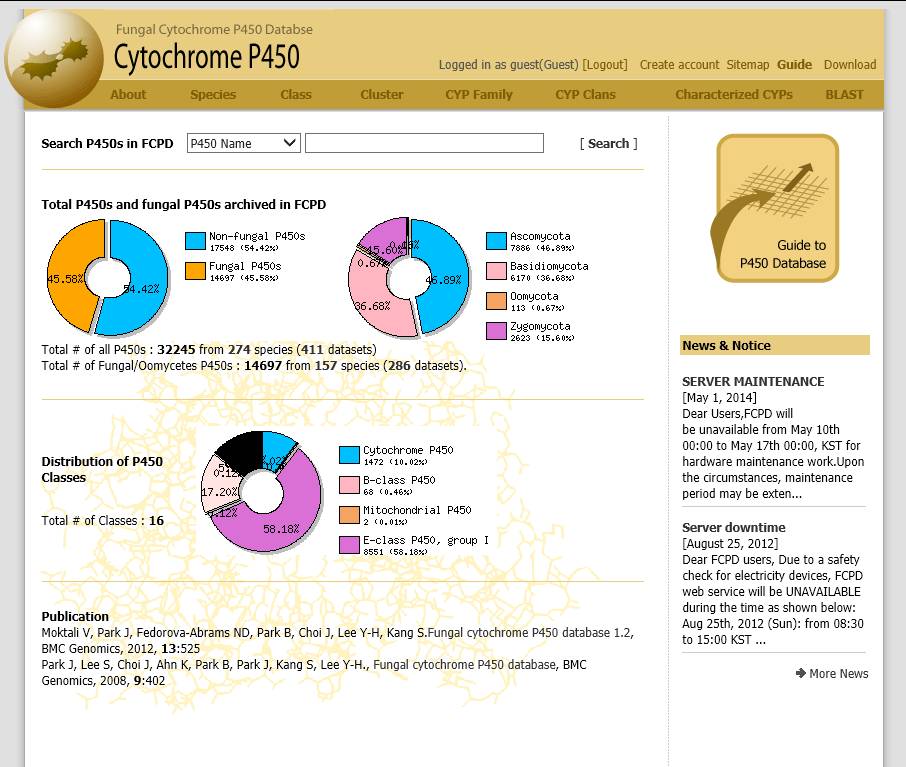
这个数据库中有收录真菌和非真菌(细菌、其他真核生物等)的细胞色素P450,一共收录274个物种的32245个CYP450,其中有157种真菌P450。虽然说这是个真菌数据库,但其实也收录了一部分的细菌、动物和植物。
原理
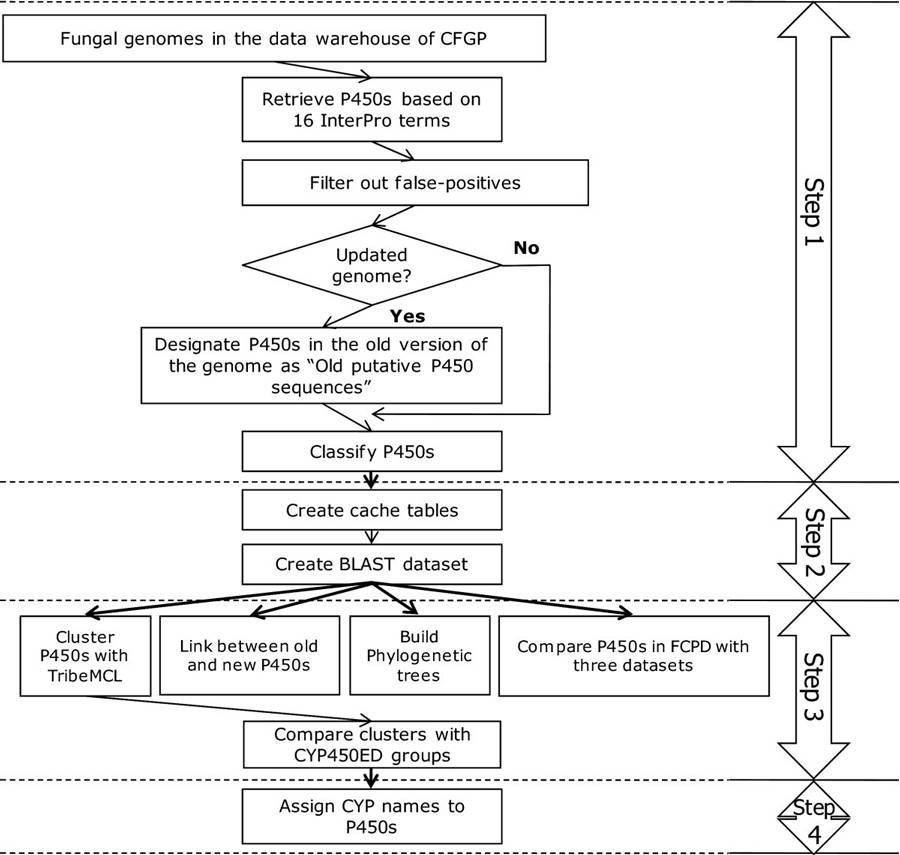
数据库识别P450主要是四个步骤:
-
首先,存入CFGP(comparative fungal genomics platform,一个比较真菌数据库平台,收录了来自432个真菌的667个基因组信息)数据库里的真菌基因组,通过InterPro scan 对每个ORF进行注释,将所有P450s分成了16个InterPro term,然后过滤掉假阳性的结果,过滤的标准是最小长度为25个氨基酸,但是由于一些假阳性的结果却是可能是新的P450,为了不丢弃它们,就标记它们为:questionable P450。
-
建立缓存表,建立BLAST的数据集。
-
基于BLAST数据集,用tribe-MCL聚类P450s,并用三个公共数据库进行比较(the Cytochrome P450 Engineering database[1]、Nelson's P450 database[2]和一个多真菌P450系统发生树分析[3-4])。
-
将数据库中所有的P450s通过BLAST匹配到Nelson's P450数据库中对应的家族,对于每个P450,大于44%的identity认为是高度可靠的,小于44%被认为是低可靠的,不过考虑到P450变化多样,Nelson's P450数据库不如该数据库多,所以低可靠的可能是由于还没有收录在Nelson's P450数据库中。
操作
点击“Species”,会显示被收录的物种,以及对应的P450数量,点击该数字可以进入界面查看具体的P450s。
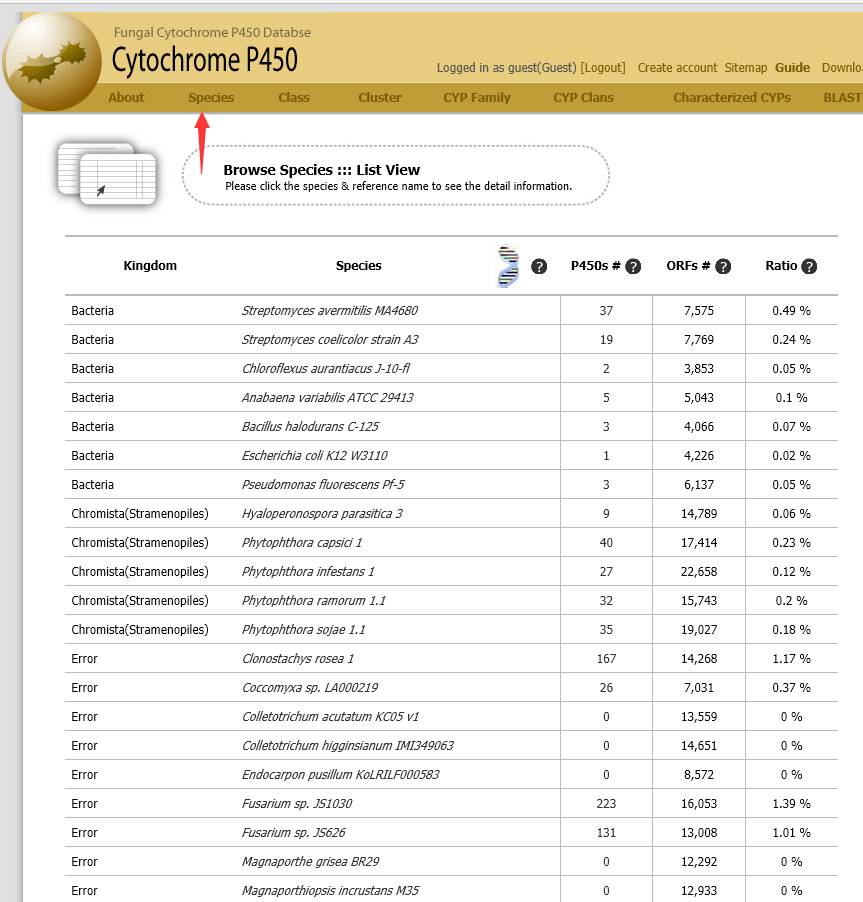
点击对应物种的可以查看具体的P450信息,数据库基于InterPro的term将这些P450分类成16个classes,通过tribe-MCL聚类分成了2579个groups。
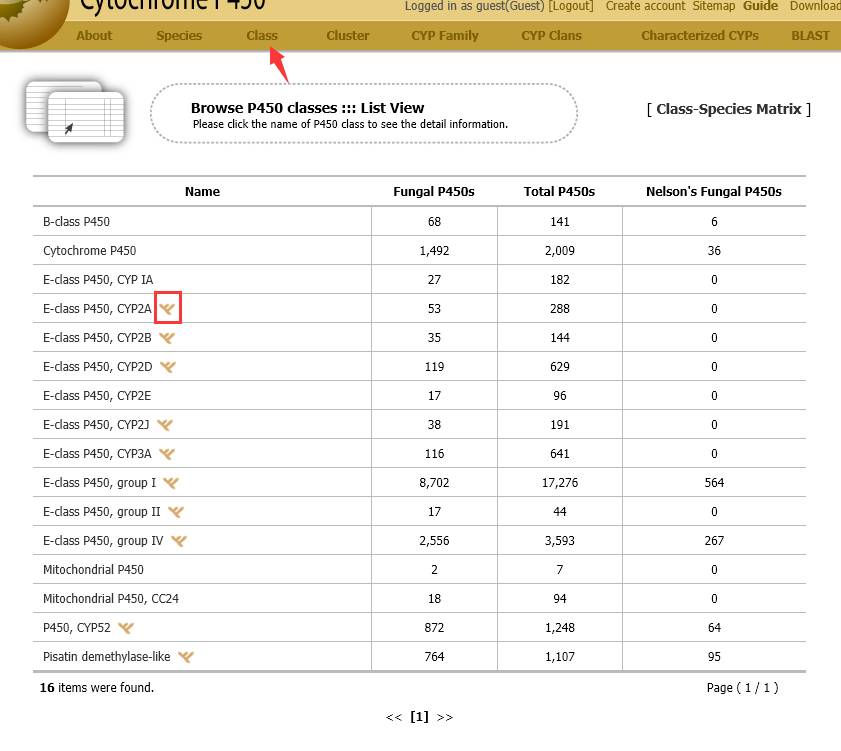
“Class”是P450的分类,同样可以点击进去查看,里面黄色的图标是进化树,点击进去可以看到该分类下的P450的进化树。
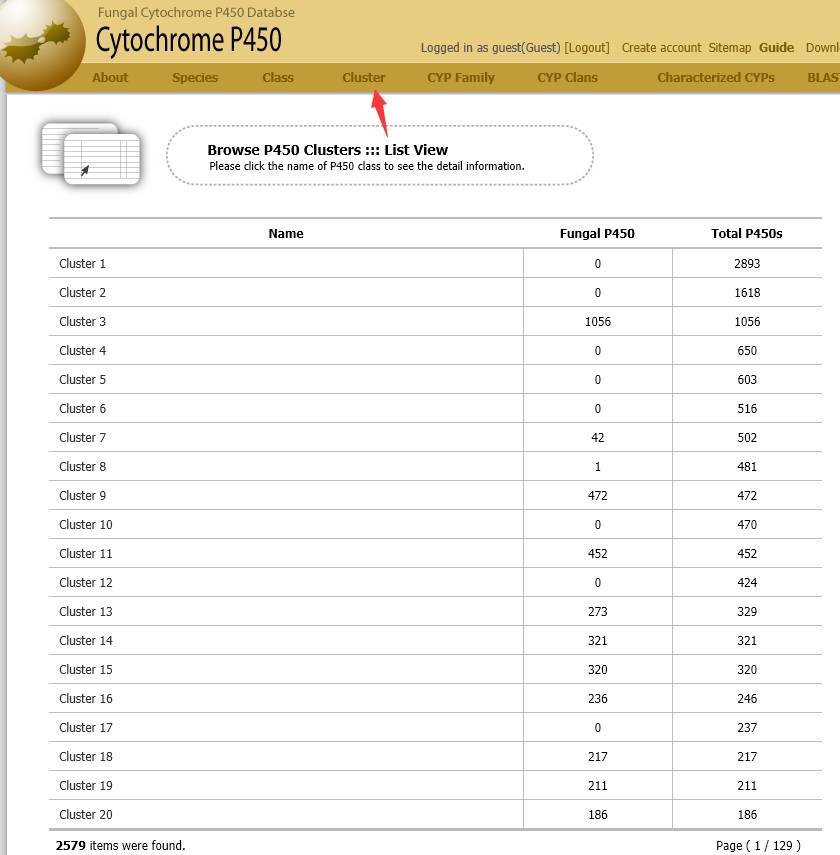
“Cluster”是对P450进行了聚类,列出了属于真菌的P450和总共的P450有多少。
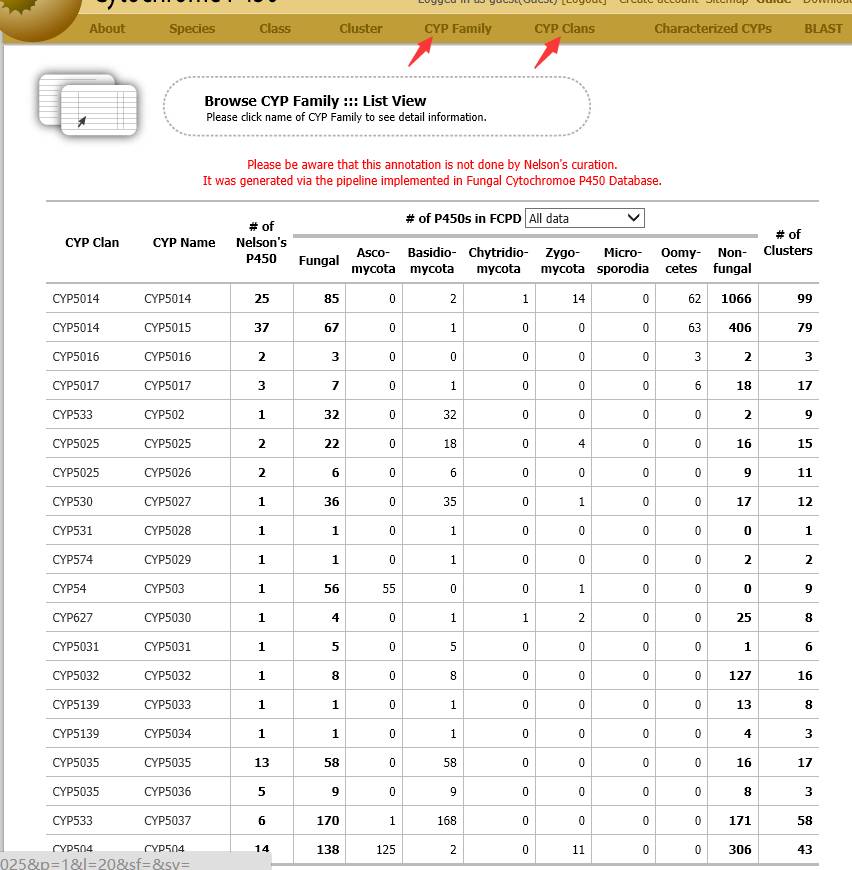
然后“CYP Family”是CYP450的家族分类,对应在真菌,和门上的数量。“CYP Clans”则是比Cluster更上一层的分类,1个clans可能会包含多个family。
最后,这个网站能在线比对。
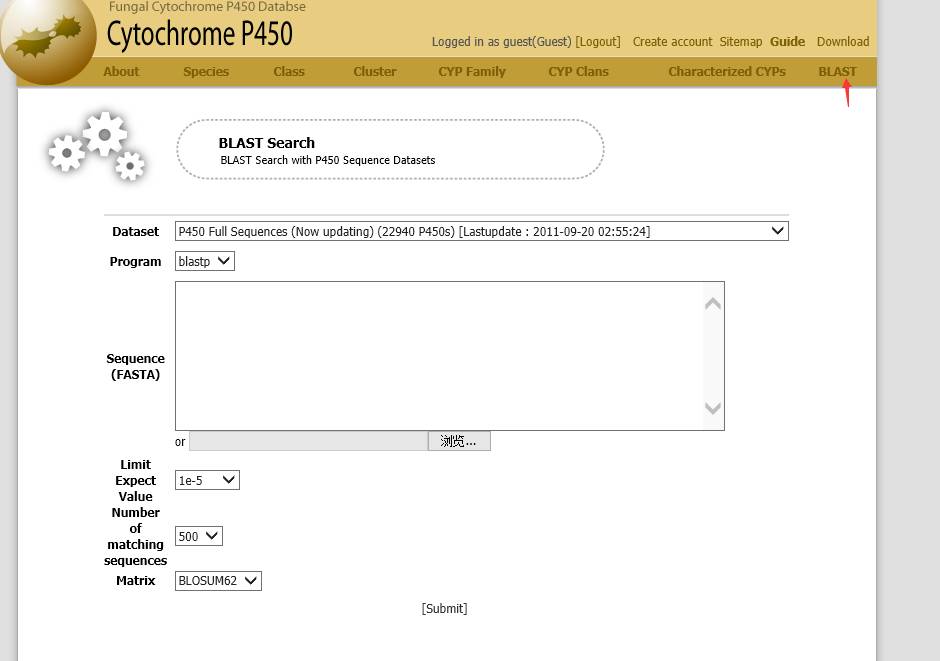
小编尝试着用龙眼的几条序列进行了比对,发现其实里面确实收录了一部分真核的P450s的,比如第三个基因比对上了大豆、茄属、水稻,比对结果和NCBI的比对结果输出形式是相似的,第一排是比对上的P450的信息、物种,接着是得分、长度、E值等信息。

比较
上文提到的Nelson's Fungal P450s(http://drnelson.uthsc.edu/CytochromeP450.html),同样收录了很多P450的信息,主要是一些模式生物或者研究的比较透彻的物种上。
主页信息
这里面的数据库划分了动物、植物、真菌、细菌和一部分菌虫类,根据13年8月的统计,总共有21,039条序列被收录。

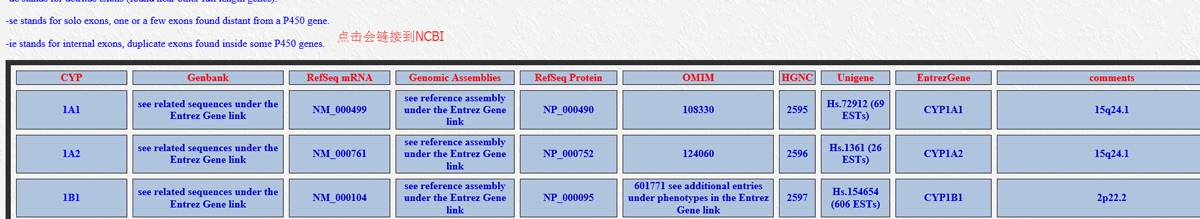
比如点击人的P450:
会出现:

如果点击第一个查看P450的具体信息,点击对应的RefSeq mRNA 、RefSeq Protein 、EntrezGene 等是会直接跳转到NCBI上对应的基因上去。
Nelson是该数据库的负责人,很逗逼的是,他把自己的照片给P到网站上所有用P450s画进化树的图(不能小编一个人辣眼睛~)。
点击有惊喜

这个网站的界面有点看得人眼花缭乱,但是主要核心的是它也能进行blast 比对,往下移可以找到跳转到blast的按钮。
找不到的同学也可以直接输入网址跳转过去(http://blast.uthsc.edu)。
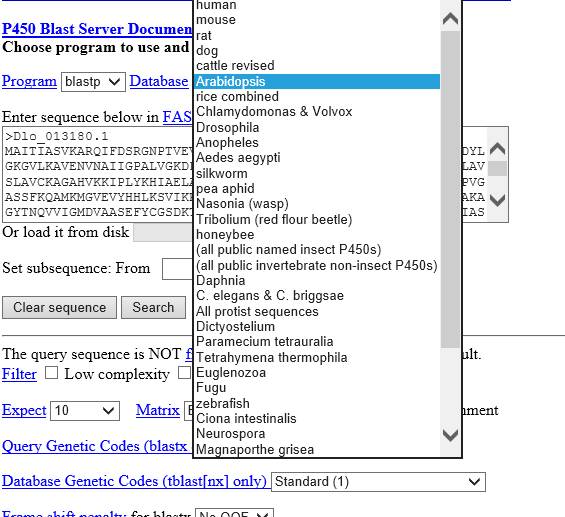
但是它比对需要选择物种,比对结果和NCBI比对的输出结果类似。

所以综上所述,如果你想做的是真菌或细菌的P450查找,可以用CytochromeP450来进行比对;如果做的是动物或者植物,那么可以用Nelson的网站来进行比对。嗯嗯,今天就到这里啦~
参考文献:
【1】Fischer M, Knoll M, Sirim D, Wagner F, Funke S, Pleiss J: The Cytochrome P450 Engineering Database: a navigation and prediction tool for the cytochrome P450 protein family. Bioinformatics. 2007, 23 (15): 2015-2017. 10.1093/bioinformatics/btm268.
【2】Nelson D R. The cytochrome p450 homepage[J]. Human genomics, 2009, 4(1): 59.
【3】Deng J, Carbone I, Dean R: The evolutionary history of Cytochrome P450 genes in four filamentous Ascomycetes. BMC Evol Biol. 2007, 7 (1): 30-10.1186/1471-2148-7-30.
【4】Doddapaneni H, Chakraborty R, Yadav JS: Genome-wide structural and evolutionary analysis of the P450 monooxygenase genes (P450ome) in the white rot fungus Phanerochaete chrysosporium: evidence for gene duplications and extensive gene clustering. BMC Genomics. 2005, 6: 92-10.1186/1471-2164-6-92.- 本文固定链接: https://www.maimengkong.com/moreshare/1186.html
- 转载请注明: : 萌小白 2022年9月30日 于 卖萌控的博客 发表
- 百度已收录
