随着抗生素药物的发现及使用,产生了越来越多的耐药菌株。耐药性会增加疾病治疗的难度和成本,因此耐药微生物的研究显得尤为重要。细菌的多种耐药机制由大量耐药基因所编码。耐药菌在基因水平上的改变主要包括固有耐药性、获得耐药性以及多重耐药性。固有耐药性来源于细菌本身染色体上的耐药基因。获得耐药性则是由于敏感的细菌发生基因突变或获得外源性耐药基因所产生的。多重耐药性则是指细菌同时对多种不同作用机制,或结构完全各异的抗菌药物具有耐药性。当前在全世界范围内,多种病原菌甚至出现了多重耐药株,甚至泛耐药株。
目前利用基因组高通量测序序列数据与生物信息学方法, 可深度剖析细菌耐药机制, 确定新药靶点,寻找新的抗生素,这在一定程度上可以帮助我们解析耐药机制,为疾病治疗、药物研发等提供参考。
这其中最核心的内容就是耐药基因数据库,一个完备、精确、更新及时的数据库,是耐药基因筛查工作的核心。
ARDB是最先整合了各种微生物中抗药基因的数据库,但它从2009年开始就不再更新。
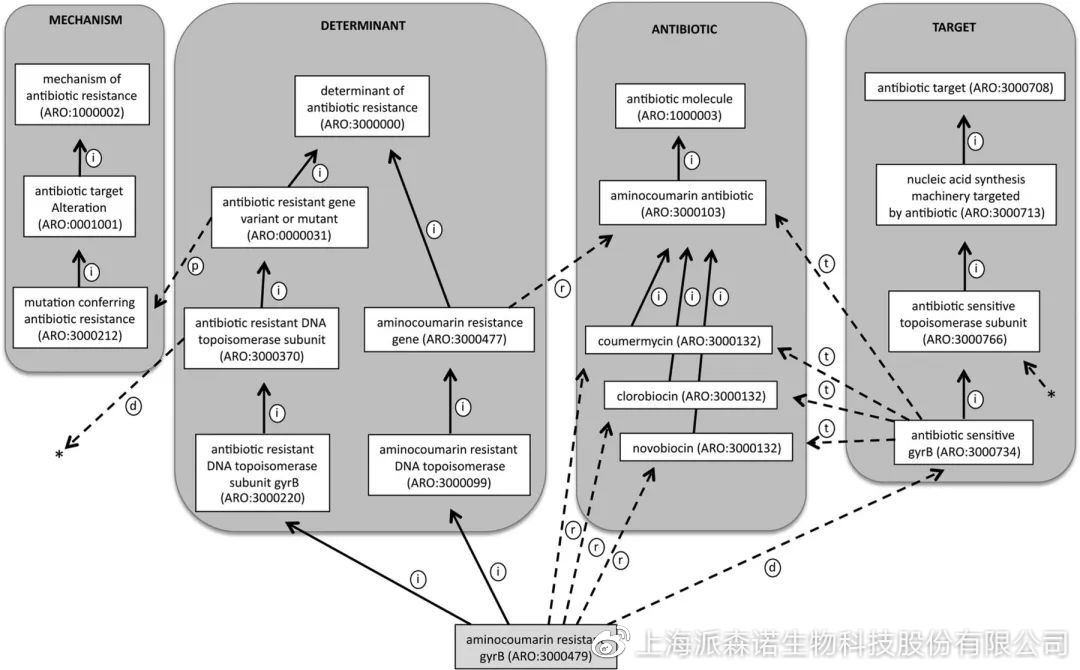
而CARD(the Comprehensive Antibiotic Research Database)数据库包含了ARDB数据库中所有抗性信息,在构建之初就充分考虑到新的耐药基因随抗生素的使用不断产生的问题,搭建了一个数据共享平台,以ARO(Antibiotic Resistance Ontology)为核心对抗性数据进行整合,以达到数据实时更新的目的。
一起来看看如何利用CARD数据库在线进行进行耐药基因预测?
地址:https://card.mcmaster.ca/,点击链接进入CARD数据库;

在CARD数据库网站,点击“Analyze”选项可进入耐药基因预测界面。
耐药基因预测分析可通过选择“BLAST”和“RGI(Resistance Gene Identifier)”两种模式来实现;
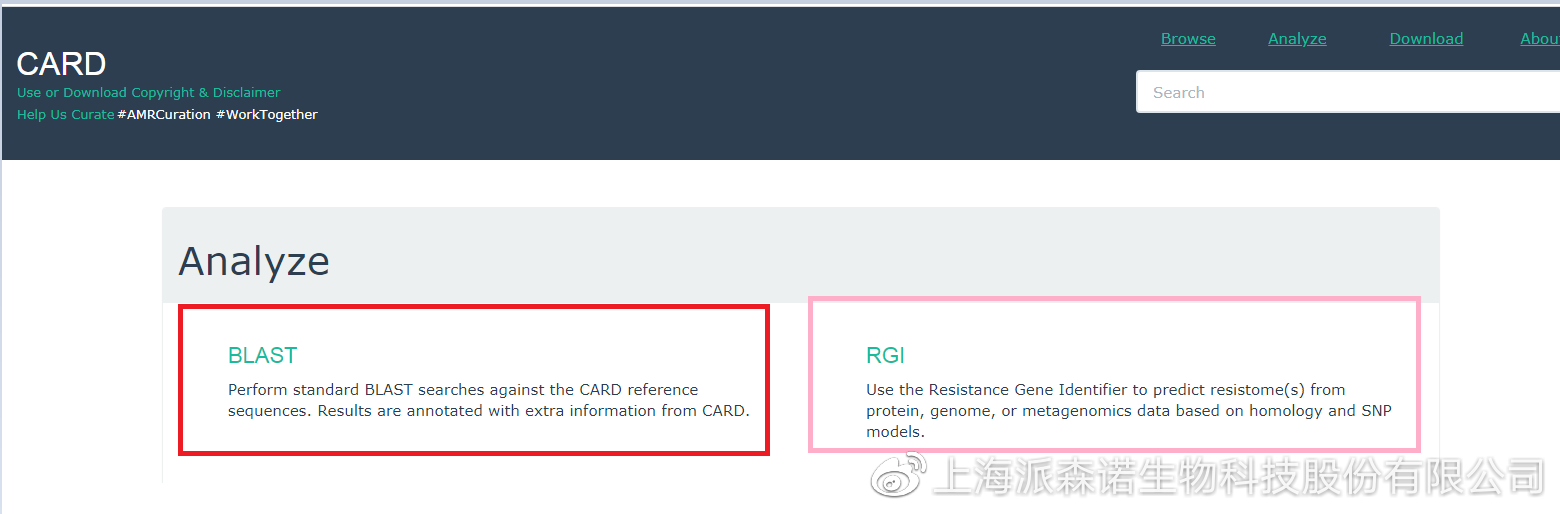
1、 BLAST是依赖NCBI中BLAST软件,将序列与CARD数据库中的参考序列进行比对,获得相关的注释信息;

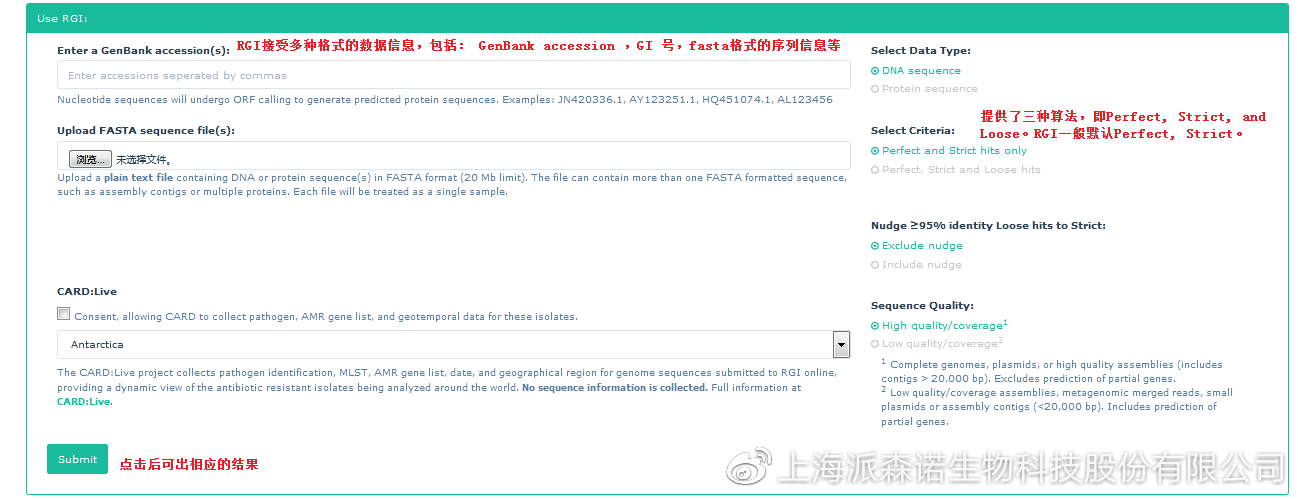
2、 RGI是CARD数据库团队开发的基于蛋白预测抗性基因序列的软件,即通过蛋白同源和蛋白变异两种模式来预测抗性基因序列,同时还提供了画图功能,兼具实用和高效的特性;
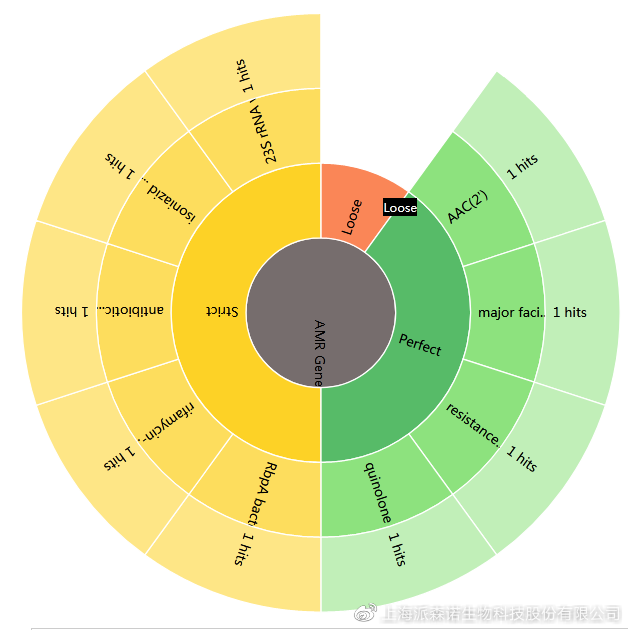
可视化结果展示
以上就是CARD数据库的在线使用,您get到了吗?
快来一起试试吧!
派森诺生物可提供小基因组(细菌、真菌、细胞器、DNA/RNA病毒)测序服务,基于高通量测序技术,获得基因组的图谱信息,并在结构基因组学、比较基因组学层面通过差异分析、同源基因分析、共线性分析、物种进化分析等手段探究小基因组的性质,为溯源和进化研究等提供证据。
- 本文固定链接: https://www.maimengkong.com/learn/767.html
- 转载请注明: : 萌小白 2021年8月25日 于 卖萌控的博客 发表
- 百度已收录
