荧光素酶因为具有灵敏度高、检测快速、方便等特点而被广泛开发应用。
萤光素酶是理想的报告基因,哺乳动物细胞中不含内源性萤光素酶,且其一转录完成立刻就生成功能性的萤光素酶。荧光素酶(Luciferase)报告系统是指以荧光素(luciferin)为底物来检测萤火虫荧光素酶活性的一种报告系统。荧光素酶可以催化luciferin氧化成oxyluciferin,在luciferin氧化的过程中,会发出生物荧光(bioluminescence)。底物过量时,光强能反映荧光素酶的表达量。
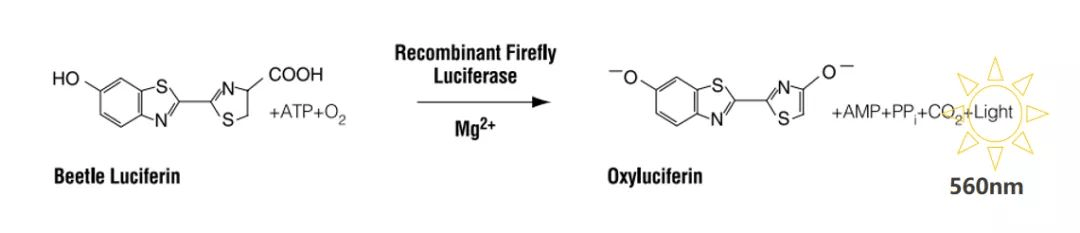
荧光强度与荧光酶活性的成正比,而荧光素酶的蛋白表达量与启动子活性、mRNA的稳定性及翻译效率等因素相关。根据此原理可以将待测启动子、UTR、增强子等序列与荧光素酶ORF框融合表达,再检测其发光强度,即可对荧光素酶报告基因的转录进行间接估计。为了减少实验误差,同时转染一个能稳定表达的海肾荧光素酶的质粒作为内参,所以叫双荧光素酶报告基因检测。
简单来讲,荧光素酶实验即以Luciferase为核心,把感兴趣的基因转录调控元件克隆在荧光素酶基因(firefly luciferase)的上下游,构建成荧光素酶报告质粒。然后转染细胞,适当刺激或处理后裂解细胞,分别加荧光素酶底物,用荧光测定仪检测荧光强度,通过数据得出基因表达量,从而确定构建序列的功能。
荧光素酶实验常用于:
A 启动子活性分析
可以用于验证待测启动子是否有活性及其活性区域;组织特异性启动子筛选及确定;新顺式作用元件发现及功能鉴定。
启动子区是RNA聚合酶的结合区,启动子的结构直接关系到转录的效率。
启动子中的-25和-70序列是RNA聚合酶所结合和作用必需的顺序。-25-30TATA框(TATAbox)和-70-78CAAT框(CAAT box-CCAAT)是RNA聚合酶与启动子的结合位点,TATA框的主要作用是使转录精确地起始;CAAT框和GC框则主要是控制转录起始的频率,特别是CAAT框对转录起始频率的作用更大。
但是启动子的其它结构,包括正调控区域、负调控区域以及增强子也能影响启动子的功能。为了研究具体的结构域,可以将待测启动子区域序列进行分段截短,缺失或突变掉特定位点,再分别插入LUC报告基因上游,检测其启动子的活性,用这种方式逐步确定启动子的功能域。
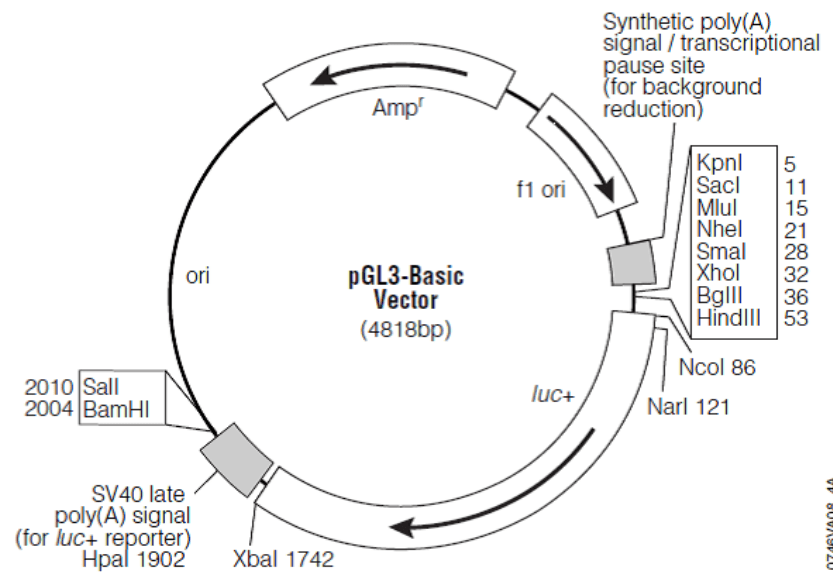
B 启动子与转录因子的结合验证
用于研究某转录因子是否调控某基因的启动子,以及它们的结合区域。
转录因子(TFs)是调节基因表达,以保证目的基因以特定的强度在特定的时间与空间表达的蛋白质分子。研究转录因子与启动子功能时,将启动子序列插入到报告基因载体(常用载体为pGL3-Basic Vector,将待检测的启动子序列构建在报告基因上游),同时在细胞实验中共表达转录因子,分析荧光素酶表达水平,反映转录因子是否调控基因的表达。
确定转录因子与对应的启动子有调控功能后,可以对启动子进行截断(网站可以预测到的,根据预测的结合位点对启动子进行截断或者突变;无法预测的可以直接每缺失500bp进行截断)。通过荧光值对比确定哪一区域为核心区,luciferase表达强度的高低说明转录因子能增强或抑制启动子的启动能力。
注意:启动子截断是逐步缺失远端序列(因为核心启动子位于靠近TSS端,缺失该处会丧失基本转录活性);
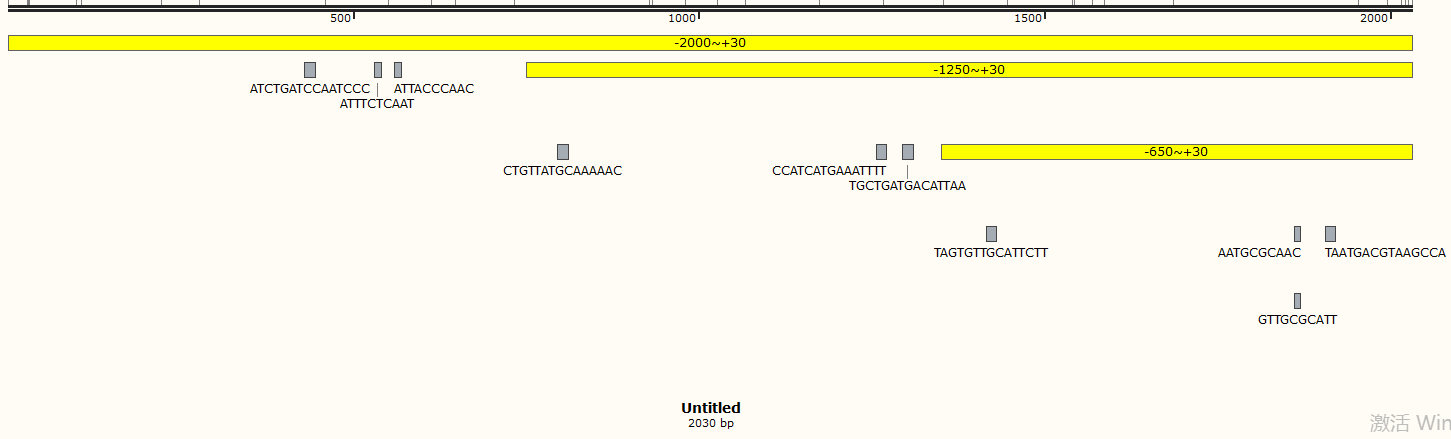
根据预测的结合位点对启动子进行截断
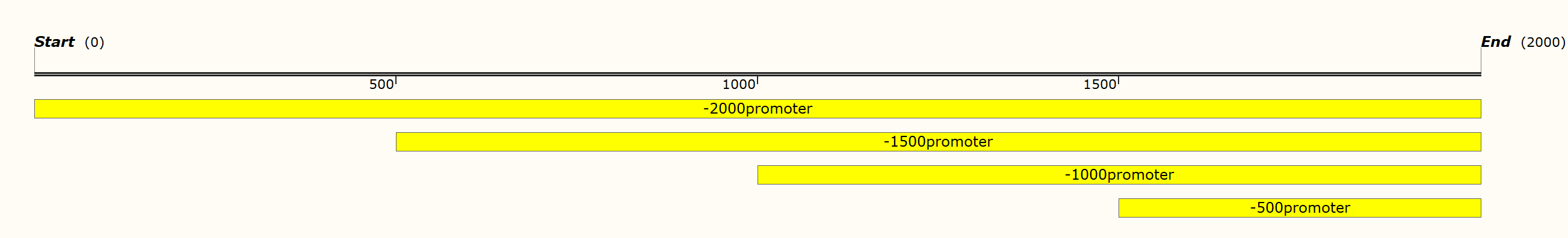
每缺失500bp进行截断
C 研究microRNA与靶基因是否存在调控关系
microRNA主要通过作用于靶基因的3’UTR起作用,microRNA大部分通过成熟体种子区与3’UTR结合抑制蛋白翻译。可以将目的基因3’UTR区域构建至报告基因luciferase的下游,再共转入microRNA,如果荧光素酶表达下降,可以说明microRNA对目的基因的抑制作用,则待验证序列是microRNA靶序列。出于严谨的考虑,通过结合定点突变进一步确定microRNA与靶基因3’UTR的作用位点。
microRNA与靶基因的荧光素酶实验分组如下:

D 5’UTR和3’UTR功能验证
对于编码基因,通常都会有5’UTR和3‘UTR,这些编码基因的5’UTR和(或)3‘UTR可能会对基因的表达有影响。
研究5’UTR:将编码基因的5’UTR构建到pGL3 promoter载体的荧光素酶报告基因的上游,SV40启动子下游,通过这种结构来判断5UTR’对基因表达的影响。
而对于3’UTR:将编码基因的3’UTR构建到pGL3 promoter载体萤火虫荧光素酶报告基因的下游,通过这种结构来预测3’UTR的功能,这里的3’UTR通常都是全长。
E 位于增强子内的非编码性SNP
单个核苷酸变异,简称SNP。
通过构建报告系统研究SNP位点的增强子与转录因子是否结合最为直接,根据SNP位置不同,构建方法也是不一样的,具体如下:
1. SNP位于启动子区域(一般5k以内):直接构建启动子全长至PGL3 basic
2. SNP位于启动子上游远端:构建SNP位置1k左右的序列与目的基因启动子联合构建至PGL3 basic
3.SNP位于基因内含子或者基因下游:构建SNP位置1k左右的序列至PGL3 promoter的luc下游,也可将载体本身SV40启动子替换为目的基因启动子。
双荧光素酶报告系统构建注意事项及常见问题解答:
1 公司的载体GV272、GV238为单标载体,若客户自己操作实验,需单独构建海肾系统,录入对照质粒出货合同,编号CV045。
2 公司推荐构建单标载体,因为文献使用多,公司实验体系成熟;客户指定双标载体也可以。
3 常规表达载体基因跟luc构建至一起,不是做调控活性验证的,luc就是一个检测标签。
4 luc不建议与目的基因融合,因为luc太大了,融合可能影响目的蛋白的结构。
5 验证结合的是不是不需要做抗性筛选呢?
293细胞非常容易转染,因此不需要抗性,带了抗性反而会增大质粒骨架导致转染效率降低。
6 用慢病毒做荧光素酶实验还需要海肾荧光素酶吗?
慢病毒做成稳转株,实际上就可以不需要海肾了;
海肾本身是平衡不同组的细胞状态,质粒转染效率会有差异,做成稳转株,就不存在转染效率等问题了。当然加了海肾更好,可以选择做一个。
- 本文固定链接: https://www.maimengkong.com/kyjc/1438.html
- 转载请注明: : 萌小白 2023年4月15日 于 卖萌控的博客 发表
- 百度已收录
