火山今始见,突兀蒲昌东。——岑参《经火山》
大家好,我是阿琛。当我们通过分析得到基因在不同分组中的表达情况,以及显著上调或下调的差异基因时,如何将该结果进行可视化展示随之出现。在生信分析中,火山图和热图是两种最为常见的展示方法。火山图,Volcano Plot,因其形状类似喷发的火山而得名。

1.R包得安装与读取
首先,自然是R包的选择与安装。经过多期内容的讲解,相信大家对于R语言绘图也基本有了一个基本的了解。 作图常用三大包,分别是base包,强大的ggplot2包,以及在ggplot2基础上发展而来的ggpubr包。对于点图的绘制,在此我们选择ggpubr包来讲讲火山图的绘制。
#install.packages( "ggpubr") #install.packages( "ggthemes") #加载ggpubr包 library(ggpubr) library(ggthemes)
2.数据集得加载与引用
rt<- read.table( "TCGA.diff.txt", sep= " ",header=T) head(rt) #显示前6行
结果显示,在该数据集中,包括了基因名,正常组和对照组的表达平均值,以及差异分析得到的logFC, P值,以及校正后的fdr值。

str(rt) #查看数据结构
通过str函数简单查看每个变量的数据类型。

3.数据内容的整理
#对fdr值进行取对数处理 rt $logP<- -log10(rt $fdr) #定义显著上/下调基因 rt $Type<- "no"#新增一列Type rt $Type[ which((rt $fdr< 0.05) & (rt $logFC> 1))] <- "up-regulated" rt $Type[ which((rt $fdr< 0.05) & (rt $logFC< -1))] <- "down-regulated" table(rt $Type) #对Type中的数量进行统计
到此,整个数据的准备工作就基本完成了,其中显著上调的基因共23个,显著下调的基因共468个。
4.绘制火山图
接下来,我们一起来看下如何一步步由浅入深,逐步绘制火山图逐步为火山图添砖加瓦,增加各种信息。
ggscatter(rt, x = "logFC", y = "logP") + theme_base + ylim( -0.2, 17)
首先,在ggscatter函数中对数据集和图形的x轴、y轴进行定义,得到整个火山图的初步框架。

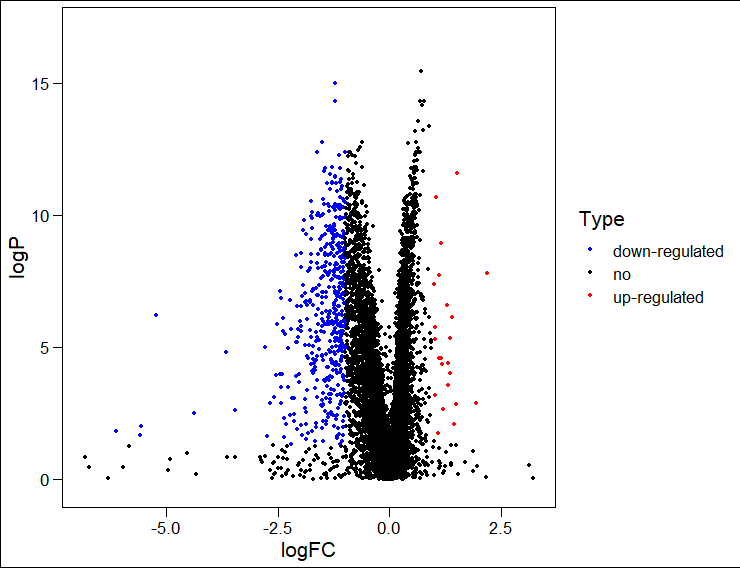
ggscatter(rt, x = "logFC", y = "logP", color = "Type", palette = c( "blue", "black", "red"), size = 1) + theme_base + ylim(- 0.2, 17)
随后,对基因中显著上调或者下调的基因颜色进行定义,赋予上调的基因红色,以及下调的基因蓝色,进行可视化的区分。
ggscatter(rt, x = "logFC", y = "logP", color = "Type", palette = c( "blue", "black", "red"), size = 1) + theme_base + ylim(- 0.2, 17) + geom_hline(yintercept = -log10( 0.05), linetype = "dashed") + geom_vline(xintercept = c(- 1, 1), linetype = "dashed")
通过geom hline和geomvline两个函数,分别在x轴和y轴方向上添加三条辅助性的虚线,将显著改变的基因与其他基因进行区分开来。
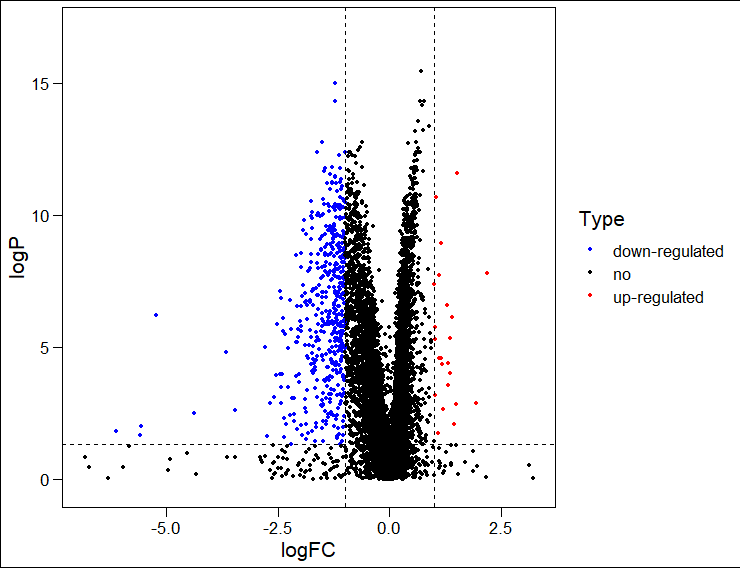
到此为止,火山图也就基本绘制完成了。当然,有些小伙伴可能还在文章中见过带基因名字标签的高级版火山图。接下来,我们就来看下如何对显著上调或下调的5个点添加基因标签。
rt $Name= ""#新加一列Name rt <- rt[order(rt $fdr), ] #对差异基因的p值进行从小到大的排序 #高表达的基因中,选择fdr值最小的5个 up.genes <- head(rt $gene[ which(rt $Type== "up-regulated")], 5) #低表达的基因中,选择fdr值最小的5个 down.genes <- head(rt $gene[ which(rt $Type== "down-regulated")], 5) #将up.genes和down.genes合并,并加入到Name中 rt.top.genes <- c(as.character(up.genes), as.character(down.genes)) rt $Name[match(rt.top.genes, rt $gene)] <- rt.top.genes
新增一个名为Name的列,通过排序,分别筛选出fdr值最小的显著上调和下调基因,并将他们的基因名赋予给Name列。
#绘制火山图 ggscatter(rt, x = "logFC", y = "logP", color = "Type", palette = c( "blue", "black", "red"), size = 1, label = rt $Label, font.label = 8, repel = T, xlab = "log2FoldChange", ylab = "-log10(Adjust P-value)") + theme_base + ylim(-0.2, 17) + geom_hline(yintercept = -log10(0.05), linetype = "dashed") + geom_vline(xintercept = c(-1, 1), linetype = "dashed")
这样,一张精美的高级版火山图就绘制完成了。

差异分析搭配火山图来展示,是不是十分完美呢?好了,今天的分享就到此为止了,大家根据讲解自己进行相关的练习~~~







