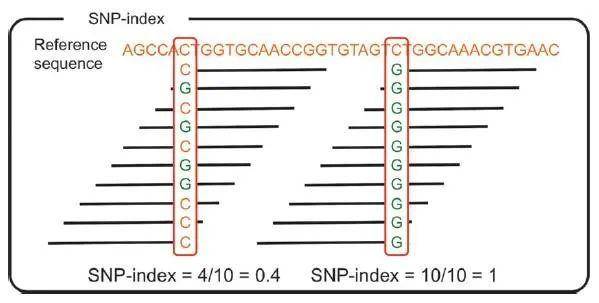
SNP-index这个词,作为BSA的最常用的核心算法,给出的是BSA类项目最重要的QTL定位结果。SNP-index通过结果的可视化,可以非常直观地看出BSA定位得到的QTL在基因组上的位置。例如下图是一个典型的SNP-index的展示结果:
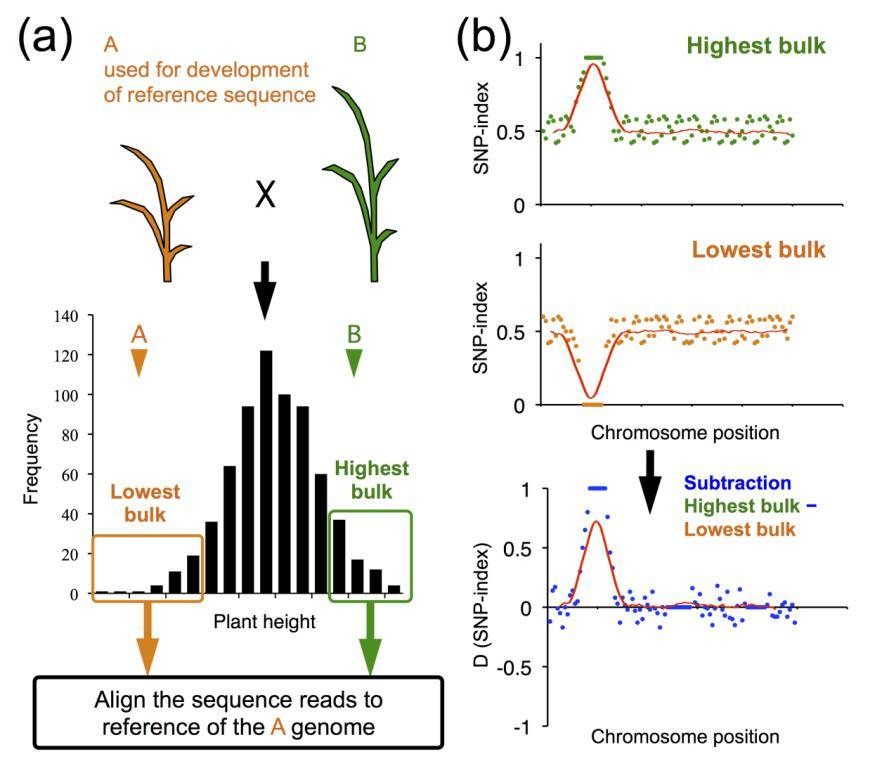
上图中,第一行和第二行分别代表两个子代极端性状混池的SNP-index结果,第三行是两个子代池的△SNP-index的结果。通过第三行我们可以看出,QTL被定为在了某一条染色体的末端位置上。但是这张SNP-index的图具体是怎么解读的,图中黑色的线条,彩色的散点以及红色的虚线分别代表什么呢?我们需要从SNP-index的原理开始讲起。
SNP-index作为BSA定位的算法,最早在2013年被提出(Takagi)。它的基本原理是,构建子代分离群体,经过挑选极端性状构建混池后对SNP进行检测,对各混池进行等位基因频率分析,并与其中一个亲本进行比较。与此亲本不同的基因型所占的比例,即为该位点的SNP-index。从下图可以看到,两个位点的SNP-index分别为0.4和1。值得注意的是,这里的reference指的并不是我们进行重测序变异检测的参考基因组,而是我们构建群体所使用的亲本。这也是为什么进行SNP-index计算必须依赖于亲本测序数据的缘故。
每个混池都得到一组SNP-index数据之后,两个混池相减,即得到了△SNP-index的结果,代表的是两个混池之间SNP基因型频率的差异。理论上说,不与性状相关的位点,△SNP-index的值应当在0左右,代表混池之间不存在差异;而QTL及其相连锁位置的SNP,△SNP-index值应当呈现较高的数值。
不过,通过高通量测序得到的SNP数据通常以数十万计,甚至可以上百万,在这其中也会存在大量因为统计偏差导致的假阳性位点,即△SNP-index高,但是实际上不与QTL连锁的SNP位点。这时我们可以通过计算一个窗口内所有SNP的△SNP-index,即滑窗拟合的方式来消除其影响,得到真正QTL所在的基因组区域。下图即形象地说明了这一过程。
在实际进行BSA分析的过程中,也经常会见到△SNP-index的峰值小于1的情况,但这并不一定说明定位的结果或者群体构建有问题。例如突变性状是显性质量性状,选取的极端性状子代中可能包含基因型杂合的个体,△SNP-index自然就会低于1。如果性状受到环境和遗传因素的共同影响,那么得到的△SNP-index信号自然也会相应降低。除此之外,还有分离群体大小不足、选取的混池样本数量不足等因素,也都会对△SNP-index的信号峰值造成一定影响,需要结合具体案例进行分析。

参考文献:
Takagi H, Abe A,Yoshida K, et al. QTL‐seq: rapid mapping of quantitative trait loci in riceby wholegenome resequencing of DNA from two bulked populations[J]. Plant Journal,2013,74(1):174-83.
Nat Eco&Evo:稗草重测序揭秘稻田里的“伪装者” | 群体遗传- 本文固定链接: https://www.maimengkong.com/zu/1215.html
- 转载请注明: : 萌小白 2022年10月2日 于 卖萌控的博客 发表
- 百度已收录
