2019年07月05日,Nature Communications在线发表了中国农业科学院棉花研究所与北京百迈客生物科技有限公司共同合作文章“Extensive intraspecific gene order and gene structural variations in uplandcotton cultivars”
详细研究内容如下:
英文题目:Extensive intraspecific gene order and gene structural variations in upland cotton cultivars;
中文题目:陆地棉栽培种广泛的基因顺序和基因结构变异;
发表杂志:Nature Communications;
影响因子:11.878;
发表时间:2019.07.05;
合作单位:中国农业科学院棉花研究所;
摘要
目前已有很多棉花基因组组装完成(包含二倍体和四倍体),然而,在世界范围内广泛种植的棉花品种——异源四倍体陆地棉(GossypiumhirsutumL.),其种间的基因组变异仍然是未知的。本研究中研究人员利用了Single-molecule long reads和Hi-C测序技术,组装了2个陆地棉栽培种TM-1和zhongmiansuo24(ZM24)的基因组。通过组装完成的TM-1和zhongmiansuo24(ZM24)的基因组与二倍体祖先种之间的比较,发现了大量的遗传变异。其中,前3个最长的结构变异位于四倍体陆地棉的A08染色体上,占该染色体总长度的约30%。这两个栽培种和种质panel的作图群体的单倍型分析显示该区域的重组率受到抑制。本研究为棉花研究领域提供了更多的基因组资源,鉴定的遗传变异,尤其是A08染色体上减少的减数分裂重组,将对未来的棉花育种具有帮助。
研究背景
陆地棉(G. hirsutum)不仅仅是再生纺织纤维的主要来源,而且是研究多倍体很好的材料。异源四倍体棉源于一次单独的多倍化事件,大约在1-2百万年前(MYA),整合了A基因组(resemblingG. arboreum)和D基因组(resemblingG. raimondii)棉花A和D基因组的组装已经发表。迄今为止,关于陆地棉基因组学的大部分研究工作都集中在TM-1上,其基因组草图于2015年发布,后续发布了更新版本基因组。总而言之,这些基因组资源使的棉花的研究进入了一个新时代,并能够剖析出多种经济和科学上重要性状特征机制的基础。
与许多其他作物物种相比,棉花种质的遗传多样性较低,这使得棉花遗传育种特别具有挑战性,克服该问题的一种解决方案是通过鉴定相关的基因组特征来提高棉花育种的分辨率,为了实现这一目标,本研究组装了2个陆地棉栽培品种:遗传研究标准种TM-1和生物技术重要品种ZM24。与TM-1不同,ZM24易于转化,目前鉴于很多国家都种植了转基因组栽培种棉花,对ZM24基因组的破译及探索其区别于TM-1的不同转化能力,可以进一步促进生物技术在棉花生产中的应用。
在本研究中,研究人员利用了PacBio SMART长read和Hi-C(high throughput chromosome conformation capture)技术组装了TM-1和ZM24的基因组,TM-1和 ZM24分别组装到~2,286 Mb和~2,295 Mb,并挂载到26条染色体上,预估覆盖度超过组装结果的99%。除了907,682 SNPs,99,329 InDels,还有超过100 Mb的PAV变异(PAVs;fragment length>100 bp),研究人员在 ZM24和TM-1的A08染色体上鉴定到了大的反转变异。值得注意的是,
在该倒位区域内的单倍型分组信息及基于300,000多个SNPs的系统发育分析同样将大量的陆地棉种质进行了类似的聚类。本研究中开发的基因组资源可以促进基础植物生物学研究和棉花应用育种。
材料方法
材料:G. hirsutumL. acc. TM-1和ZM24 ;181 RILs(ZM24,maternal parent;TM-1 ,paternal parent);
基因组denovo策略:三代测序组装(PacBio Sequel,SMRT,20 Kb)+转录组RNA-seq基因预测(PacBio Sequel,叶,花,愈伤组织,胚珠,棉纤维,鱼雷形胚和子叶胚)+二代测序(HiSeq 2500,PE150;TM-1,~29.64×;ZM24,~49.63×;RILs parent:~33.32×,RILs progeny:~1.46×)+Hi-C染色体挂载(LACHESIS)+遗传图(已发表);
基因组组装注释:基因组组装:Canu,Falcon,Pilon;BUSCOs 完整性评估:TM-1,1,420 (98.61%) ;ZM24,1,419 (98.54%) ;重复序列预测:LTR-FINDER,MITE-Hunter,RepeatScout v1.0.5和PILER-DF,PASTEClassifier v1.0,RepeatMasker program v4.0.6;蛋白编码基因预测:从头预测(Genscan v1.0, Augustus v2.5.5,GlimmerHMMv3.0.1,GeneID v1.3和SNAP);同源蛋白预测(GeMoMav1.4.2)和转录组Iso-Seq预测(BLAT,PASA),EVidenceModeler进行预测结果整合,TM-1和 ZM24分别预测了73,624 和73,707个基因;基因注释:COG, KEGG 67 , NCBI-NR(version May 2013)和Swiss-Prot,Blast2GO;
结构变异检测:染色体结构变异鉴定:SNPs,InDel,Inversions,Translocations,GAP (gap between two mutually consistent alignments),DUP (inserted duplication),BRK (other inserted sequence),JMP (rearrangement),INV (rearrangement with inversion),SEQ (rearrangement with another sequence)鉴定(MUMmer v3.1);直系同源基因与结构变异分析(SVs):同源基因鉴定:Inparanoid v4.1,GeMoMa;结构变异分析:BWA等;
棉花种质SNPs和InDels多态性:BWA,SAMtools,Picard,GATK4,Unified Genotyper,SnpEff v4.3T等。群体分化和单倍型多样性分析:FastTree,EIGENSOFT/SmartPCA,FST,π等;
遗传图谱构建:RILs,SOAP,HighMap;
1)基因组测序组装
本研究利用三代Pacbio SMART单分子荧光测序平台,对异源四倍体陆地棉TM-1 (~89×) 和ZM24 (~54×)进行测序,分别获得了~205 Gb和~125 Gb long reads,TM-1和ZM24共计获得 1,823和3,718 个contig。利用Illumina short reads纠错后,组装TM-1基因组2.286Gb(contig N50=4.760Mb);组装ZM24基因组2.309 Gb(contig N50=1.976 Mb),见表1;进而利用Hi-C将Congtigs组装到染色体水平,TM-1的挂载效率为97.4%(~2.23 Gb在染色体上定向:含At亚基因组上~1.41 Gb,458 contigs;含Dt亚基因组上~0.82 Gb,241 contigs),TM-1基因组组装结果相比前期研究中的组装结果有显著的提升:contig N50由34.0 Kb提升到了4,760 Kb;染色体挂载效率由先前的遗传图1.9 Gb (79.2%)挂载到染色体水平,到现在的2.23 Gb (97.4%) 挂载到染色体水平。通过与近期发表的陆地棉基因组进行比对,TM-1基因组连续性显著提升(2.52-fold against the G. hirsutum,HAU),本研究中的超长contigs使得本研究中的基因组组装更具准确性,通过两版TM-1参考序列的比对,发现除了D08染色体,其余染色体之间保持良好的共线性。Hi-C热图结果显示,在TM-1的D08染色体上的4个倒位区组装正确,又将TM-1 D08染色体分别与G.raimondii_D08,TM-1_ZJU_D08,Hai7124_ZJU_D08和TM-1_NAU_D08进行比较,均显示具有较强的共线性关系。
表1 异源四倍体陆地棉TM-1和ZM24基因组组装结果统计比较
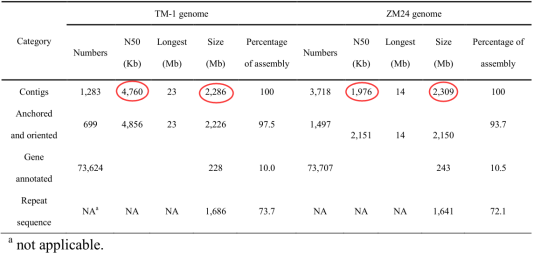
为了评估组装结果,将组装的两个基因组与前期已发表的遗传图谱进行比对,发现了每个基因组的每条染色体都有较高的共线性。此外,通过58个BAC文库测序分析,进一步证实了两个基因组组装的准确性和完整性。研究中又分别将Illumina short reads回比到TM-1 和ZM24基因组上,每个基因组大约99%的reads双端比对率。BUSCO评估,基因组完整性在99.5%以上,这一结果比最近公布的陆地棉参考基因组完整性评估98.2%还高。
重复元件(Repetitive Elements)广泛分布在大多数基因组中,并在基因组分化中发挥重要作用。通常,TM-1与ZM24的重复元件非常相似,在TM-1和ZM24中分别有约73.7% 和72.1%重复序列被注释到,这一比例低于亚洲棉(G. arboreum),但要高于雷蒙德氏棉(G. raimondii)。在这些序列中,反转录转座子占TM-1和ZM24组装序列的62.8%和61.5%, Long terminal repeats (LTR)占TM-1组装序列的~49.0%,包含Gypsy-type反转录转座子(41.7%),Copia-type反转录转座子(7.4%),ZM24的LTR组成类似于TM-1。Transposable Elements (TEs) 分析发现,亚洲棉(G. arboreum),TM-1 At 亚基因组,TM-1 Dt亚基因组,ZM24 At亚基因组和ZM24 Dt亚基因组可能非常活跃。此外,利用EMBOSS package(在亚洲棉A2,雷蒙德氏棉D5,陆地棉TM-1和ZM24 基因组上分别鉴定了33908, 6933, 41085和38,206高可信度的全长LTR)进一步估算LTR插入时间,在A2, TM-1和ZM24 At亚基因组中,LTR反转录转座子的爆发约发生在200万年前,在TM-1和ZM24 Dt亚基因组中的爆发时间大约在100万年前。基于A基因组及At亚基因组的TE分化及LTR插入时间,得出结论,大多数TEs在祖先基因组中扩增,然后在异源多倍体形成后保留。
2)四个基因组(陆地棉与其祖先种)之间的比较
已知异源多倍体中不同亚基因组的整合会导致基因组的变化,如在古多倍体玉米和异源多倍体小麦,芸苔属植物和棉花中。本研究中高质量的陆地棉基因组的组装将使得At和Dt与其各自的祖先种A2 (G. arboreum)和D5(G. raimondii)进行比较,以评估多倍化对棉花基因组重排的可能影响。At亚基因组和A2基因组整体的共线性及Dt亚基因组和D5基因组的共线性非常保守(图1 a和b),TM-1 At亚基因组的75.3%与A2基因组的72.1%共线性相关,同样的,TM-1 Dt亚基因组的大约78.1 %与 D5基因组的~85.6%共线性相关。
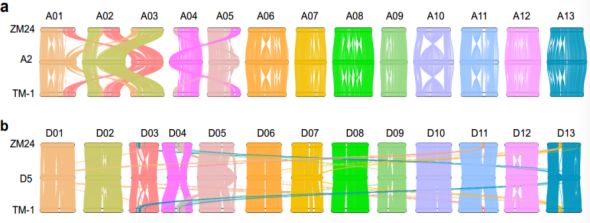
图1a. TM-1和ZM24的At亚基因组与亚洲棉G.arboreum基因组 (A2)共线性分析;b. TM-1和ZM24的Dt亚基因组与亚洲棉G. raimondii基因组 (D5)共线性分析
非共线性序列包含重复元件,特异性低拷贝序列及结构变异区域。TM-1 At和A2之间有13,819个重排(translocations和inversions),TM-1 Dt和D5之间有7,492个重排(translocations和inversions),TM-1和ZM24之间具有2,254个,这些结果清晰地表明,种间基因组重排的数量远远大于种内基因组重排。然而,TM-1 At/Dt和A2/D5染色体重排的总长度占~620 Mb,是TM-1和ZM24(occupying 51.2 Mb)的约12倍左右,进而鉴定了TM-1和ZM24亚基因组内部的倒位与易位。说明了A衍生的亚基因组在进化过程中明显比D-衍生的亚基因组更活跃。
在TM-1和ZM24中At和Dt亚基因上,发生在26号染色体上的重排和大规模的变异暗示了结构重排发生在多倍化后,但在两个陆地棉种质分化之前。通过At亚基因组和A2基因组的比较,鉴定了三个大的相互易位;此外,同过Hi-C证实了陆地棉A06染色体和亚洲棉 Chr06之间的到位(图2 c-e)。
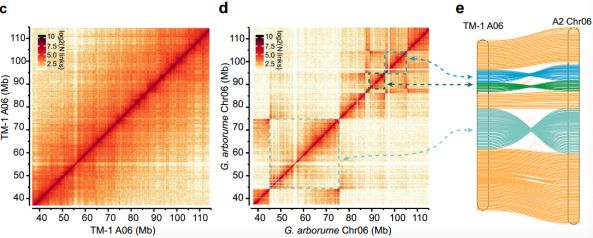
图2c-d TM-1 Hi-C数据比对到TM-1 A06染色体和G. arboreum Chr06染色体(A06和Chr06染色间发生倒位);e TM-1 A06和G. arboreumChr06染色体基因比较
3)TM-1和ZM24间基因组变异分析
ZM24 At亚基因组与TM-1进行比对,ZM24的99.3%基因组序列比对到了TM-1的 95.2%基因组序列;同样,ZM24 Dt亚基因组98.1%序列比对到了TM-1 Dt亚基因组94.3%序列,暗示了在陆地棉基因组中大部分区域是稳定的。在TM-1与ZM24之间,鉴定出了127个倒位,234个染色体内易位和1,893个染色体间易位,占~51.2 Mb。3个最大的结构变异来自于A08染色体,这些区域的总长度占TM-1 A08染色体总长度的 ~30%,占非共线性区域的~71.8 %。进而对TE转座子进行了相关分析。
PAVs(presence/absence variations)分析,共计鉴定了TM-1特异性PAVs 7,953个,ZM24特异性PAVs 13,160个。PAVs在染色体上的分布不均匀,如 ZM24 A08染色体 (1,847 PAVs, ~7.9 Mb) ,显著多于其它染色体(图3)。与TM-1 A08染色体相比,ZM24 A08染色体具有更多的PAVs,特别是在倒置区域,暗示了结构变异与PAVs相关。TM-1 Dt亚基因组与ZM24 Dt亚基因组上PAVs数量相似,分别为4,639和4,875,暗示了与Dt亚基因组亚基因相比,At亚基因组上具有更多的遗传变异。进一步将TM-1和ZM24特异基因比对到亚洲棉和雷蒙德氏棉染色体上,TM-1 At亚基因组和ZM24 At亚基因组上分别有大约58%和69%的PAVs基因与亚洲棉具有同源性,与此类似,在TM-1 Dt亚基因组和ZM24 Dt亚基因组上分别有大约73%和61%的PAVs基因与雷蒙德氏棉具有同源性,暗示了大部分PAVs基因存在于祖先基因组内,自二倍体棉花形成以来,二倍体棉花中没有明显直系同源PAV基因,可能在多倍体的形成过程中已经出现。
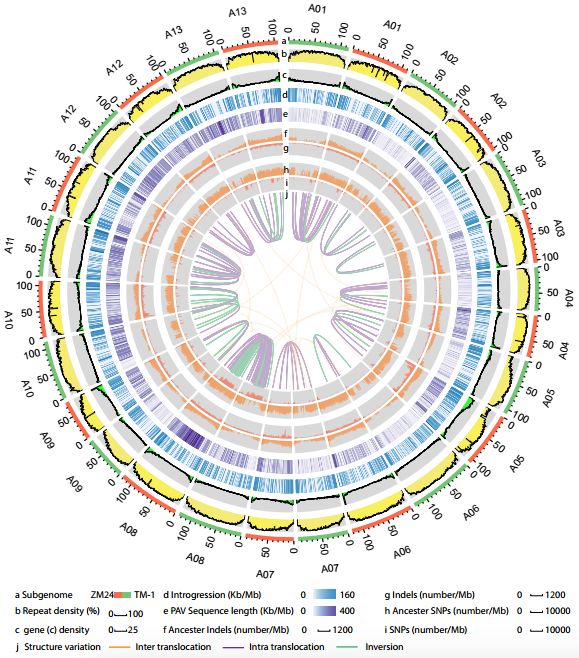
图3TM-1和ZM24基因组变异信息比较
基因组渗入研究,研究中共计鉴定了At亚基因组到Dt亚基因组上~8.3 Mb基因渗入区间,Dt亚基因组到At亚基因组上~7.8 Mb基因渗入区间,在 ZM24 At和Dt亚基因组共计有~8.8 Mb和~7.0 Mb的渗入片段,明显看出,在At到Dt亚基因组上的基因组渗入明显高于Dt到At亚基因组上的基因组渗入。
SNPs和InDels变异分析:在TM-1和ZM24的共线性区域,研究人员共计鉴定了907,682 SNPs和99,329 InDels,通过在全基因组范围内比较SNP密度(0.50 per Kb),A08染色体上SNP密度明显增加(图3),与SNP相同,A08染色体上的InDel密度明显高于其它染色体,并且在ZM24 A05 (0~7 Mb)上发现了一个插入热点区,该区域含有~7600 InDels (~1.08 per Kb),其InDels密度明显高于整体基因组的平均值 (0.049 per Kb),与之类似的是,在TM-1的A05染色体上也发现了1个InDel热点区域。
4)TM-1和ZM24之间的基因顺序和结构变异比较
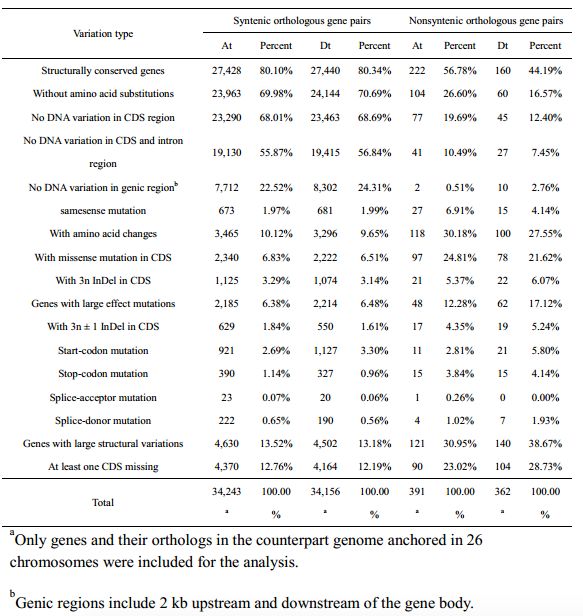
在TM-1和ZM24之间,鉴定了71,794个直系同源基因对,以分析基因顺序,在4个亚基因组上,58,913个直系同源基因对具有显著保守型,At和Dt亚基因组上分别具有5,570和5,400个,At亚基因组上34,634 (~96.1%) 直系同源基因对和Dt亚基因组上34,518 (~96.3%) 直系同源基因对锚定到26条染色体上,进一步用于基因组顺序和结构的比较,发现At和Dt亚基因组共线性区域共计有34,243和34,156个直系同源基因对,非共线性区域有391和362个直系同源基因对,分别占~1.1 %和~1.0 %。这些基因富含几种代谢途径,包括维生素B6代谢和糖胺聚糖降解。研究人员鉴定了TM-1和ZM24的At亚基因组和Dt亚基因组上共有3,465和3,296个直系同源基因对,仅在CDS/或非移码InDels区具有错义突变(表2),如玉米中所报道的,这些基因缺乏任何氨基酸的变化一起被归类为结构上保守的基因。研究人员分别对 ZM24和TM-1中共线性区域与非共线性区域中结构保守的基因进行了分析(表2),共鉴定了13,902个具有大效突变或大结构变异的直系同源基因对,在TM-1和ZM24之间大约有10%的注释到的直系同源基因对具有氨基酸变异,这些变化的生物学意义都需要进一步研究。研究人员进一步检验了TM-1和ZM24基因组中基因扩增含量,鉴定到了相应的单基因含量。
表2 TM-1和ZM24基因组间基因内的变异变异比较分析
虽然建立高再生频率的体细胞胚胎发生系统(SE)促进了转基因棉花的产生,但仍然存在棉花转化的重大挑战,包括基因型限制等,为了探究ZM24相对于TM-1易于转化的潜在机制,研究人员用SE期间取样的植物材料进行了mRNA-seq分析实验:差异表达基因(DEGs)在TM-1和ZM24愈伤组织(培养的第20天)和胚性愈伤组织(培养的第50天)之间,转录组分析结果显示虽然TM-1和ZM24愈伤组织之间的比较检测到几百个差异表达基因(培养第20天),但在ZM24胚性愈伤组织和第50天TM-1愈伤组织的比较中检测到一个数量级更多的差异表达基因,该结果与样本类型之间巨大的形态差异一致。鉴于先前报道的棉花和大豆中激素代谢基因具有高度影响的基因型特异性转化效率等相关作用,研究人员专注于具有注释到的此类差异表基因,鉴于其已知的生长素介导的调节对胚胎发生愈伤组织发育的贡献,值得注意的是,来自LEC1-like基因的6个基因座(Gh_D05G177600,Gh_A05G160900,Gh_A08G030500,Gh_D08G030600,Gh_D13G152700,Gh_A13G151400)的转录本在第50天愈伤组织中,ZM24中的表达水平显着高于TM-1。通过第20天与第50天之间的比较,两个生长素生物合成基因GhYUC10(Gh_D08G133100,Gh_A08G079800)在ZM24中同样发生了特异性表达,进一步暗示生长素积累和转运通过LEC1介导的生长素生物合成(GhYUC10)和生长素极性转运(GhPIN1)的调节,以促进棉花转化效率。这种假设可以通过在低转化效率栽培种的愈伤组织中过表达GhLEC1基因来测试,进而监测转化效率的变化,有或没有伴随的化学遗传学实验,直接改变生长素水平。
与生长素不同,增强的合成代谢促进胚性愈伤组织发育,已经表明赤霉素(GA)分解代谢促进体细胞胚胎发生。因此,研究人员在第50天愈伤组织中检测到来自编码已知GA-分解代谢调节转录因子GhAGL15(Gh_A12G116000,Gh_D12G115800,Gh_D08G112500,Gh_A08G163400)的同源物的四个基因座的转录物水平的ZM24特异性增加是一致的。似乎ZM24愈伤组织中的活性GA含量低于第50天难以转化的TM-1栽培种。这一假设同样可由GhAGL15或GhGA2ox基因的过表达实验证实。非常值得注意的是,研究人员检测到TM-1和ZM24品种之间的遗传多态性,许多遗传多态性来自激素代谢中的转化障碍相关基因:例如GhAGL15(Gh_A12G116000和Gh_A08G163400)基因编码区的SNPs以及GhAGL15(Gh_A12G116000和Gh_D08G112500)和GhLEC1(Gh_A13G151400)启动子中的SNP或InDel。
5)大的结构变异导致独立的单倍型
在组装的四倍体基因组的A08染色体上发现了三次大规模倒位(大于4 Mb);这些变异可通过Hi-C数据进一步证实(图4a),Hi-C热图的放大显示这些倒位的信号不连续性(图4b),通过比较基因组序列准确鉴定了SV1和SV3的左右断点,并用PacBio长读数据,PCR和Sanger测序进一步证实了这些断点。
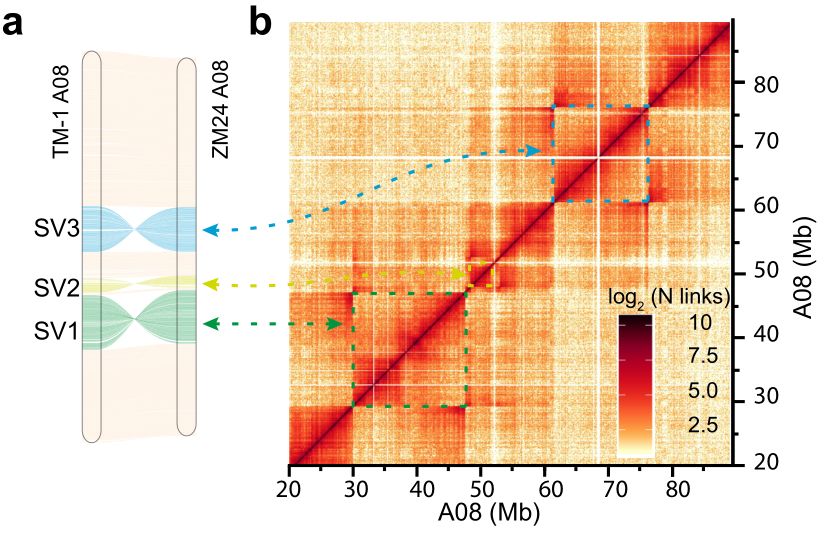
图4 TM-1和ZM24基因组A08染色体大规模染色体倒位(SV1,SV2和SV3)
已知结构变异区域中一个等位基因的选择压力可显着影响其整个倒置区域的等位基因频率和单倍型多样性,因此,研究人员利用全基因组重测序数据对全球搜集的419份种质进行了单倍型分析,具体来说,对于每个倒位断点,以确定mate-pair reads是否跨越类似于TM-1的断点,值得注意的是,SV1与SV3遗传连锁,大多数short reads同时支持特定陆地棉种质的共生的SV1和SV3;该分析鉴定了66份TM-1-like种质(不含SV1或SV3)和348份ZM24-like种质,具有这两种大插入变异(图5 c),研究中在跨越SV1和SV3的区域中鉴定了来自ZM24和TM-1的共226个和248个基因,因此表明结构变异区域中的严重基因缺失。令人兴奋的结果是,当利用这些种质的315,868个SNPs构建系统发育树时,该结果强烈支持这些倒位结构,进一步以区分这些种质多样性panel中有意义的单倍型(图5 d)。此外,主成分分析(PCA)分析也强烈支持A08单倍型的分类和系统发育分析的结果(图 5e)。值得注意的是,研究中发现TM-1-like基因型和ZM24-like基因型显示出显着的遗传分化(F ST),并且这种趋势在邻近断点的区域也是明显的(图5f)。ZM24-like基因型的单倍型多样性在倒置区域大大减少,并且在断点临近的区域中效果也很明显(图5g)。
接下来,考虑已知减数分裂重组倒置基因组区域中被显着抑制的事实,此外,考虑到已知抑制对两个倒置等位基因之间的遗传交换具有最小化影响,尽管已知这些区域具有强烈的遗传影响,但并未发现任何关于陆地棉中这种倒位的大规模分析的报道,这可能是由于缺乏高质量的基因组组装结果以便于识别断点。为了进一步探索这些想法,研究中通过TM-1和ZM24杂交,产生了181个RILs的群体,以用于计算重组率。当将TM-1和ZM24之间的 SV1 和SV3区的重组数据重叠时,观察到抑制的重组率与倒位区域内的基因座之间存在很强的相关性,且这种受抑制的重组趋势在断点附近的区域也很明显(图5h)。位于倒位区域外但在染色体断点区域附近的多态性相对于整个基因组倾向于具有减少的遗传交换,这种情况类似于倒位中发现的多态性。为了进一步研究倒位和遗传多样性之间的关系,研究人员在ZM24-like和TM-1-like基因型中统计了A08染色体上的SNPs,发现SNPs密度在倒置区域内显着降低。
进而进行了核苷酸多态性(π)分析,同样也揭示了TM-1-like基因型和ZM24-like基因型倒位区域内的遗传多样性显著降低,在断点附近的区域,核苷酸多样性减少的趋势也很明显。综合上述分析结果显示,重组率和单倍型结果共同表明由于A08染色体上的倒位区域导致的重组(以及因此引起的遗传交换)抑制随着时间的推移将陆地棉种群分成两个不同的群体。因此有必要对陆地棉分化如何发生以及该过程如何特别影响陆地棉种群中现存的核苷酸和性状多样性的理解。
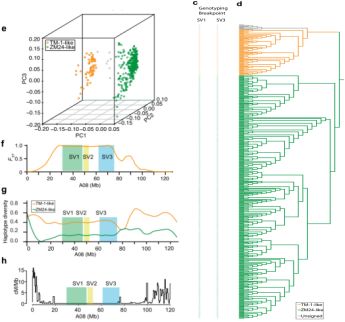
图5 c SV1和SV3中断点的基因分型;d 基于SNPs的单倍型聚类分析;e 421份种质的PCA主成分分析;f TM-1-like群体和ZM24-like群体的遗传分化分析;g TM-1-like群体和 ZM24-like群体的单倍型多样性分析;h 大的倒位对源自TM-1和ZM24杂交的RIL群体减数分裂重组率的局部影响;
总结
1. 本研究利用纯三代(PacBio)+Hi-C研究技术,成功组装了两种陆地棉重要种(遗传育种品种TM-1和转基因品种ZM24)基因组):组装TM-1基因组2.286Gb(contig N50=4.760Mb),ZM24基因组2.309 Gb(contig N50=1.976 Mb);
2. 以高质量的陆地棉TM-1和ZM24基因组为参考,与其祖先种亚洲棉A2 (G. arboreum)和雷蒙德氏棉D5 (G. raimondii)进行比较,进行共线性及染色体重排分析,证明了陆地棉A06染色体和亚洲棉 Chr06之间的倒位;
3. 陆地棉TM-1和ZM24间基因组变异比较分析,如SNPs,InDels,PAVs和SVs等,推测了大部分PAVs基因存在于祖先基因组内,并可能在多倍体的形成过程中同源PAV基因已经出现。
4. 在组装的四倍体基因组的A08染色体上发现了三次大规模反转(大于4 Mb),并利用Hi-C数据进一步证实,并利用两个栽培种TM-1,ZM24和种质panel的作图群体RIL的单倍型分析显示该区域的重组率受到抑制。
- 本文固定链接: https://www.maimengkong.com/zixun/1465.html
- 转载请注明: : 萌小白 2023年4月21日 于 卖萌控的博客 发表
- 百度已收录
