
基于RNA依赖性RNA聚合酶在宏基因组数据中分类RNA病毒
RdRp-based sensitive taxonomic classification of RNA viruses for metagenomic data
作者:汤旭博,商家煜,孙燕妮*(通讯作者)
摘要
随着第二代测序技术的进步,病毒宏基因组测序已经成为发现新型病毒的主要来源。对宏基因组数据中的读数进行生物学分类,已经成为确定样本中病毒成分的重要手段。然而,RNA病毒种类丰富且高度多样化,导致基于比对的方法灵敏度降低。为了提高在读数上分类的灵敏度,作者开发了一个基于RNA依赖性RNA聚合酶(RdRp)基因的读数分类工具--- RdRpBin。它利用RdRp的序列特性,将序列比对与机器学习模型相结合,对宏基因组中的RNA病毒读数进行分类。在模拟和真实测序数据上,特别是当测试读数与数据库相似度较低(大约0.4)时,RdRpBin都取得了比其他工具更好的结果。
方法
作者分析了NCBI上的RdRp序列,发现RdRp有以下三个特性:
1. 来自同一生物分类群的RdRp并不总是表现出高度的相似性。
2. RdRp序列上不同区域的保守性是不同的。
3. 来自不同分类群中的RdRp的数量差异很大。
基于上述提到的RdRp的特性,RdRpBin在分类读数时分为两步(如图1所示)。第一步是通过计算序列相似度将高相似性的读数与数据库的序列建图,图上的点是序列,边由序列相似度定义,然后利用 概率关系分类器(Probabilistic Relational Neighbor Classifier, PRC)对图上的读数分类。第二步是针对第一步中无法分类的读数,在第一步的基础上利用序列提取的motif,在共享相同motif的序列之间建边,然后通过 图卷积神经网络(Graph Neural Network, GCN)进行分类。
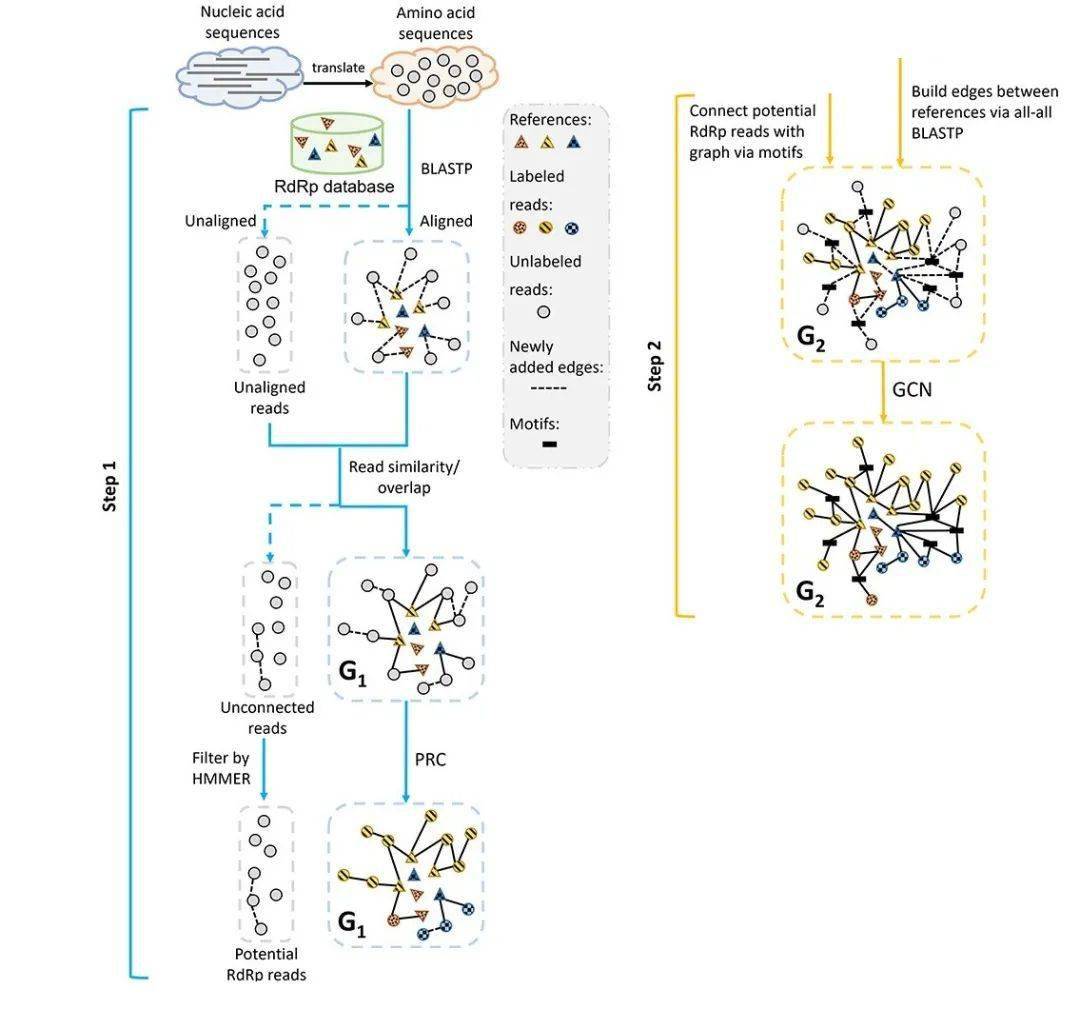
实验结果
1.在模拟RNA病毒测序数据上的表现
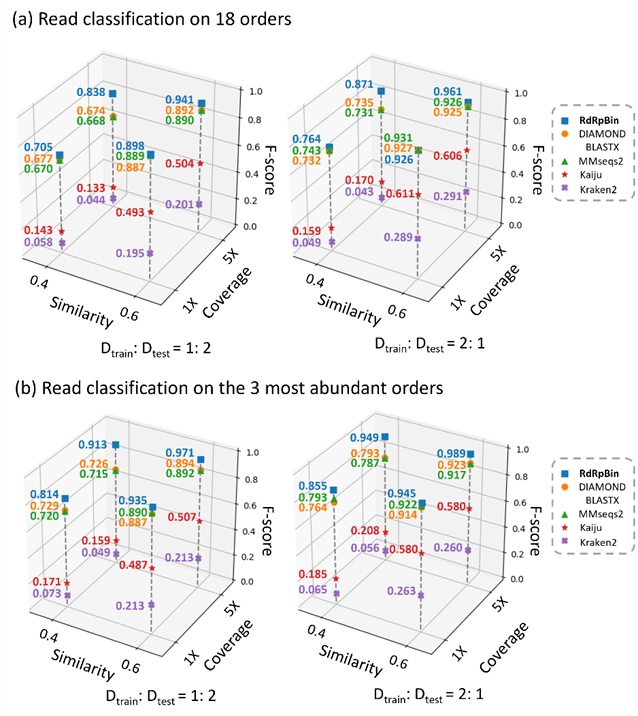
图2. 模拟的RNA病毒测序数据上的表现
结果表明,相似性对分类性能起着重要作用。依靠精确匹配的工具,包括Kaiju和Kraken2,当相似度从0.6下降到0.4时,其平均f-score分别减少了72.7%和80.1%。 相比之下,当相似度为0.4时,RdRpBin的f-score仍然很高。尽管所有工具的F-score都随着参考数据库的变小而下降, 但RdRpBin在小的数据库上也有最好的表现。当覆盖率增加时,RdRpBin的平均F-score要比其他工具高得多。
2.在模拟海洋病毒宏基因组数据上的表现
为了在更现实的情况下测试RdRpBin的表现,考虑到真实的病毒宏基因组数据仍然受到来自真核生物和原核生物等的污染,作者创建了一个模拟的海洋宏基因组数据集,其中混合了来自RNA病毒和其他海洋物种的读数。为了更好的评估工具的表现,作者还删除了数据库中与测试序列相似度超过40%的读数。测试的表现如图3所示。
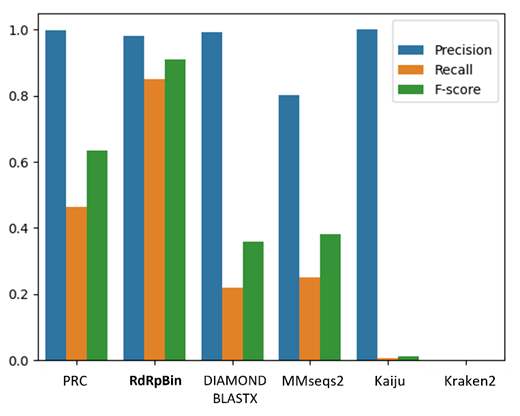
图3. 模拟的海洋病毒宏基因组数据上的表现
结果表明,Kaiju和Kraken的表现并不理想。虽然Kaiju的精度为1,但其召回率只有0.007左右。与DIAMOND BLASTX相比,使用PRC可以将召回率提高到0.464,而精度保持在0.997,而 RdRpBin可以进一步提高召回率至0.849。
3.在真实宏基因组数据上的表现
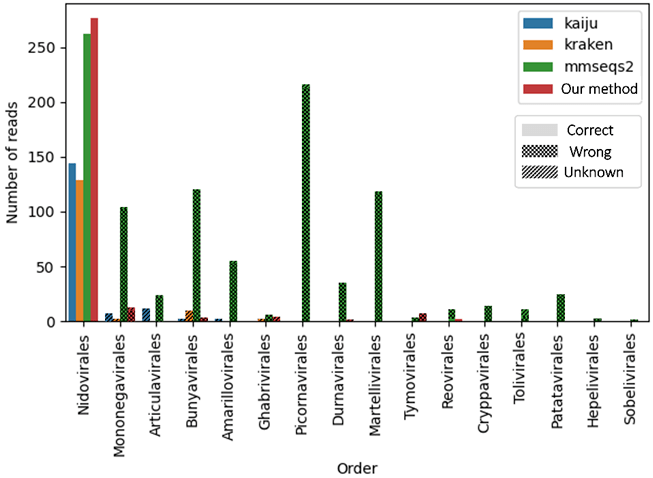
图4. 真实宏基因组数据上的表现
结论
RNA病毒具有高度的多样性,而RdRp是大多数RNA病毒所拥有的唯一基因。通过采用 RdRp和基于图的分类方法,RdRpBin 实现了RNA病毒读分类的召回率和精确度之间的权衡,这有助于更准确的从宏基因组数据中评估RNA病毒组成以及丰度的概况。特别是 当测试的RNA病毒与参考数据库的相似度较低时(如0.4),RdRpBin仍能保持较高的F分数(>0.8)。
作者介绍
汤旭博,目前在香港城市大学攻读博士学位。他的主要研究兴趣包括计算生物学和生物信息学,特别是应用 深度学习模型来分析基因组数据。
商家煜,在中山大学获得学士学位。他现在正在香港城市大学攻读博士学位。他的研究兴趣是生物信息学,重点是 分析微生物测序数据的算法设计。
通讯作者:孙燕妮,香港城市大学电机工程系(Electrical Engineering)副教授,博士生导师。在美国圣路易斯的华盛顿大学(Washington University in Saint Louis)取得计算机系博士学位后在密西根州立大学计算机系担任助理教授和副教授(with tenure)。2018年加入香港城市大学。主要研究方向是 生物信息学,序列分析,宏基因组学,和病毒基因组学。具体的研究课题,发表的论文,以及实验室的位置请参加作者个人主页:https://yannisun.github.io/
- 本文固定链接: https://maimengkong.com/morejc/1127.html
- 转载请注明: : 萌小白 2022年7月21日 于 卖萌控的博客 发表
- 百度已收录

