一、软件下载以及安装
1.进入NCBI官网
https://www.ncbi.nlm.nih.gov/
2.点击All Resources
3.Downloads
4.BLAST (Stand-alone)
ftp://ftp.ncbi.nlm.nih.gov/blast/executables/blast+/LATEST/
5.根据电脑系统下载相应的安装包(*.exe)
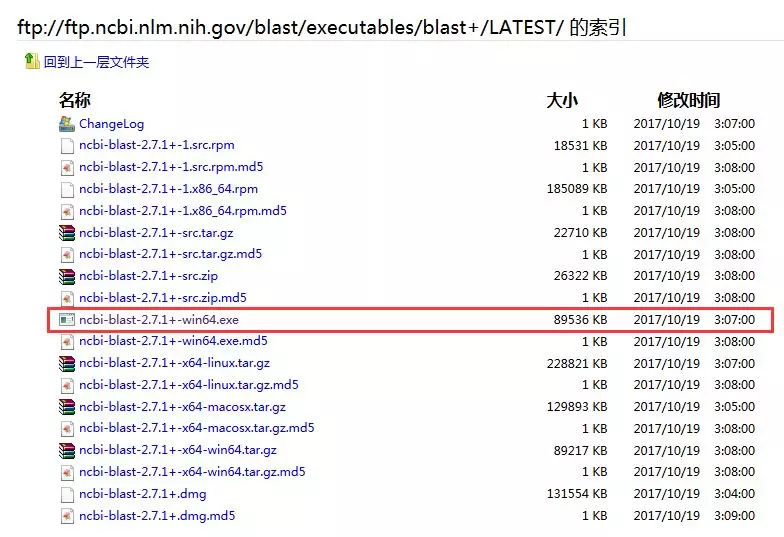
6.双击安装,默认即可
二、软件运行
1.点击电脑的“开始”,输入“cmd”,回车键,调取DOS命令。
2.切换工作目录到blast的bin下,DOS命令是:cd C:\Program Files\NCBI\blast-2.7.1+\bin(实际安装目录),回车。(>之前的路径为当前工作目录)这里需要说明一下,切换到bin目录下方便新手操作,熟练之后可以自行创建工作目录。
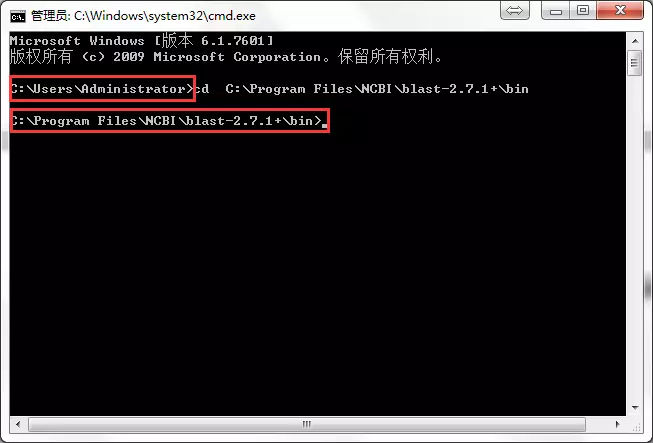
3.准备好需要比对的两个序列文件(一个是需要比对的序列,一个是物种序列,格式为fasta/fa),放入到上一步的bin目录下。
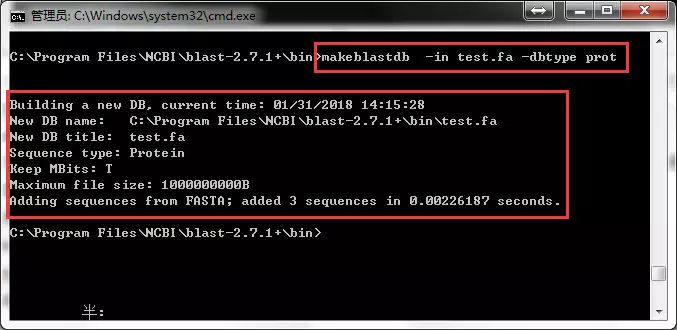
4.建库
对物种序列进行建库处理,转换成blast软件可用的格式(可以不是物种序列,只要是一条或者一组序列即可)
DOS命令:makeblastdb -in input_file -dbtype nucl/prot -out database_name
Input_file:建库的序列文件名,比如拟南芥蛋白序列
nucl/prot:建库的序列属性,核苷酸库/蛋白质库
database_name:自定义库名字,如果不填写参数,则默认是文件名
其它详细参数可以通过DOS命令makeblastdb -help进行查看
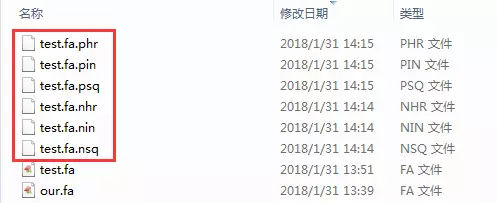
生成的结果文件(可多次使用):
5.比对
DOS命令:blastn/blastp/blastx/tblastn/tblastx -query text_query.txt -db refseq_rna.00 -out output.txt -evalue 10 -outfmt 0-18
Blastn:核苷酸与核苷酸库比对(核苷酸层面)
Blastx:核苷酸与蛋白质库比对(核苷酸翻译成蛋白质,在蛋白层面进行比对)
Blastp:蛋白质与蛋白质库比对
Tblastn:蛋白质与核苷酸库比对(核苷酸库翻译成蛋白质再进行比对)
Tblastx:核苷酸与核苷酸库比对(蛋白质层面)
text_query.txt:需要比对的序列文件
refseq_rna.00:建库文件
Output.txt:比对结果输出文件
10:比对期望值,软件默认为10,一般大家会设置为1e-5
0-18:输出文件格式,0-18代表不同的文件格式,常用6,7,10
其它详细参数可以通过DOS命令blastn -help进行查看
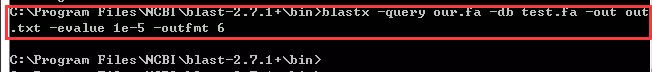
结果文件:out.txt(bin目录下)

输出的结果文件是没有表头的,这里小编已为各位整理好~
拿走,不谢!
☼
相关测试数据和软件包见链接:
https://pan.baidu.com/s/1bqejWOR
更深入的使用介绍见官网:
https://www.ncbi.nlm.nih.gov/books/NBK52637/- 本文固定链接: https://www.maimengkong.com/morejc/1126.html
- 转载请注明: : 萌小白 2022年7月19日 于 卖萌控的博客 发表
- 百度已收录
