近几十年,系统生物学慢慢走近科研者的视野,主要着眼于研究细胞信号转导、基因调控网络、生物系统组成。多组学联合分析可以由表及里,解析直接原因和根本原因,深入阐释作用机理。在多组学的联合应用中,最终都要落脚到功能注释和通路富集分析,其中基于通路富集分析的图形展示丰富多样,通过此分析可找到与研究相关的通路、关键基因/代谢物,以便进行后续的数据挖掘。
进行富集分析的软件有很多,本篇主要安利一下clusterProfiler,它是一个专门用于GO、KEGG以及GSEA富集分析的R包,我们以代谢组数据分析为例,进行KEGG通路富集分析。
安装:
BiocManager::install(‘clusterProfiler’)
加载:
library(clusterProfiler)
计算富集所用的函数为enricher,其基本的参数:(1)gene是指用于富集的差异代谢物ID;(2)TERM2GENE是一个2列的数据框,第一列为通路ID如ko01230,第二列为注释到该通路代谢物ID,这个数据是背景集即包含所有通路注释的代谢物(3)TERM2NAME也是一个2列的数据框,第一列为通路ID,第二列是通路的具体名称。其他参数可参考官方文档(https://bioconductor.org/packages/release/bioc/manuals/clusterProfiler/man/clusterProfiler.pdf)。
查看输入
head(TERM2meta)
# KEGG_ID metabolite_ID
#1 ko01230 meta4
#2 ko01230 meta20
#3 ko01230 meta23
head(TERM2NAME)
# KEEG_ID KEGG_name
#1 ko00010 Glycolysis / Gluconeogenesis
#2 ko00020 Citrate cycle (TCA cycle)
head(metaids)
#[1] "meta10" "meta1017" "meta1020" "meta1024" "meta1031" "meta1034"
富集分析
#为了展示所有结果,将p值设为1
enrich<-enricher(gene=metaids,TERM2GENE=TERM2meta,TERM2NAME=TERM2NAME,pvalueCutoff= 1,qvalueCutoff = 1)
#查看富集结果
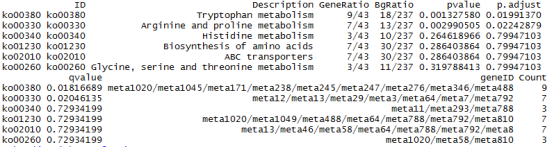
由于该软件原本是用于基因分析的,所以部分列名显示的是Gene
head(enrich@result)
接下来,咱们就可以作图啦~

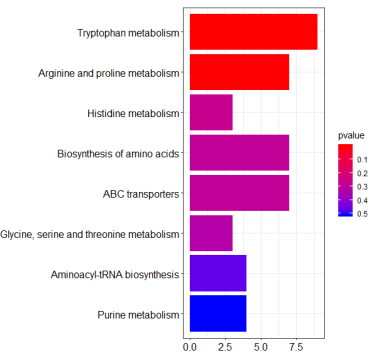
绘制柱图
barplot(enrich,color='pvalue',bg=NULL,grid=NULL)
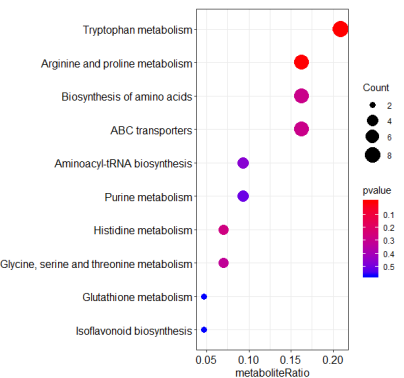
绘制点图
为了修改横轴名称,加载ggplot2
library(ggplot2)
dotplot(enrich,color='pvalue',)+xlab('metaboliteRatio')
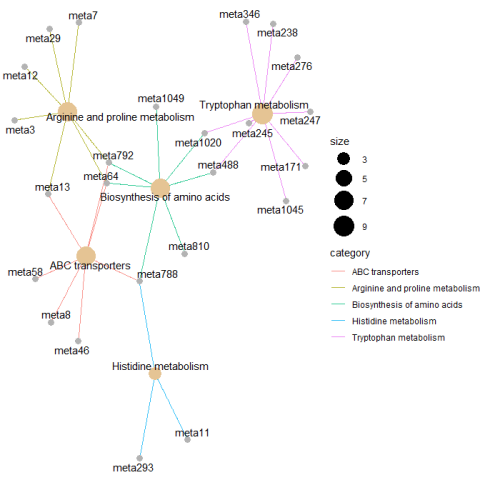
绘制网络图
cnetplot(enrich,vertex.label.cex=0.1,colorEdge = TRUE)


富集分析是多组学联合分析的基础,基于此百迈客可以实现三个组学之间的联合分析,优质美图一键绘出。
为了缩短分析周期,提升个性化挖掘空间,百迈客云提供专业的生信分析平台(http://www.biocloud.net/),最新上线的代谢组云APP兼具标准分析、联合分析和个性化分析功能,同时提供海量在线资源和视频,欢迎一起开启代谢组准确检测,深入挖掘新时代!
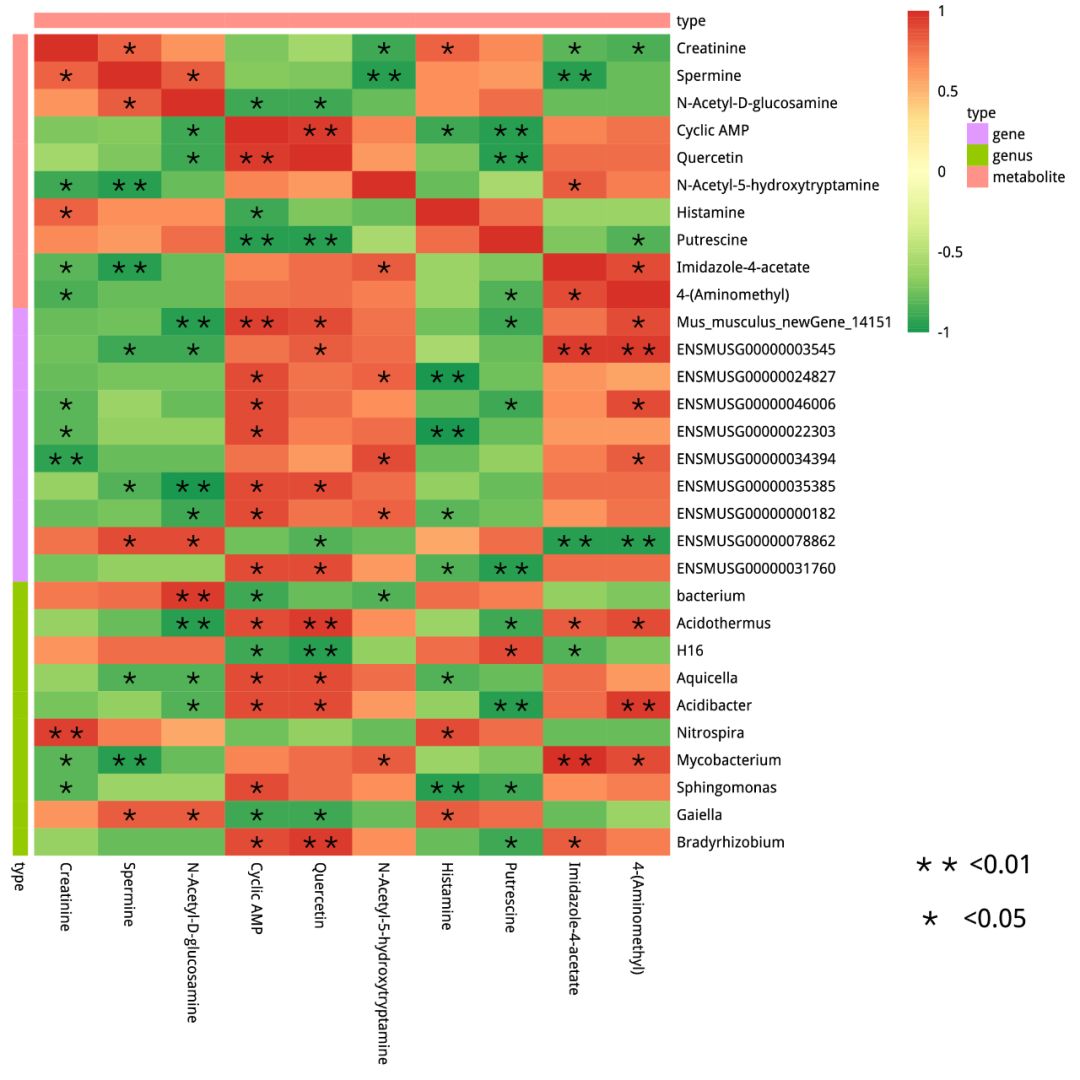
代谢物、基因、菌群之间的相关性热图
*为P<0.05,**为P<0.01
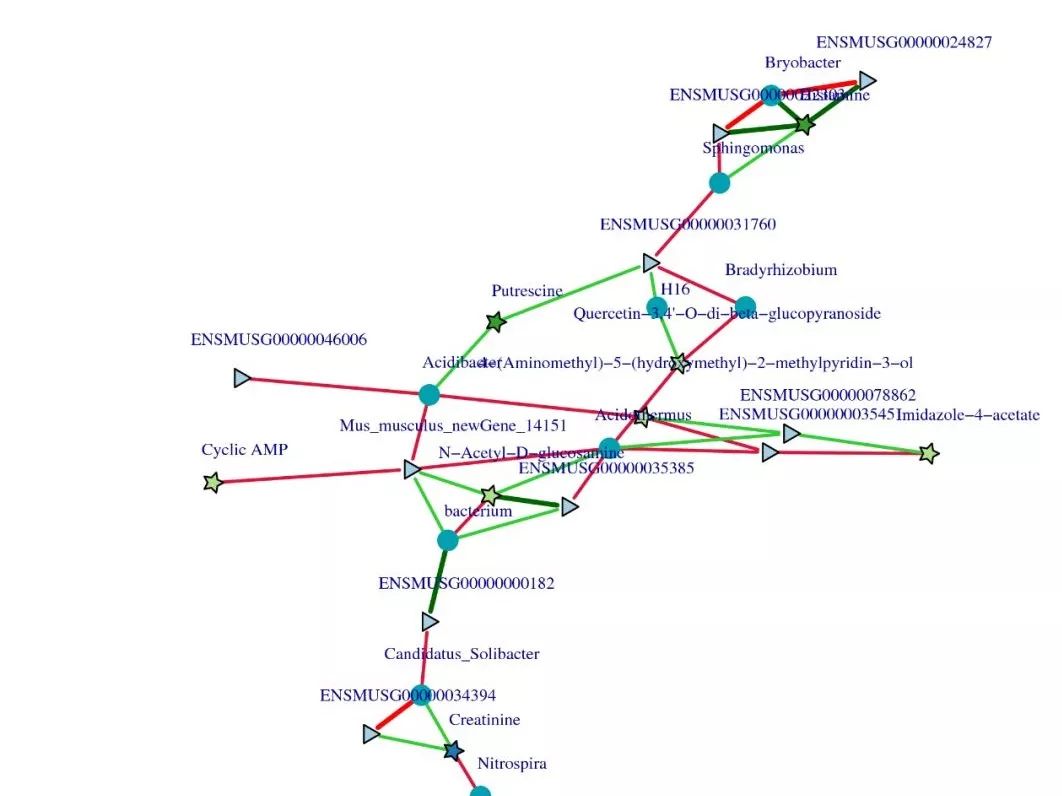
代谢物、基因、菌群之间相关性网络图
线颜色表示正负相关,线粗细表示相关性强度

代谢物、基因、菌群之间CCA图
基因、菌群与原点的连线和代谢物构成的夹角表示了之间相关性
转自:百迈克
- 本文固定链接: https://www.maimengkong.com/image/1015.html
- 转载请注明: : 萌小白 2022年6月23日 于 卖萌控的博客 发表
- 百度已收录
