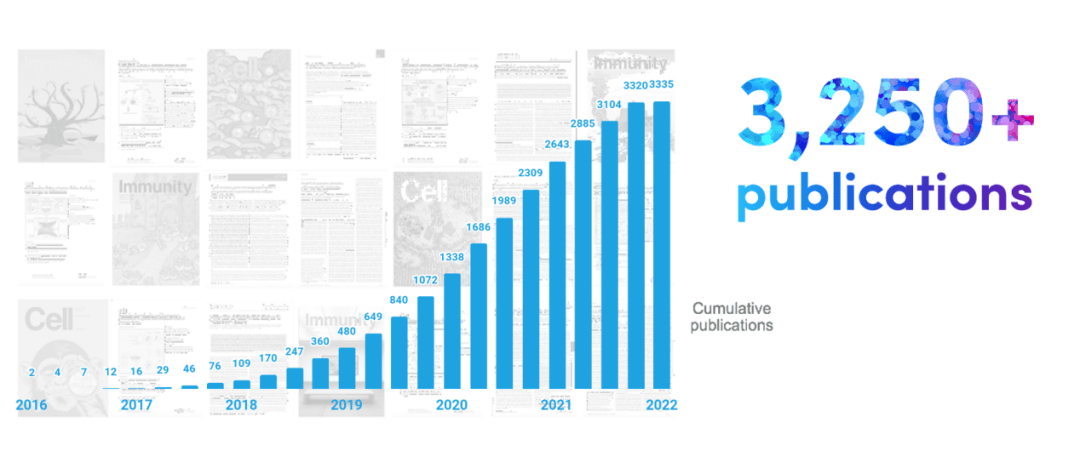
众所周知,单细胞测序已然是发表高分文章的利器,几乎每天都有不止一篇单细胞文章发表。目前,单细胞技术已经广泛应用于肿瘤,免疫,发育生物学,神经科学,细胞治疗,药物研发及动植物研究中。看着别人的单细胞文章发发发不停,不知您是否也心痒痒?其实要想开展单细胞研究并不难,今天小编就先带您了解一下最常见的单细胞转录组测序的基本流程,希望您也能尽快的开展起单细胞实验哦!
送样要求
迄今为止,研究的样本类型已有来自肿瘤,干细胞,胚胎,神经系统等多个不同部位,理论上真核生物都能进行10x单细胞RNA测序。在样本选择好之后,实验结果是否理想,很大程度取决于样本的质量,下面是样本制备与运输一个详细的介绍:
如果样本为细胞悬液,需满足下面条件:
1. 细胞直径小于40um(直径超过40um细胞需要制备成细胞核);
2. 细胞活率大于80%(用台盼蓝或者AO/PI染色检测,官方建议,当单细胞悬液活性低于 70% 时应进行去除死细胞操作);
3. 单细胞总数大于3万;
4. 细胞成团比例小于10%;
5. 无细胞碎片或其他颗粒杂质;
6. 红细胞比例小于30%;
7. 建议细胞悬液的浓度为700~1200个细胞/ul,体积不小于50ul;
8. 细胞悬液运输,根据细胞类型,一般冰上运输,1h内到达实验室,检测细胞质量,合格后上机;如果是中性粒细胞,常温运输,越快越好。
注意:
1. 重悬单细胞使用的Buffer不能含有钙镁离子和EDTA;
2. 为了保护细胞的活性可以添加BSA和FBS;
3. 常用的Buffer:PBS+0.1%BSA(容易成团细胞推荐);DMEM+10%FBS(容易死的细胞推荐)。
对于组织样本,需进行以下处理:
1.手术样本取出后,用1xPBS清洗表面血水,修理组织,剪去脂肪,纤维,坏死的组织;
2.建议一次消化的量为黄豆粒大小(100-200mg左右),如果组织偏大需指定区域,否则任意剪取;
3.组织浸没在组织保存液中,4℃保存,冰上运输不能冻。
4.组织离体后48小时内需进行组织解离,尽量在24小时内送至实验室,然后实验室将尽快处理。
实验流程
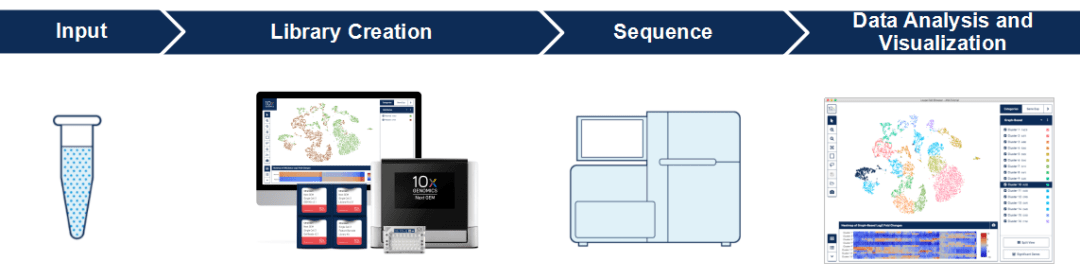
10x Genomic配套有相应的仪器与分析软件,首先将制备好的单细胞悬液上样到10x Genomic 单细胞捕获系统中,构建带有10x标签的cDNA文库,并在Illumina测序仪上进行短读长NGS测序,得到的数据经Cell Ranger可获得单个细胞水平的基因表达谱和差异分析情况。通过Loupe Browser将数据可视化,使目的基因的表达模式,细胞类型,细胞簇和样本之间比较分析得以直观地展现。
技术原理
Gel Beads(凝胶微珠)
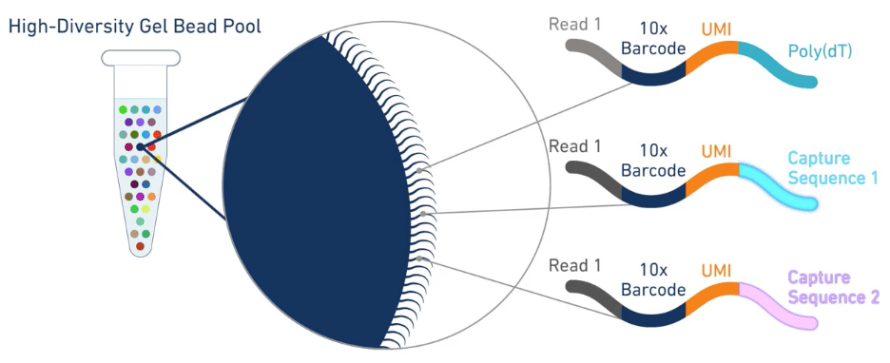
Gel beads在转录组测序中的作用是捕获细胞中的mRNA,每个微珠表面携带有几十万个Oligo序列,具体结构如上图所示:
Oligo dT序列(图中第一条)由4个部分组成:read1用于上机测序;第二段是16bp 的10x barcode序列,每一个Gel Beads都携带不同的barcode , 可标记获取的mRNA都来自于哪一个细胞;第三段UMI长度为12bp, 可以将转录本序列进行绝对定量,避免扩增产生偏好;第四段是30bp的ployT,用于捕获有ployA尾的转录本。
捕获和建库
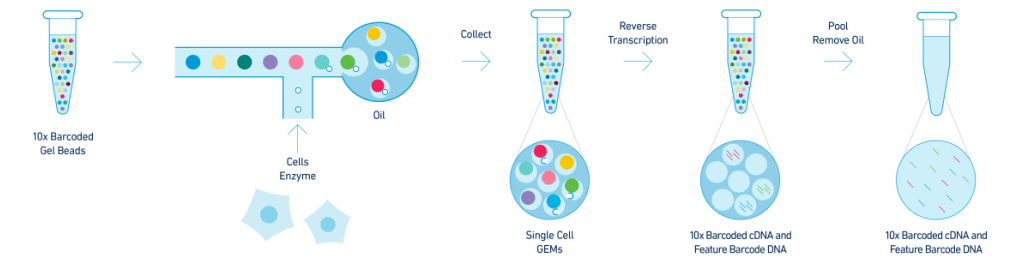
第一步对制备好的单细胞悬液先进行质检,质检合格后与Gel Beads和油分别加入到Chromium Chip G的不同通道,如上图所示,经由微流体“T字”交叉系统形成GEM(油包水结构体系)。一个通道产生8-10万个油滴,其中10%的油滴中含有细胞和Gel Beads。接着细胞裂解,Gel Beads自动溶解释放大量引物序列,与带有PolyA的mRNA进行逆转录,生成带有10x Barcode和UMI信息的cDNA第一链。GEM破碎后,磁珠纯化一链cDNA,并进行PCR扩增,得到稳定的cDNA。然后将扩增后的cDNA进行酶切片段化,筛选合适长度片段,通过末端修复、加A、接头连接Read2测序引物,构建含有P5和P7接头以及双端index的3端表达谱文库(见下图)。
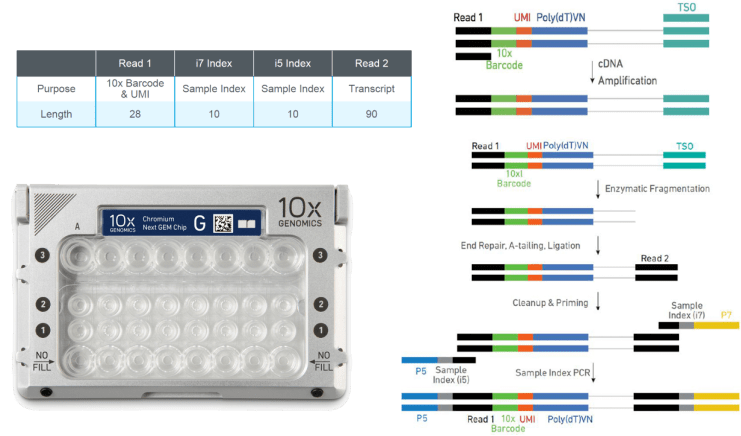 Chromium Chip G(左下)和3‘端建库(右)
Chromium Chip G(左下)和3‘端建库(右)
测序深度选择
接下来大家会好奇,测序深度为多少合适呢?这个需要权衡需求和成本:对于低频稀有细胞类型,增加测序细胞数,保持低深度测序;对特定细胞,提高测序深度。
一般来说,首次测序量建议25k reads/cell (例:预估捕获10000个细胞,10000*25000=250M reads)。测序结束后,先Cell Ranger对数据进行初步分析,然后根据 Sequencing Saturation的数值判断是否需要加测,以及加测量的多少。
数据分析
分析过程
经过上机测序,拿到原始数据后,会先用Cell Ranger提取细胞的barcode和UMI ,通过STAR 比对到参考基因组,校正barcode ,过滤校正UMI并进行计数,接着根据算法区分包含细胞的 barcode 和背景 barcode,以便提取出真实的单细胞数据,最终得到各细胞的基因表达矩阵。后续用 Seurat做进一步的细胞过滤和标准化,通过降维(PCA)进行聚类(K-Means)、可视化(t-SNE)以及UMAP降维,得到常见的细胞聚类分型结果、Marker 基因及差异表达基因信息。
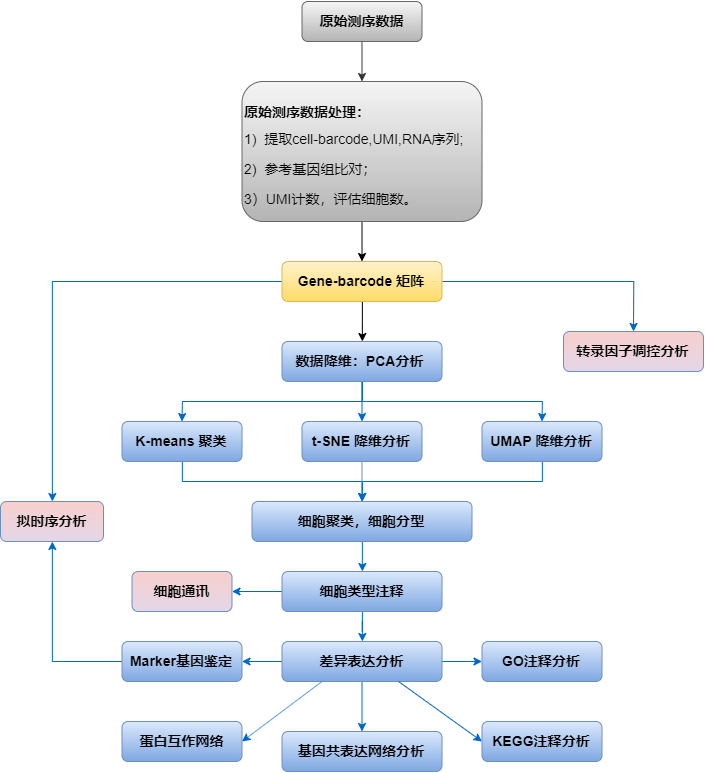
分析内容
t-SNE/UMAP图:标准化过滤后的数据,使用降维(PCA)和聚类进行细胞分型,根据聚类结果,采用t-SNE/UMAP降维算法,将细胞的分布进行展示,并使用Single Cell Identificator Based on E-test (SciBet)和参考数据集对细胞类型进行注释(如下图)。
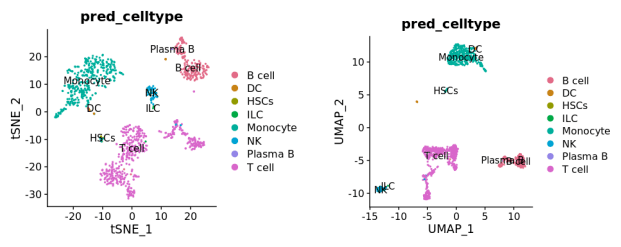 t-SNE和UMAP图
t-SNE和UMAP图
Violin图和热:差异基因分析后进行展示。
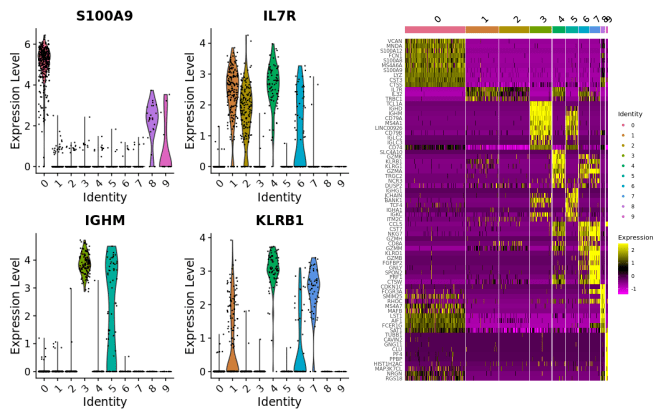 左:小提琴图展示高表达基因在不同cluster中的表达情况 右: 每个cluster鉴定的top 差异基因热图
左:小提琴图展示高表达基因在不同cluster中的表达情况 右: 每个cluster鉴定的top 差异基因热图
个性化分析:
KEGG/GO 富集分析:通过不同聚类方式对每个cluster差异高表达基因进行富集分析,寻找每个cluster差异高表达基因都与哪些生物学功能或通路(KEGG pathway)显著相关。
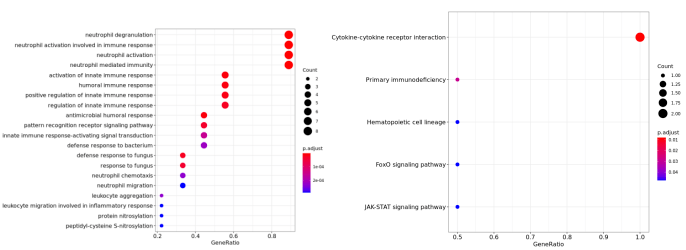 KEGG/GO分析图
KEGG/GO分析图
我们还能提供:蛋白质互作网络分析、基因网络共表达分析(WGCNA相比于差异表达基因可以获得跟多信息)、拟时序分析(用于观察细胞类型的初始-发育的变化状态)、细胞通讯信息(推测细胞互作关系)、转录因子调控分析以及定制服务,满足多种需求。
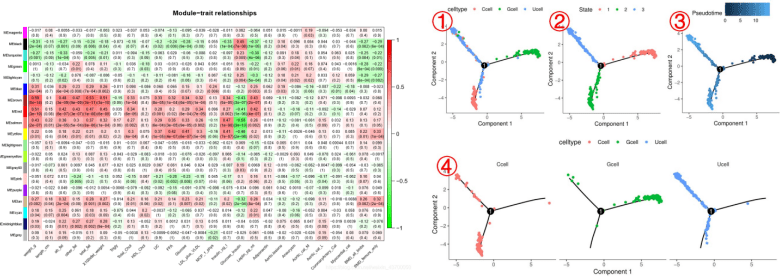 左:模块与性状的相关性热图 右:拟时序分析图
左:模块与性状的相关性热图 右:拟时序分析图
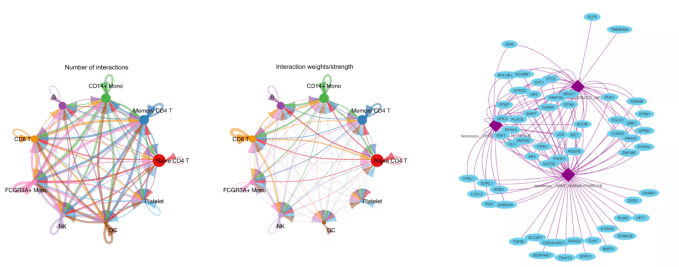 左一、二:细胞通讯图 右一:转录因子调控分析图
左一、二:细胞通讯图 右一:转录因子调控分析图
到这里,相信您已经对10x Genomics单细胞转录组测序有了基础的了解,云准科技可为您提供一站式单细胞多组学优质的服务,如您想了解更多关于10x单细胞转录组的信息,欢迎留言或来电咨询。

转自“云准科技”
- 本文固定链接: https://www.maimengkong.com/zu/1523.html
- 转载请注明: : 萌小白 2023年5月12日 于 卖萌控的博客 发表
- 百度已收录
