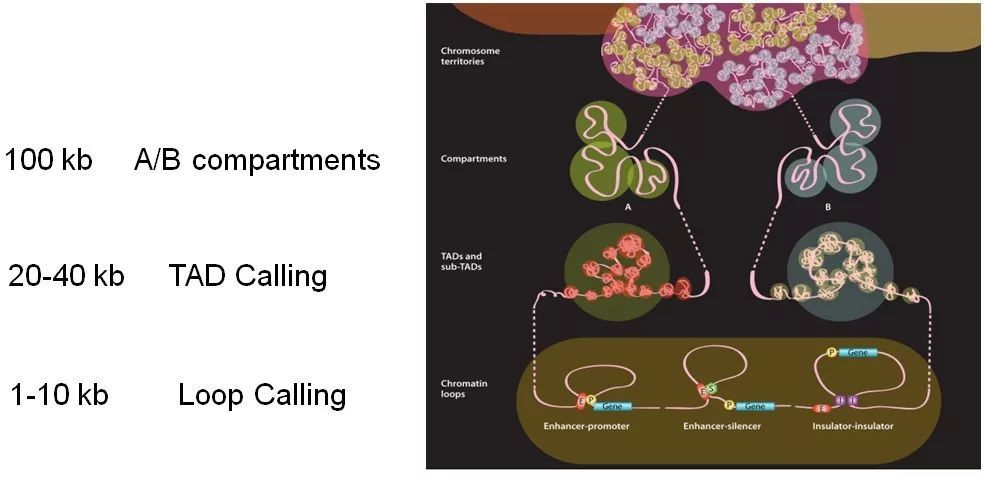
随着研究的深入,越来越多的研究者希望通过Hi-C技术去分析DNA loop结构。在染色体的折叠过程中,DNA loop环的形成让线性距离上相隔很远的位点有机会靠近并发生互作,这些loop的类型有很多种,包括enhancer–promoter loop, Polycomb-mediated loop, gene loop, architectural loop等(图1),准确的call loop是远距离互作分析的关键,对Hi-C分析云里雾里的你,是否和小编有过同样的困惑:测多少数据、分析到多深的分辨率才能有效的获得loop信息呢?
其实,我们一直有一套科学的数据量推荐方法,今天小编就从已发表文献的数据整理说起,总结Hi-C分析,特别是高分辨率下的loop分析对测序数据量的真实需求,另有安诺Hi-C实测大数据的首次展示,掰开了揉碎了给大家说道说道我们Hi-C数据量推荐的依据和实战经验~
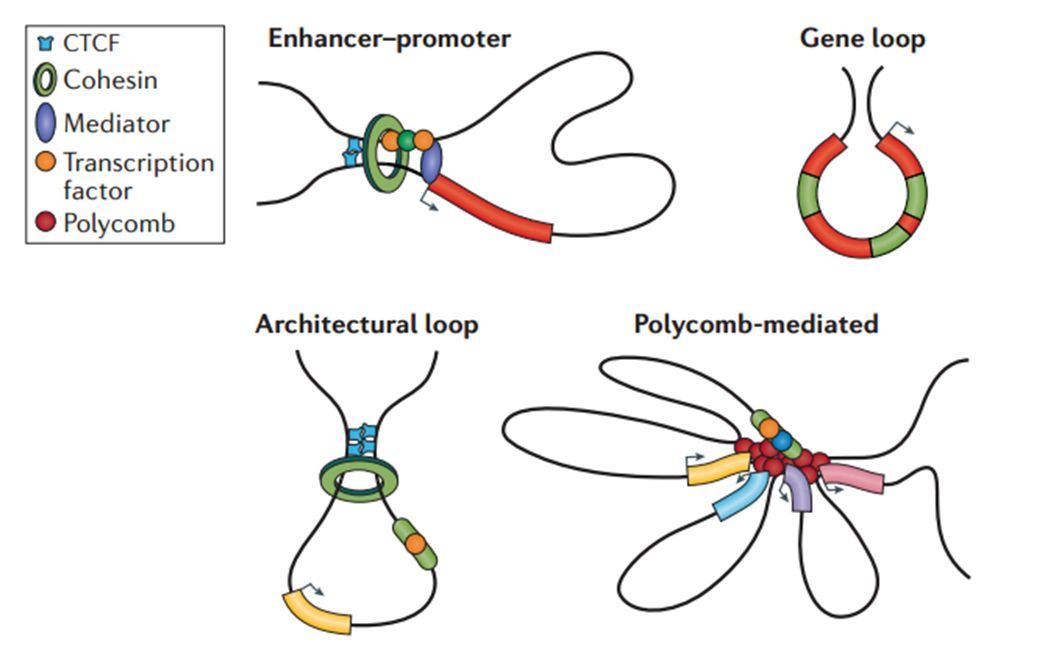
图1 不同类型的染色质loop结构
(enhancer–promoter loop, Polycomb-mediated loop, gene loop, architectural loop)
发表文章数据统计
首先,我们看一下Cell期刊上的两篇经典文献对于数据量的要求~
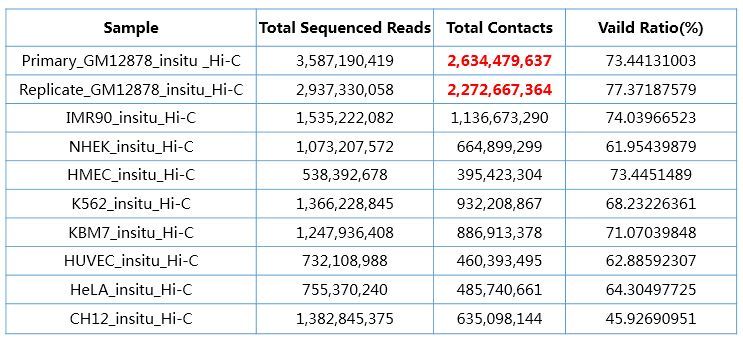
A 3D map of the human genome at kilobase resolution reveals rinciples of chromatin looping. Cell, 2014.
说到已发表文章,自然少不了Erez Lieberman Aiden教授在2014年发表的1kb分辨率文章,达到了当时最高的分辨率。在该篇文章中,人淋巴细胞样本GM12878的测序数据为6.5 billion reads,用于分析的有效数据量为4.9 billion contacts,valid ration(%)在70%以上,达到了950bp的分辨率。
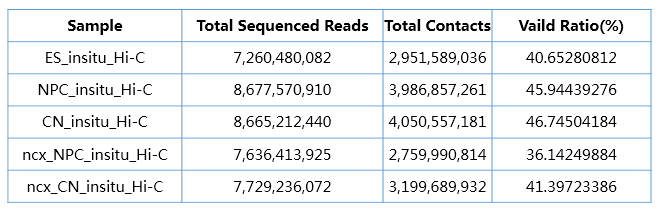
Multiscale 3D Genome Rewiring during Mouse Neural Development.
Cell, 2017.
接着,我们看一下2017年发表在Cell上的文章,该篇研究样本最高分辨率为750 bp,call loop所测最低数据量为7.2 billion reads,用于分析的有效数据量为2.9 billion contacts,valid ration(%)在40%左右。
安诺实测大数据
为了与发表文章进行对比,小编在此公布安诺实测大数据结果,注意哦!是大数据,不是小数据评估结果,大数据可要比小数据难把控很多滴~
下表为安诺两例小鼠细胞样本(Sample A、Sample B)的实测大数据结果:
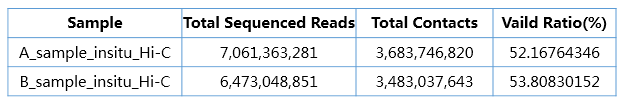
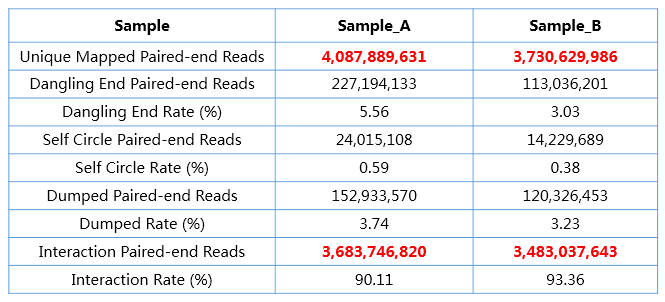
两个小鼠细胞样本测序数据量分别为7.1和6.4 billion reads,用于分析的有效数据量为4.9 billion contacts,valid Ratio>50%,根据文章中的计算公式,此数据达到的分辨率在2kb左右(图2),与发表文献的推荐数据量基本吻合。
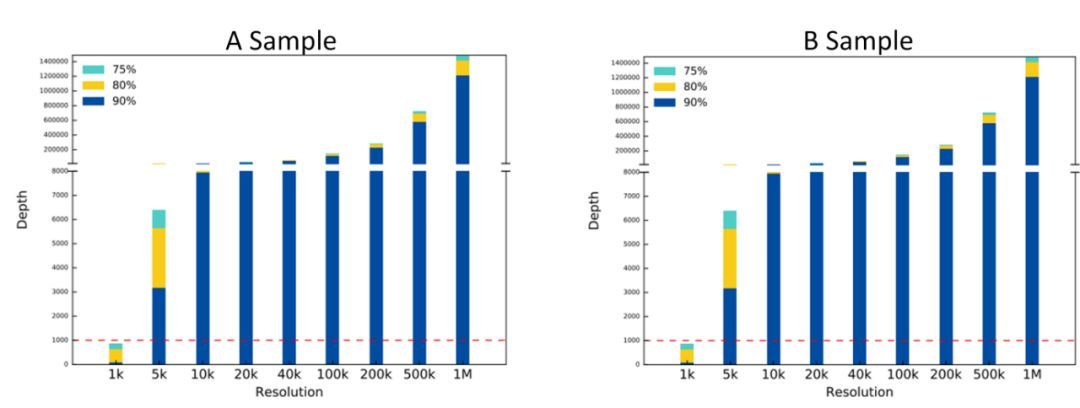
图2 分辨率统计
分析软件大比拼
除了数据量的要求,分析软件的使用也非常关键。2017年发表在Nature Methods上的文章,对10多款分析软件进行了比较分析,在高精度的数据中,HiCCUPS具有最高的保守性。为了更好的展示目前两款主流软件Fit Hi-C和HiCCUPS在call loop分析上的差异,我们进行了实测数据的分析结果对比,见下图。
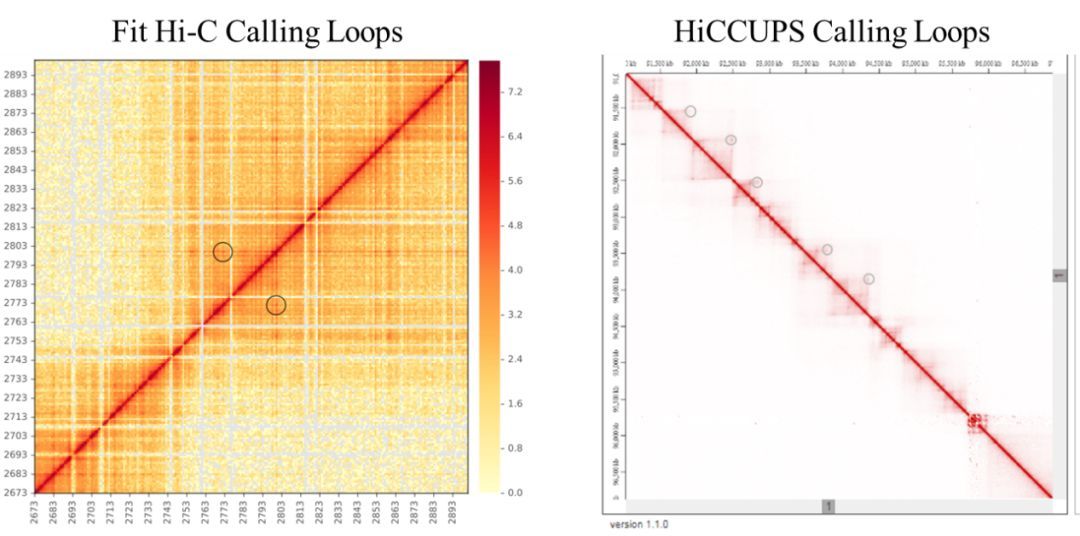
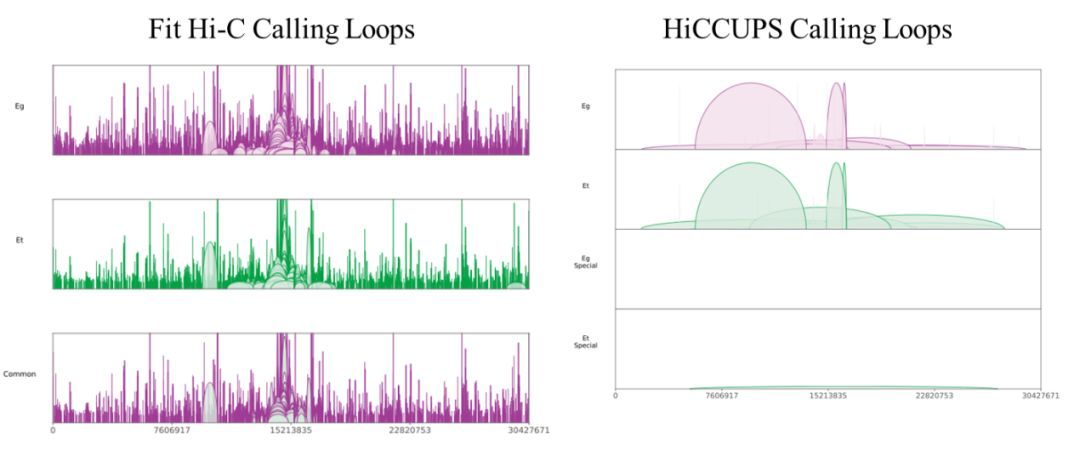
图3 Fit Hi-C和HiCCUPS软件Call Loop比较
从这两张对比图中,我们可以明显看出Fit Hi-C 获得的loops数量非常多,特别是近距离的互作,这样很难找到有效的互作位点。相比之下,HiCCUPS可以获得更有效的loops,calling 得到的loops更加准确。因此,对于差异loops分析而言,HiCCUPS更适合。
安诺Hi-C数据量推荐
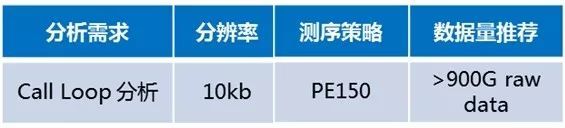
为了给大家提供更专业和更准确的数据分析结果,安诺基因Hi-C分析团队开发了基于GPU的HiCCUPS软件分析流程,做到了更准确的loop分析结果。基于目前高分文章对HiCCUPS软件的认可,我们强烈推荐人类细胞样本的loop分析测序数据量如下:
安诺Hi-C专注三维基因组学研究
1
合作发表文章平均IF 13.20;
2
建库水平一流,低起始量建库无压力;
3
采用多款主流分析软件,HiCCUPS实现更准确Loop calling;
4
目前国内唯一Hi-C云分析服务提供商,让分析更自主。
参考文献
Rao, S. S. et al. A 3D map of the human genome at kilobase resolution reveals principles of chromatin looping. Cell, 2014, 59, 1665-1680.
Bonev B, Cohen N M, Szabo Q, et al. Multiscale 3D Genome Rewiring during Mouse Neural Development. Cell, 2017, 171(3):557-572.e24.
- 本文固定链接: https://www.maimengkong.com/zu/1379.html
- 转载请注明: : 萌小白 2023年3月1日 于 卖萌控的博客 发表
- 百度已收录
