导语
GUIDE ╲
表观遗传学是与遗传学(genetic)相对应的概念。遗传学是指基于基因序列改变所 致基因表达水平变化,如基因突变、基因杂合丢失和微卫星不稳定等;而表观遗传学则是指基于非基因序列改变所致基因表达水平变化,如DNA甲基化和染色质构象变 化等;表观基因组学(epigenomics) 则是在基因组水平上对表观遗传学改变的研究。
数据库介绍
01
DNA甲基化数据库
1. SurvivalMeth
网址:http://bio-bigdata.hrbmu.edu.cn/survivalmeth
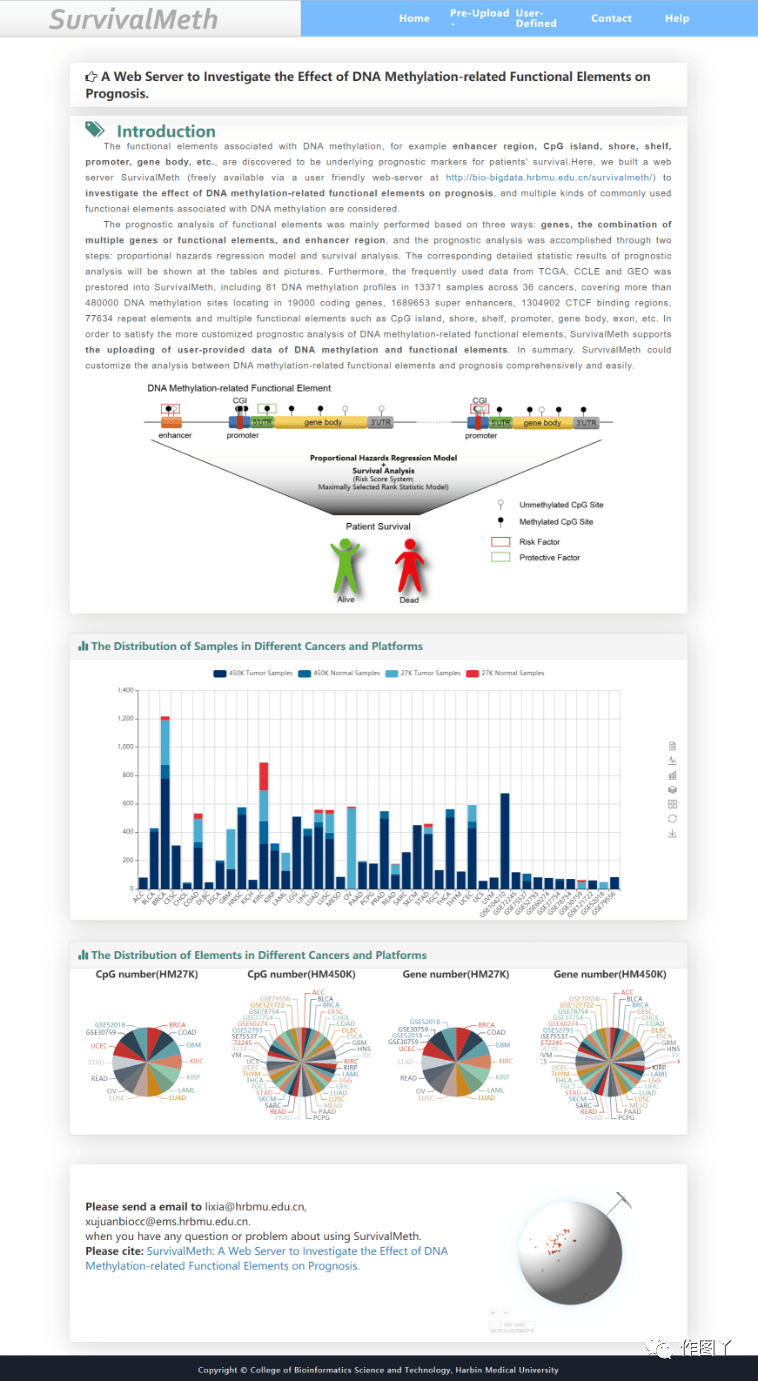
引用:Zhang C, Zhao N, Zhang X, et al. SurvivalMeth: a web server to investigate the effect of DNA methylation-related functional elements on prognosis [published online ahead of print, 2020 Aug 11]. Brief Bioinform. 2020;bbaa162. doi:10.1093/bib/bbaa162
2. MethDB 数据库
网址:http://www.methdb.de
功能:全称DNA Methylation database, 该数据库包含了多种组织多种表型的甲基化模式、甲基化谱和总甲基化内容数据。大约14%的数据没有在其他地方公布。这些数据可以 直接检索。并且有在线提交工具,允许科研人员直接将新数据输入到MethDB 。
引用:Amoreira C, Hindermann W, Grunau C. An improved version of the DNA Methylation database (MethDB). Nucleic Acids Res. 2003;31(1):75-77. doi:10.1093/nar/gkg093
3. PubMeth
网址: http://www.pubmeth.org
功能:PubMeth 收集和整理了文献中与癌症相关的甲基化数据,并进行了人工校对和注释,提供了一个高质量的癌症相关的发生了甲基化基因的数据库。这个数据库只提供在线检索功能,并不开放下载。 也可以提交自己的数据。
在检索时,提供了两种检索方式:①基因②癌症

引用:Maté Ongenaert, Leander Van Neste, Tim De Meyer, Gerben Menschaert, Sofie Bekaert, Wim Van Criekinge, PubMeth: a cancer methylation database combining text-mining and expert annotation, Nucleic Acids Research, Volume 36, Issue suppl_1, 1 January 2008, Pages D842–D846, https://doi.org/10.1093/nar/gkm788
4. DiseaseMeth2
网址: http://bioinfo.hrbmu.edu.cn/diseasemeth
网页打开失败
引用:Xiong, Y., Wei, Y., Gu, Y., Zhang, S., Lyu, J., Zhang, B., Chen, C., Zhu, J., Wang, Y., Liu, H. Zhang Y. (2016) DiseaseMeth version 2.0: a major expansion and update of the human disease methylation database. Nucleic Acids Research.
5.MethCancer
网址:http://methycancer.psych.ac.cn/
功能:首先来看看神器长啥样?界面干净清爽,知道大家也喜欢简约。癌症作为人类健康的头号杀手,引起了科研工作者的官方关注。鉴于DNA甲基化修饰在肿瘤中的重要作用,MethyCancer (database of human DNA Methylation and Cancer)数据库 整合了DNA甲基化数据和基因表达谱数据,将癌症与相关的基因联系起来。
它拥有来自公共资源的高度整合的DNA甲基化数据、癌症相关基因、突变和癌症信息,以及该数据库研究人员大规模测序得到的CpG Island (CGI) clones。分析并给出了不同数据类型之间的互连关系。开发了搜索工具和图形MethyView,以帮助用户访问所有数据和数据连接,并在基因组学和遗传学数据的背景下查看DNA甲基化。
引用:He, X.M., Chang, S.H., Zhang, J.J., Zhao, Q., Xiang, H.Z., ..., and Wang, J. 2008. MethyCancer: the database of human DNA methylation and cancer. Nucleic Acids Res. 36: D836-841
6.MethHC
网址:http://MethHC.mbc.nctu.edu.tw
功能:在MethHC数据库中,提供了18种癌症相关的DNA甲基化,microRNA表达谱和基因表达谱的数据,这里的数据来源于TCGA数据库。同时采用线性回归的方法计算甲基化和表达谱数据之间的关联。
网页打开失败
引用:Huang WY, Hsu SD, Huang HY, et al. MethHC: a database of DNA methylation and gene expression in human cancer. Nucleic Acids Res. 2015;43(Database issue):D856-D861. doi:10.1093/nar/gku1151
7.MethSurv
网址:https://biit.cs.ut.ee/methsurv/
功能:TCGA数据库中收录的主要是450K芯片的数据,也有一些早期27K芯片的数据。本文所述的MethSurv就是基于TCGA数据集中的450K数据构建的可视化分析工具 。MethSurv适用于没有特定生物信息学技能(不熟悉编程分析)的研究人员和临床医生,主要用于探索与癌症患者生存相关的甲基化生物标记物。MethSurv支持对位于查询基因内或附近的CpG进行生存分析。为了进一步挖掘,本文提供了查询基因的聚类分析,以将甲基化模式与临床特征相关联,以及浏览每种癌症类型的顶级生物标记。MethSurv包括来自25种不同人类癌症的7358个甲基 数据。
下面小编随意点了个位点进行分析:
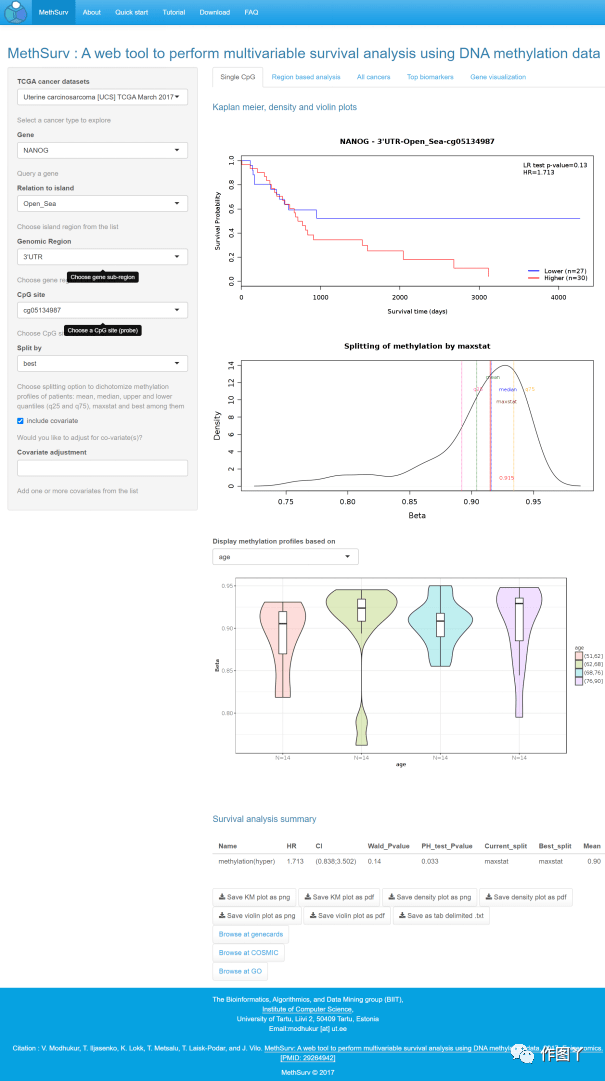
8.MethBank
地址:http://bigd.big.ac.cn/methbank

引用:Li R, Liang F, Li M, et al. MethBank 3.0: a database of DNA methylomes across a variety of species. Nucleic Acids Res. 2018;46(D1):D288-D295. doi:10.1093/nar/gkx1139
9.Lnc2Meth
网址:http://www.bio-bigdata.com/Lnc2Meth/
功能:Lnc2Meth 是由哈尔滨医科大学李霞课题组开发,是lncRNA与DNA甲基化调控关联数据库 ,是基于已有发表文献结果,对其中涉及到在特定研究疾病中的lncRNA及其甲基化结果进行了整理归纳。 使用数据库时可检索的信息主要有两个,一个是lncRNA,一个是某个疾病。对于每一个lncRNA会提供DNA甲基化区域,类型和调控机制,是否在文献报道中有作为诊断分子,发表文献信息等都有整理。对于某种疾病,显示疾病相关的lncRNA。

引用:Zhi H, Li X, Wang P, et al. Lnc2Meth: a manually curated database of regulatory relationships between long non-coding RNAs and DNA methylation associated with human disease. Nucleic Acids Res. 2018;46(D1):D133-D138. doi:10.1093/nar/gkx985
10.MEXPRESS
链接: https://mexpress.be/
功能:MEXPRESS 是一个可视化TCGA 的DNA甲基化,表达和临床数据 ,以及它们之间的工具。http://cancergenome.nih.gov/abouttcga/policies/publicationguidelines是使用指南。

引用:Koch A, Jeschke J, Van Criekinge W, van Engeland M, De Meyer T. MEXPRESS update 2019. Nucleic Acids Res. 2019;47(W1):W561-W565. doi:10.1093/nar/gkz445
11.Wanderer
网址:http://maplab.imppc.org/wanderer/
功能:从TCGA中检索表达和甲基化数据是一项繁琐且耗时的任务。Wanderer是一个直观的网络工具,可以实时访问和可视化TCGA的基因表达和DNA甲基化谱。给定一个TCGA数据集的基因查询和选择(例如,结肠腺癌),数据库提供该癌型表达谱,在单一外显子水平,和DNA甲基化水平的HumanMethylation450 BeadChip位点内部或附近的查询基因。图形和表格输出包括个体和总结数据以及统计测试,允许比较肿瘤和正常概况和探索沿基因组位点和跨肿瘤集合。
网页打开失败
12.表观组关联分析(Epigenome-wide Association Study,EWAS)
(1)EWAS Data Hub
网址:https://bigd.big.ac.cn/ewas/datahub/index
功能:EWAS Data Hub由中国科学院开发, 整合了来自GEO、TCGA、ArrayExpress和ENCODE数据库的共计75,344个样本的DNA甲基化芯片数据和对应的元数据,并采用了有效的归一化方法来消除不同数据集的批次效应。EWAS Data Hub利用海量高质量DNA甲基化数据和标准化元数据的优势,为485,512个探针和36,397个基因提供了一系列重要的评估值(包括组织特异性、年龄相关性、性别差异和种族特异性)和不同背景下的参考DNA甲基化图谱,涉及81种组织/细胞类型(包含25个脑部和25种血细胞类型),67种疾病(包括39种癌症),不同年龄、性别、种族和BMI。同时,EWAS Data Hub 还提供了高效的查询方式:(1)通过年龄,疾病等信息快速查询感兴趣样本的数据和元数据,并能打包下载。(2)通过基因ID、探针号等信息浏览该探针/基因在不同背景下的参考DNA甲基化图谱。
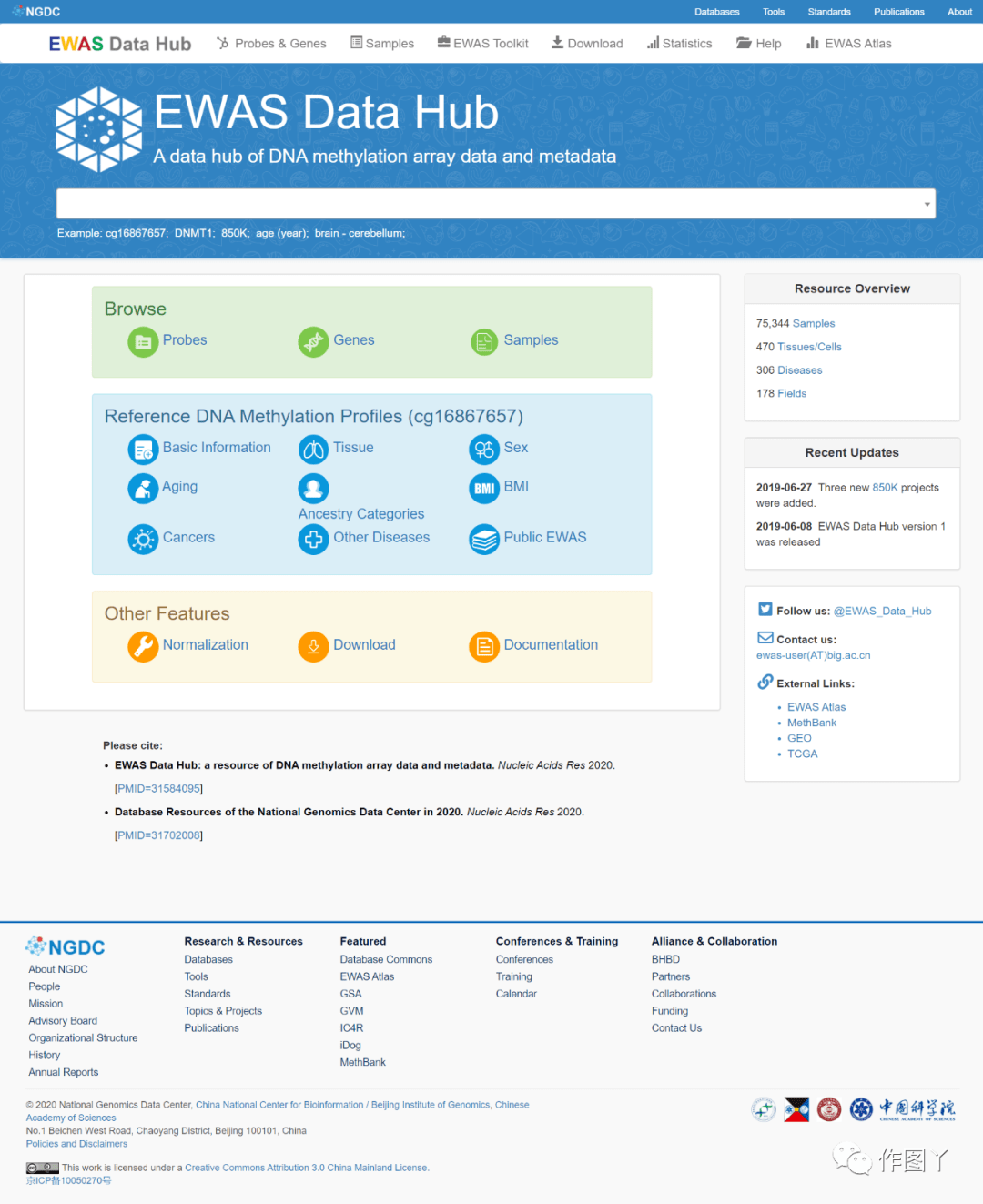
引用:Xiong Z, Li M, Yang F, et al. EWAS Data Hub: a resource of DNA methylation array data and metadata. Nucleic Acids Res. 2020;48(D1):D890-D895. doi:10.1093/nar/gkz840
(2)IHEC Data Portal
网址:https://epigenomesportal.ca/ihec/
功能:该网站提供来自七个国际联盟——ENCODE、NIH Roadmap、CEEHRC、Blueprint、DEEP、AMED-CREST和KNIH——的数据,包括来自600多个不同组织的7000多个表观基因组参考数据集。用户可以选择和浏览各种数据集。 还可以依靠相关工具来比较所选的数据集。数据也可以下载,但原始数据需要数据访问请求。同时,用户可以共享他们选择的数据集。
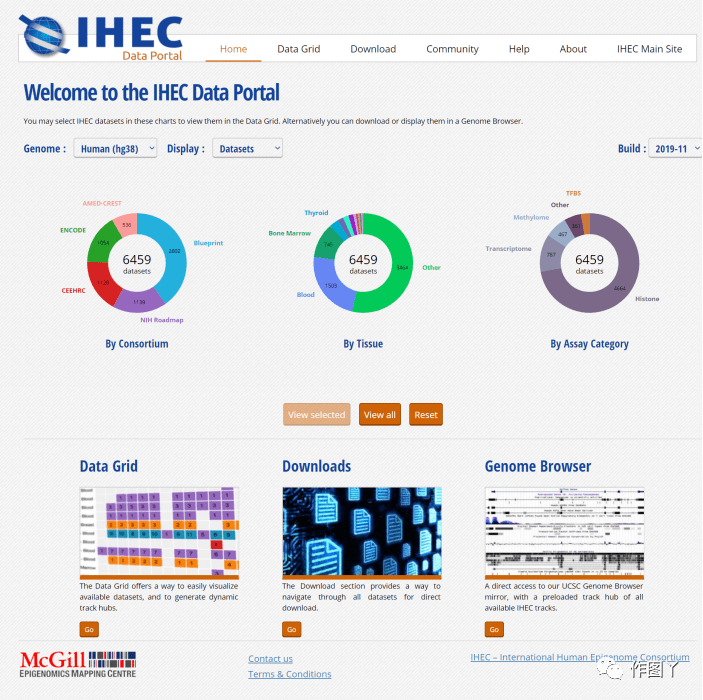
引用:Bujold D, Morais DAL, Gauthier C, et al. The International Human Epigenome Consortium Data Portal. Cell Syst. 2016;3(5):496-499.e2. doi:10.1016/j.cels.2016.10.019
(3)Blueprint Data Analysis Portal
网址:http://blueprint-data.bsc.es
功能:Blueprint Data Analysis Portal用于分析来自Blueprint Consortium的数据。该联盟生成了造血细胞谱系的参考表观基因组,其数据集包括ChIP-seq、DNAsel-seq、全基因组重亚硫酸盐测序和RNA-seq数据,涵盖了60多种细胞类型。在该门户网站中,研究人员使用表观基因组学比较网络基础设施(epigenomics comparative cyber-infrastructure,EPICO)平台,该平台包括五个部分:数据模型;数据验证和加载程序;空数据库,用于存储来自数据验证和加载程序的数据和元数据;应用程序界面(API);以及数据分析门户网站。除了EPICO外,这种方法还需要存储空间来创建数据库,获取原始数据的连接,以及接收查询和发送结果的模块。该门户网站允许几乎没有生物信息学背景的用户,可视化和比较感兴趣血细胞类型的表观基因组和转录组数据。
网站一直在加载...不知道怎么回事
引用:Fernández JM, de la Torre V, Richardson D, et al. The BLUEPRINT Data Analysis Portal. Cell Syst. 2016;3(5):491-495.e5. doi:10.1016/j.cels.2016.10.021
(4)eFORGE
网址:http://eforge.cs.ucl.ac.uk/
功能:eFORGE允许用户从表观基因组关联分析(EWAS)中筛选数据 ,发现与疾病相关的细胞类型。eFORGE可以估计哪些差异甲基化位点在某些组织或细胞中可能是起作用的。它是通过分析一组差异甲基化位点和DNase I超敏位点参考图谱之间的重叠来实现的。这些参考集包括来自各种组织,原代细胞类型和来自ENCODE、Roadmap Epigenomics和Blueprint consortia的细胞系的454个样品。
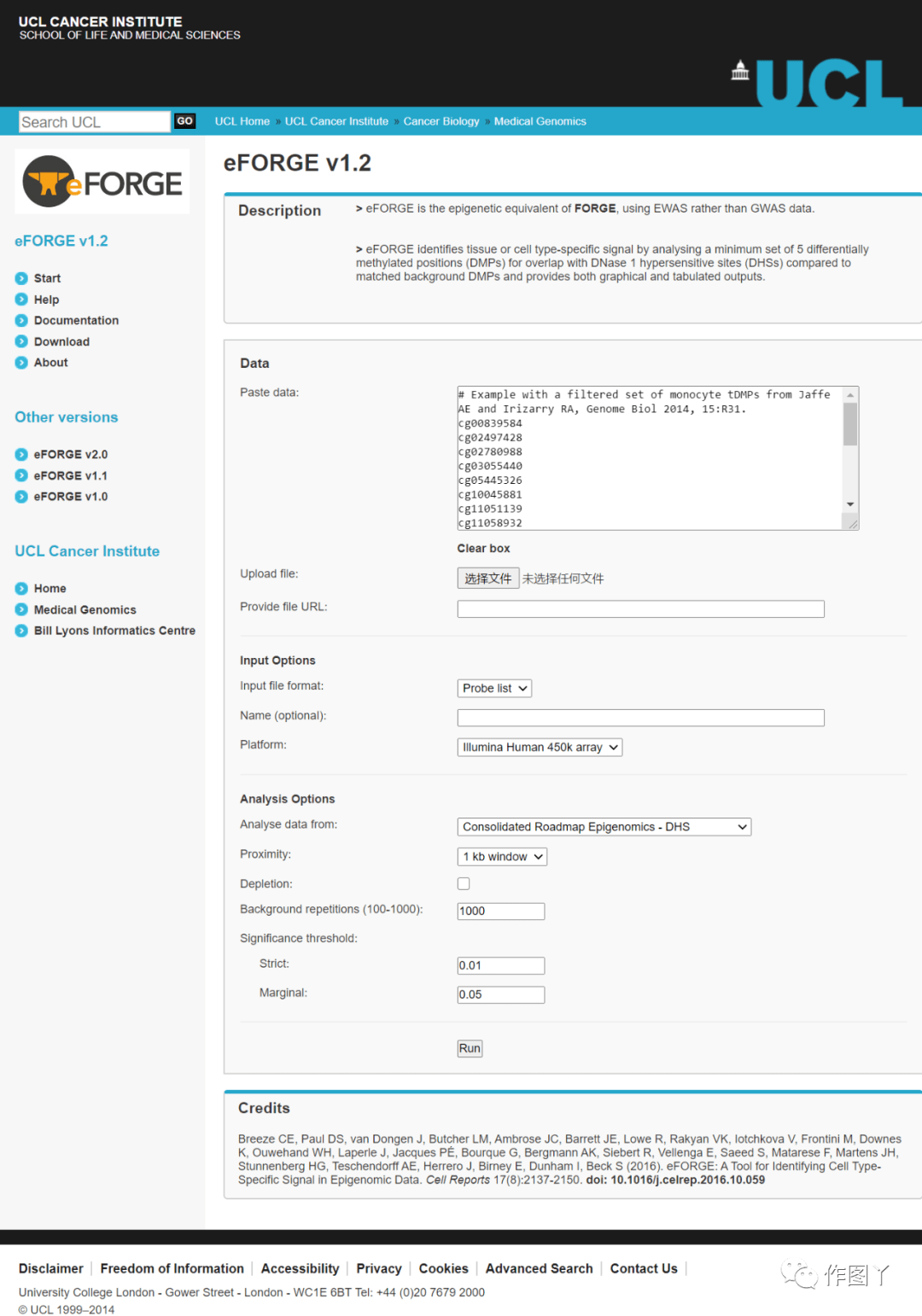
引用:Breeze CE, Paul DS, van Dongen J, et al. eFORGE: A Tool for Identifying Cell Type-Specific Signal in Epigenomic Data. Cell Rep. 2016;17(8):2137-2150. doi:10.1016/j.celrep.2016.10.059
13.iMETHYL
网址:http://imethyl.iwate-megabank.org/index.html
功能:iMETHYL提供了大约100名受试者的CD4+ t淋巴细胞、单核细胞和中性粒细胞的全DNA甲基化(约2400万个常染色体CpG位点)、全基因组(约900万个单核苷酸变异)和全转录组(> 14000个基因)数据。这些数据来自亚硫酸氢盐全基因组测序、全基因组测序和全转录组测序,使iMETHYL成为一个全面的数据库。iMETHYL整合了SNP, DNA甲基化和RNA表达谱的数据,并进行了两两之间的关联分
在STATISTICS页面,提供了两个基本信息。 三种组学数据的分布。可以看到志愿者的性别,年龄的基本情况,以及DNA甲基化,基因表达和SNV的汇总信息。
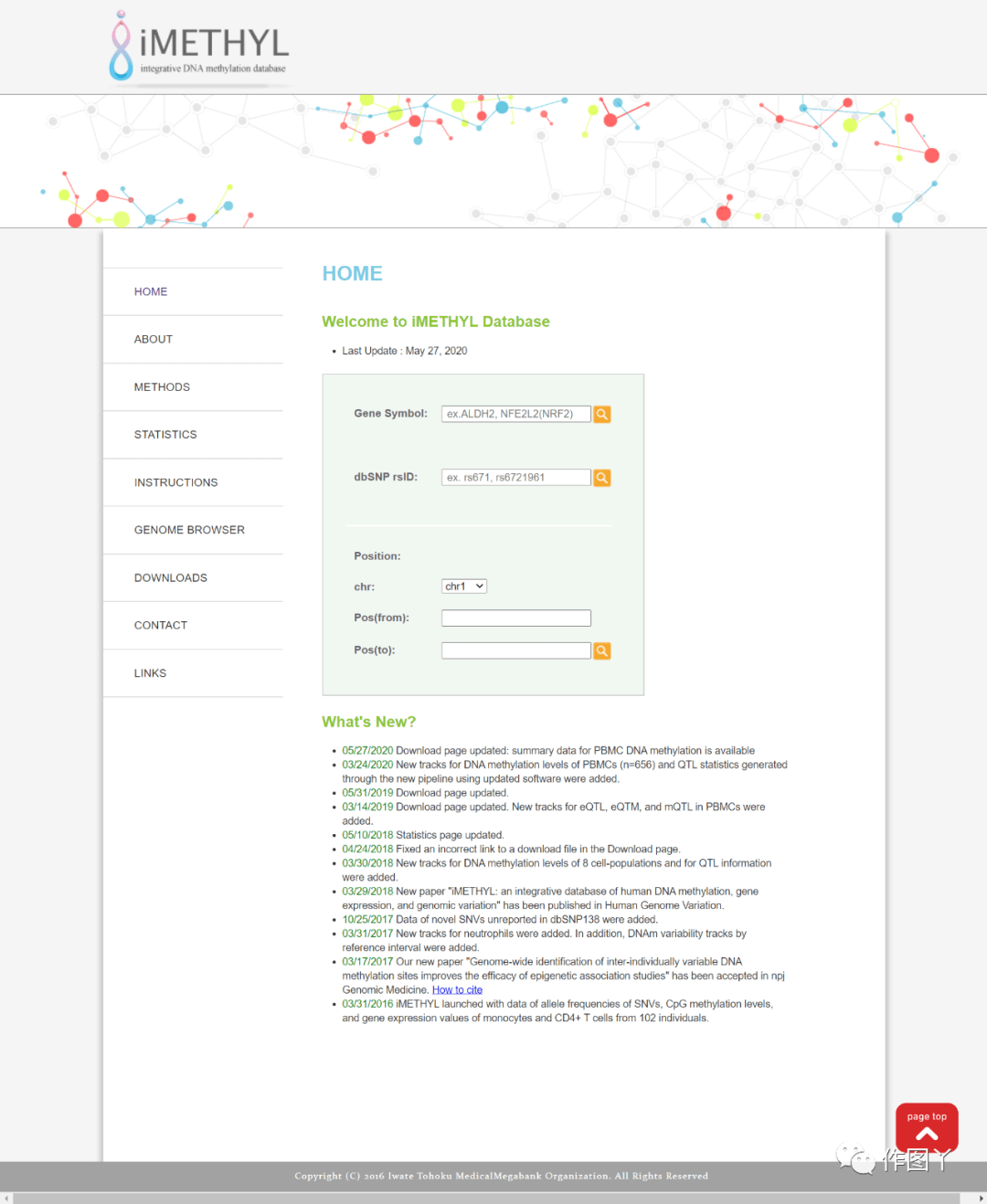
引用:Komaki S, Shiwa Y, Furukawa R, et al. iMETHYL: an integrative database of human DNA methylation, gene expression, and genomic variation. Hum Genome Var. 2018;5:18008. Published 2018 Mar 29. doi:10.1038/hgv.2018.8
14. HeteroMeth
网址:http://qianlab.genetics.ac.cn/HeteroMeth
功能:是由中国科学院开发的首个DNA甲基化异质性数据库 ,用于检索、浏览、可视化和下载多个物种、突变体、发育阶段和组织的DNA甲基化异质性数据库HeteroMeth。
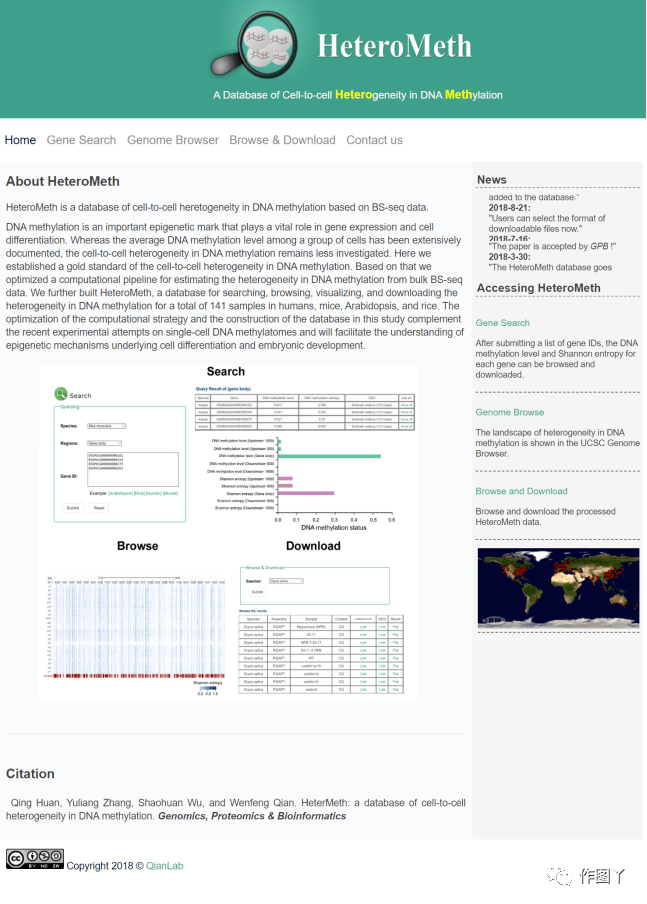
引用:Huan Q, Zhang Y, Wu S, Qian W. HeteroMeth: A Database of Cell-to-cell Heterogeneity in DNA Methylation. Genomics Proteomics Bioinformatics. 2018;16(4):234-243. doi:10.1016/j.gpb.2018.07.002
15.DNA甲基化预测免疫浸润
我们之前写过一个 MethylCIBERSORT方法,可以针对DNA甲基化数据预测细胞的浸润 Nature Communication| DNA甲基化评估细胞浸润
02
M6A数据库
n6 -甲基ladenosine (m6A)是真核生物中最常见的转录后修饰,在剪接、RNA降解、RNA-蛋白相互作用等多种生物过程中发挥着关键作用。
1. Whistle
网址:http://180.208.58.19/whistle/index.html
功能:WHITSLE实现对转录组m6A RNA甲基化位点预测。通过把测序数据和机器学习的整合来寻找m6A调控的位点。只需要输入想要检索的基因即可,如果我们想要看某一个功能收到m6A甲基化的影响也是可以通过输入功能的名字来查看的。
引用:Chen K, Wei Z, Zhang Q, et al. WHISTLE: a high-accuracy map of the human N6-methyladenosine (m6A) epitranome predicted using a machine learning approach. Nucleic Acids Res. 2019;47(7):e41. doi:10.1093/nar/gkz074
2. RMBase v2.0
网址:http://rna.sysu.edu.cn/rmbase/
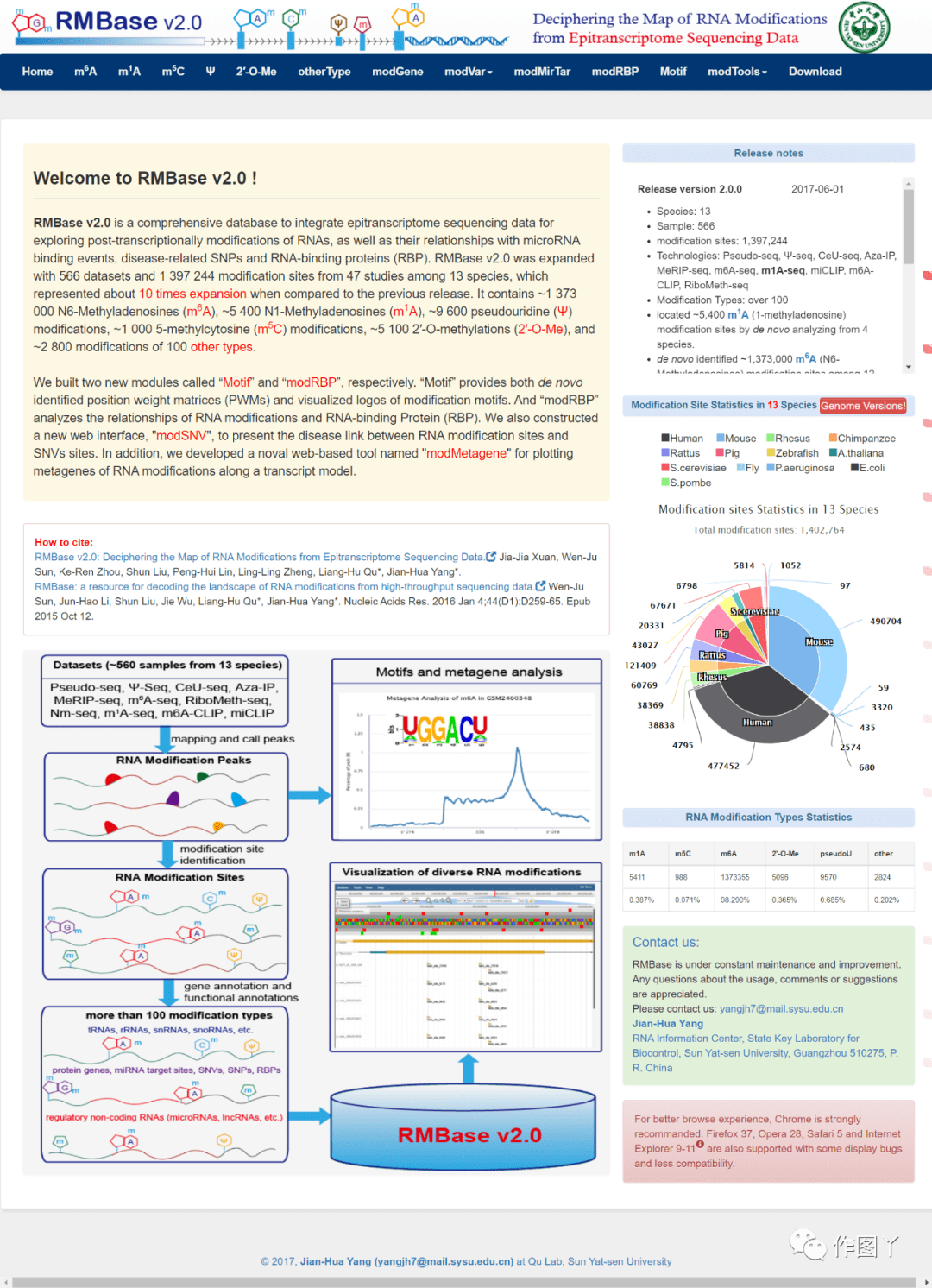
引用:Xuan JJ, Sun WJ, Lin PH, et al. RMBase v2.0: deciphering the map of RNA modifications from epitranome sequencing data. Nucleic Acids Res. 2018;46(D1):D327-D334. doi:10.1093/nar/gkx934
3. MeT-DB V2.0
网址:http://www.xjtlu.edu.cn/metdb2
功能:这个数据库收集了8个m6A相关调控因子(FTO, KIAA1429, METTL14, METTL3, WTAP, HNRNPC, YTHDC1, YTHDF1)的 ParCLIP-seq 和 MeRIP-seq 数据来构建的,通过相关的测序数据,我们可以查看具体的调控位置。还提供了和其他类似 miRNA 数据库以及剪切因子数据库交叉分析的结果 。
对比以前的版本,以更侧重于说明特定背景的m6A功能。根据26项独立研究对7个物种的185个样本进行的预测,Met-DB v2.0在特定 背景的m6A峰值和单碱基位点上有显著增加。此外,还 有m6A readers, erasers 和writers 的靶点的新数据,并扩展了更多功能性数据。
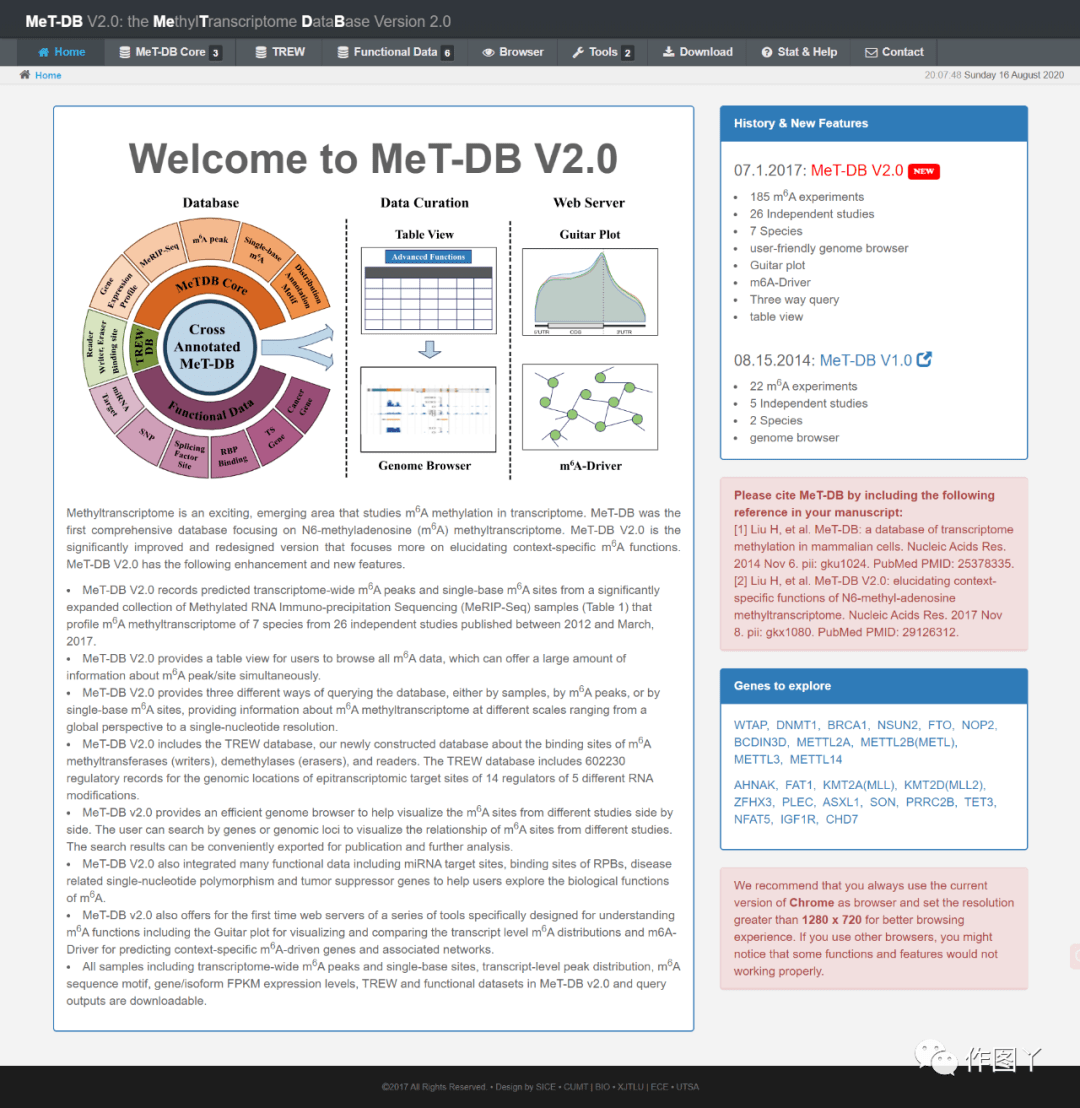
引用:Liu H, Wang H, Wei Z, et al. MeT-DB V2.0: elucidating context-specific functions of N6-methyl-adenosine methyltranome. Nucleic Acids Res. 2018;46(D1):D281-D287. doi:10.1093/nar/gkx1080
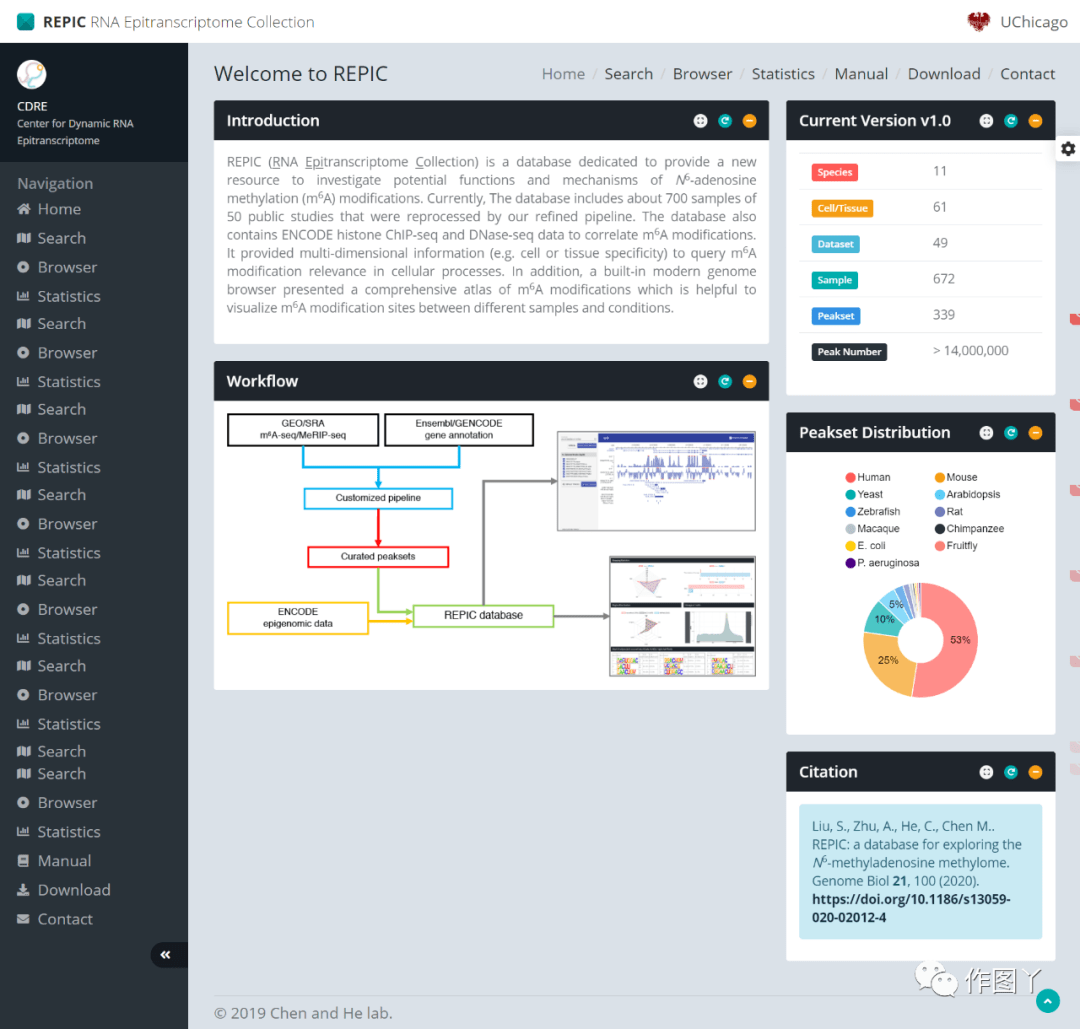
4. REPIC
网址:https://repicmod.uchicago.edu/repic
功能:REPIC (RNA EPItranome Collection)数据库是20年4月 新发的数据库,对于测序数据的纳入的比较多。记录了从公开的m6A-seq和MeRIP-seq数据调用的大约1000万个峰值。这些数据来自49项研究的672份样本,涵盖了11个生物体的61个细胞系或组织。REPIC允许用户通过特定的细胞系或组织类型查询m6A修饰位点。此外,它整合了来自ENCODE项目的1418个组蛋白ChIP-seq和118个DNA -seq数据 的m6A/MeRIP-seq数据,呈现了一个完整的m6A甲基化 位点图谱。
5. m6A2Target
网址:http://m6a2target.canceromics.org/#/
功能:m6A2Target是一个完整的 m6A 修饰的writers, erasers 和readers (WERs)靶基因的数据库。它将低通量实验验证的高机密性靶标与高通量测序(如CLIP-Seq、RIP-seq和ChIP-seq)显示的证据结合或从m6A应答扰动和随后的高通量测序(如RNA-Seq、m6A-Seq和Ribo-Seq)推断的潜在靶标整合在一起。
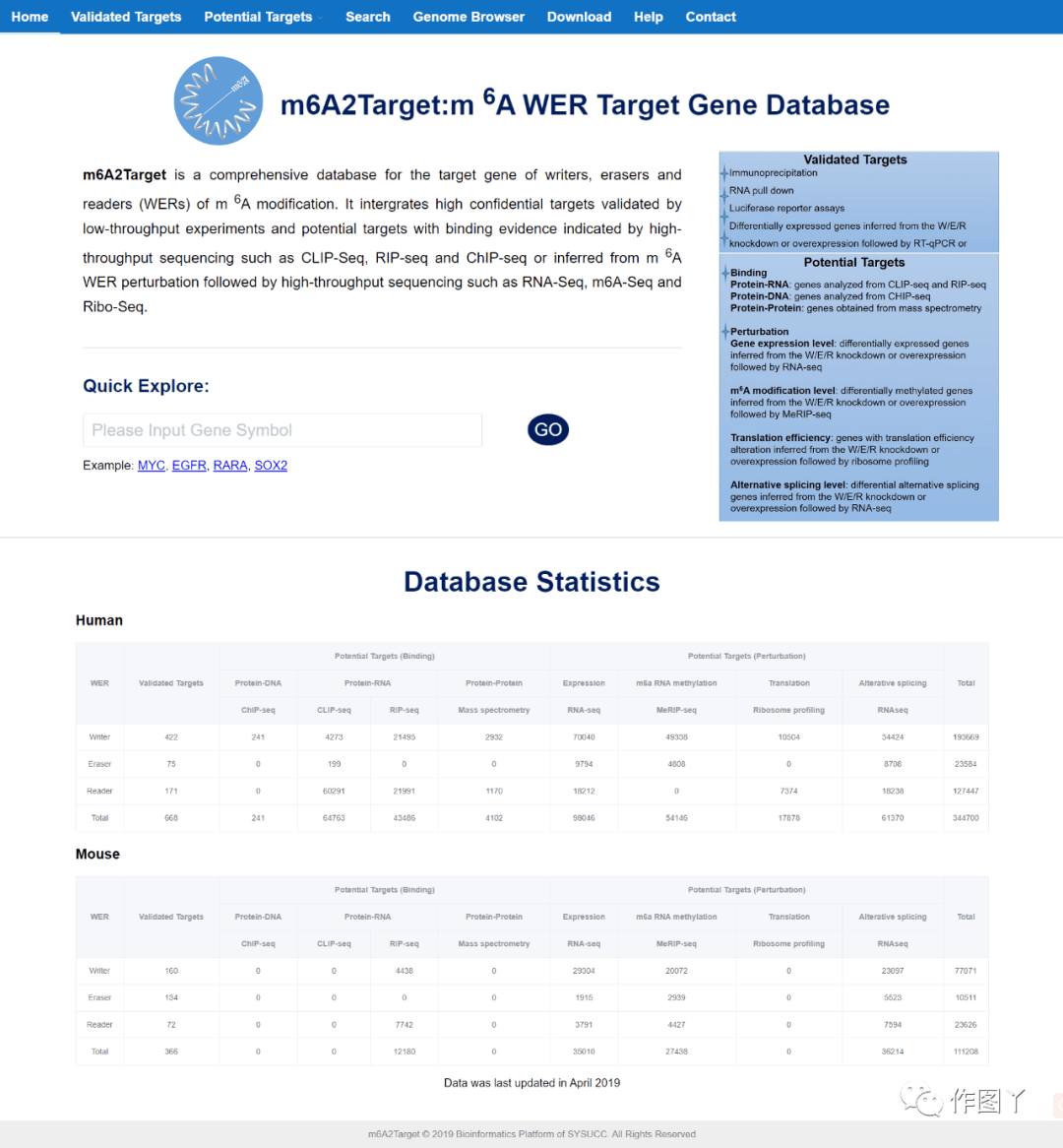
引用:Deng S, Zhang H, Zhu K, et al. M6A2Target: a comprehensive database for targets of m6A writers, erasers and readers [published online ahead of print, 2020 May 11]. Brief Bioinform. 2020;bbaa055. doi:10.1093/bib/bbaa055
03
组蛋白修饰数据库
1. HIstome
网址:http://www.actrec.gov.in/histome/
功能:HIstome( The Histone Infobase)是一个可浏览的,手动管理的,关系数据库,提供关于人类组蛋白的信息,包括位点修饰,变异和修饰酶。HIstome是一个包含55种人类组蛋白、106种不同的翻译后修饰位点(PTMs)和152种组蛋白修饰酶的知识库。条目被分为5种组蛋白,8种翻译后修饰和14种酶催化添加和去除这些修饰。
网页发生错误
引用:Khare SP, Habib F, Sharma R, Gadewal N, Gupta S, Galande S. HIstome--a relational knowledgebase of human histone proteins and histone modifying enzymes. Nucleic Acids Res. 2012;40(Database issue):D337-D342. doi:10.1093/nar/gkr1125
2. HHMD
网址:http://bioinfo.hrbmu.edu.cn/hhmd
功能:HHMD(Human Histone Modification Database)是由哈尔滨医科大学李霞教授团队开发的,HHMD重点对实验室实验获得的组蛋白修饰数据集进行存储和集成。包含了43种人类定位特异性组蛋白修饰。可以通过组蛋白修饰、基因ID、功能类别、染色体位置和肿瘤名称进行检索。HHMD还包括一个用户友好的可视化工具命名为HisModView,通过该基因组组蛋白修饰地图可以显示。HisModView有助于组蛋白的获取和可视化。
网页打开失败
引用:Zhang Y, Lv J, Liu H, et al. HHMD: the human histone modification database. Nucleic Acids Res. 2010;38(Database issue):D149-D154. doi:10.1093/nar/gkp968
3.ChIPBase v2.0
网址:http://rna.sysu.edu.cn/chipbase/
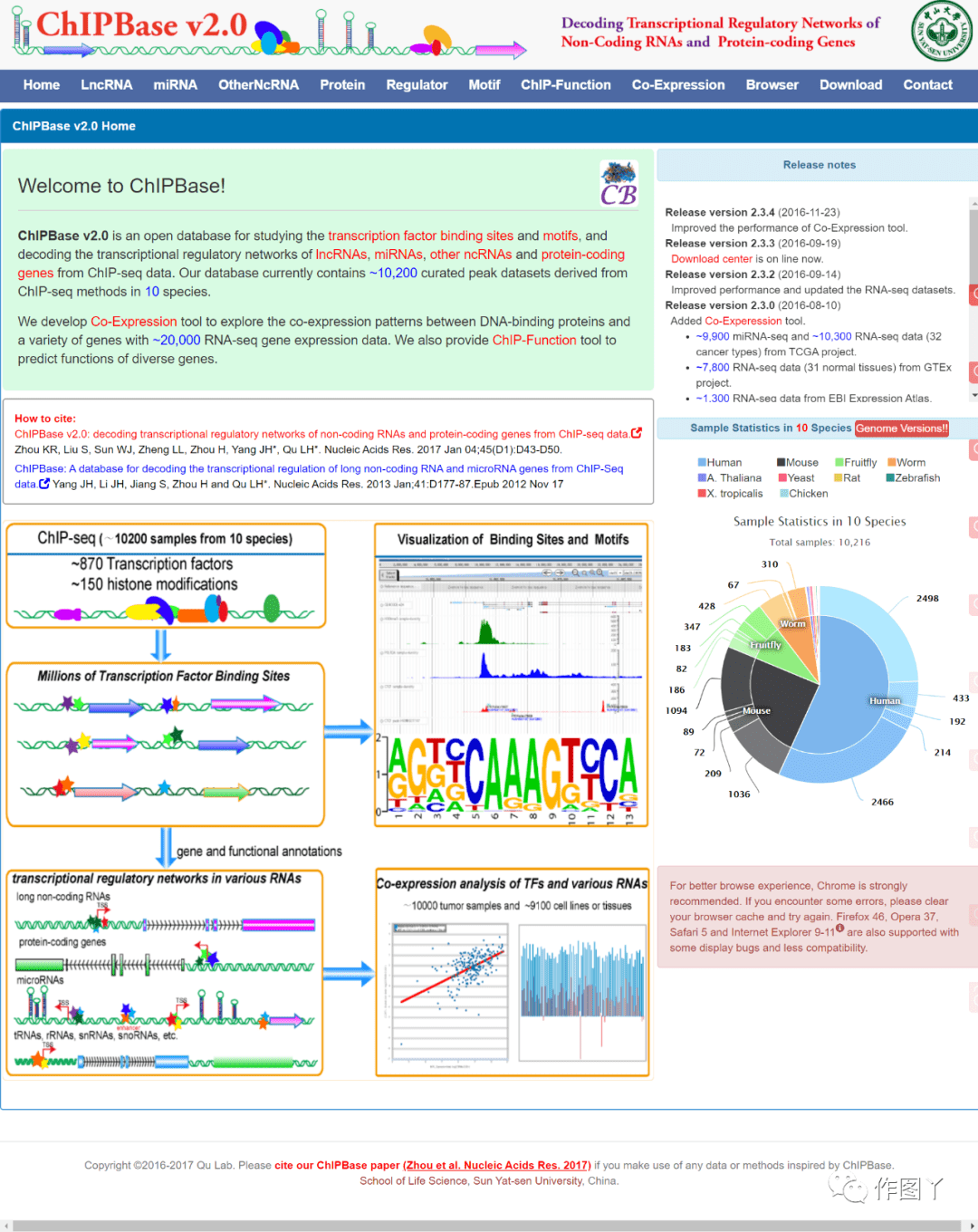
引用:Zhou KR, Liu S, Sun WJ, et al. ChIPBase v2.0: decoding tranional regulatory networks of non-coding RNAs and protein-coding genes from ChIP-seq data. Nucleic Acids Res. 2017;45(D1):D43-D50. doi:10.1093/nar/gkw965
04
其他
1.HEDD
网址:http://zdzlab.einstein.yu.edu/1/hedd.php
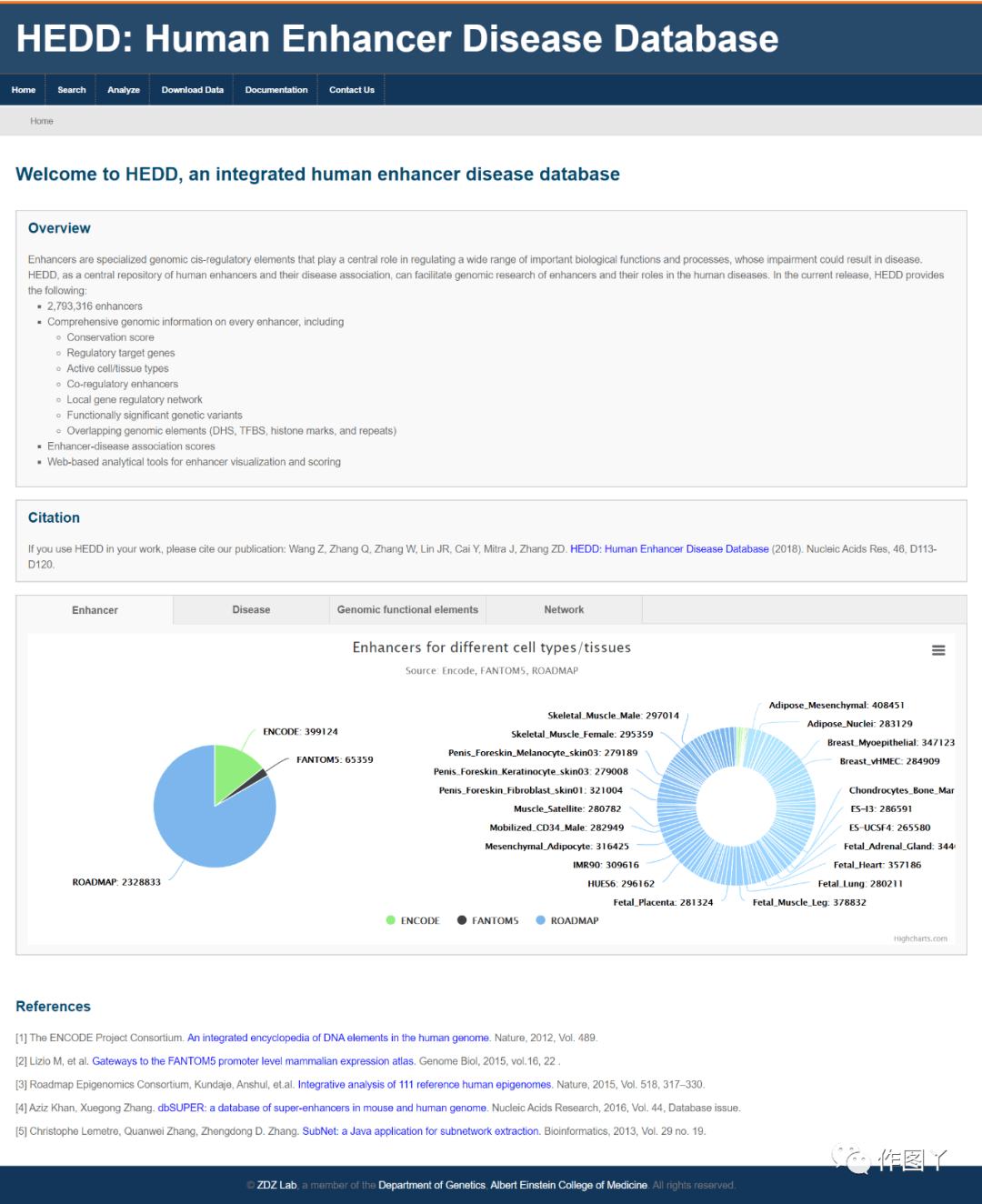
引用:Wang Z, Zhang Q, Zhang W, et al. HEDD: Human Enhancer Disease Database. Nucleic Acids Res. 2018;46(D1):D113-D120. doi:10.1093/nar/gkx988
2.Epifactors
网址:http://epifactors.autosome.ru/
功能:对于DNA甲基化,组蛋白修饰,染色质重塑等表观遗传标记分子的研究迅猛发展,在相关领域已经积累了大量的认知。通过对研究表明的参与这些表观遗传机制的蛋白对应的基因,功能进行整理和归纳,Epifactors数据库在传统的直接参与表观遗传的蛋白的基础上,定义了表观因子这个概念,将直接参与表观遗传的核心蛋白和其周边蛋白联系起来,进一步丰富了表观遗传中相关分子的网络结构。
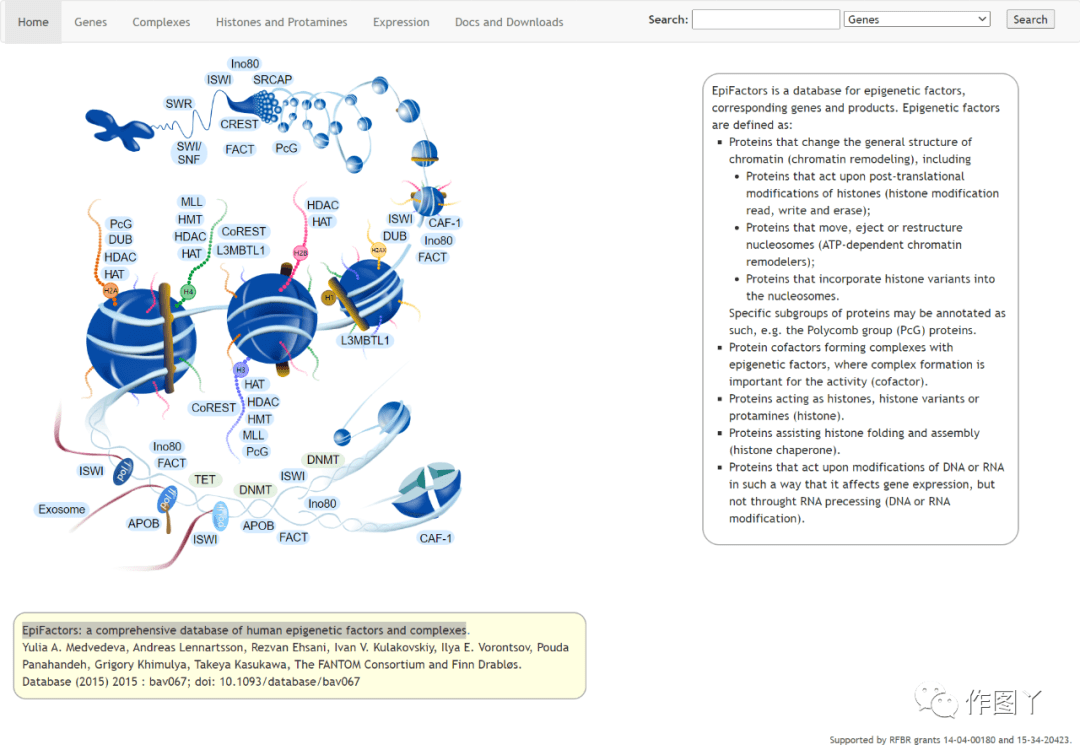
引用:Medvedeva YA, Lennartsson A, Ehsani R, et al. EpiFactors: a comprehensive database of human epigenetic factors and complexes. Database (Oxford). 2015;2015:bav067. Published 2015 Jul 7. doi:10.1093/database/bav067
小编总结
DNA甲基化,组蛋白修饰,染色质重塑等表观遗传标记分子的研究迅猛发展,在相关领域已经积累了大量的认知。这些标志与癌症的发展有密不可分的联系,小编今天分享了一些表观基因组的数据资源,大家快点学习起来吧~
注:封面图片来源网络
- 本文固定链接: https://www.maimengkong.com/moreshare/1335.html
- 转载请注明: : 萌小白 2023年1月13日 于 卖萌控的博客 发表
- 百度已收录
