Life is just a series of trying to make up your mind
生活只是由一系列下决心的努力所构成
下定决心去做一件让你改变的事的开始是一件很痛苦的事情(至少我有这种感觉),其实人长大积累了各种各样的习惯,也是这些习惯构成了你现在的生活,让你抛弃一种习惯去学习新的习惯(即使这个习惯会使你成为更好的自己)就犹如蛇蜕皮迎新生,旧的习惯就像包裹我们的一层皮,丢掉它会有刮肤之痛但成就全新的自己,而我目前就处在改变的早期,每天都要鼓励自己几十遍(我以前真的。。是有多懒啊。。),做的不够好,还是很情绪化,希望以后进步一些,加油!自己!
这周的学习感觉是对脑子的考验,开始要用服务器了,要写那些以前看到只会吐槽什么鬼的符号,感觉智商被嫌弃了,不过好歹也是正常长大的孩子,认真起来自己都爱。。。废话少说,言归正传,下面讲一下对于一个零基础的服务器运用者怎么进行数据分析的第一步:数据的质量控制(FastQC)
(一)服务器的基本使用:
-
在Xshall5上登录服务器:名称和主机部分需要填写
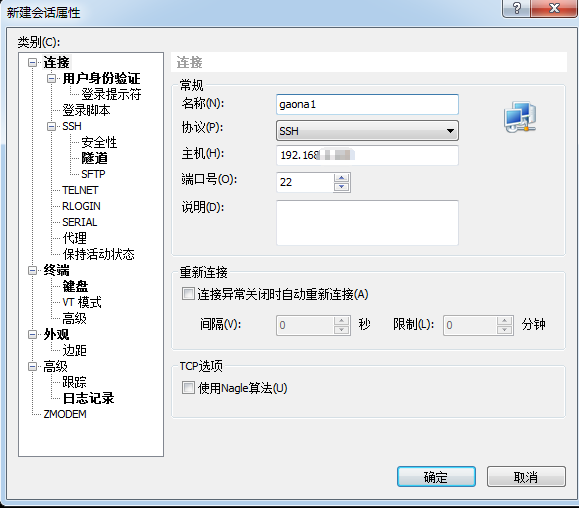
2. 点击确定后会弹出会话框填写用户名和密码之后点击链接来连接服务器,出现以下页面:系统默认提示符是美元符号$,这个符号表明shell在等待用户输入。$前面中gaona为用户ID名,localhost为系统名,如下图:
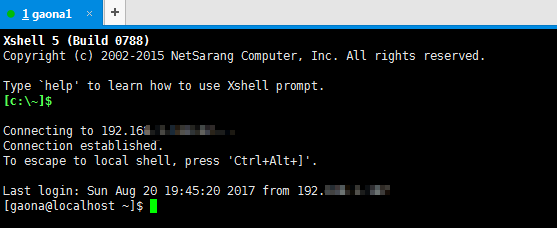
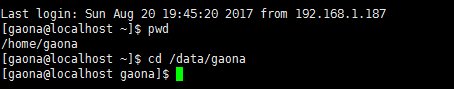
3. 接下来就开始输入命令运行,pwd命令可以显示出shell会话的当前目录,即home目录中的gaona目录,然后使用切换目录命令cd将shell会话切换到另一个目录,比如切换到data目录中的gaona目录,显示如下:
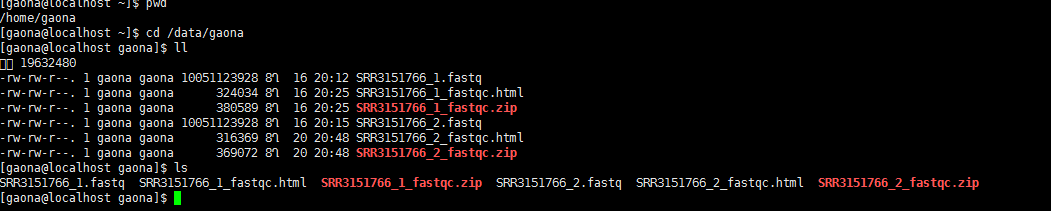
4. 接下来就是查看gaona目录中的内容,ls命令只会显示简单的文件名。而ll命令会显示更全面的内容,如下:
5. 我们的目的是对原始数据进行fastqc,既然已经看到了要找的文件就可以进行下一步了,输入命令fastqc SRR3151733_2.fastq,即输入fastqc加文件名全称,运行结果如下图:出现最后一行即运行结束。
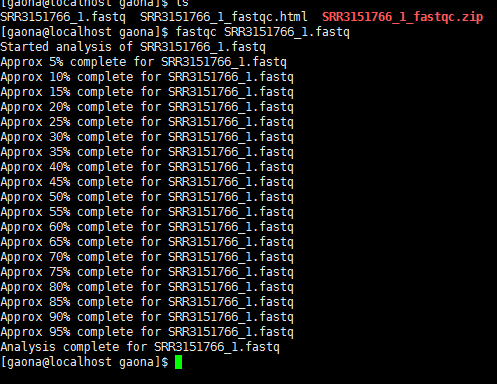
6. 运行结束后查看运行结果即:用ll命令:
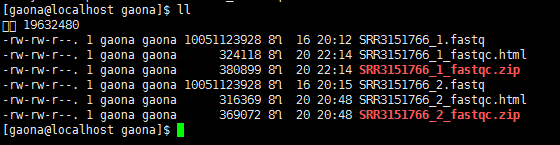
7. 输出的压缩文件将是SRR3151766_1_fastqc.zip,解压后,查看html格式的结果报告,结果分为绿色的“PASS”,黄色的“WARN”和红色的“FALL”结果如下图:

(二)下面详细介绍以上信息的内容
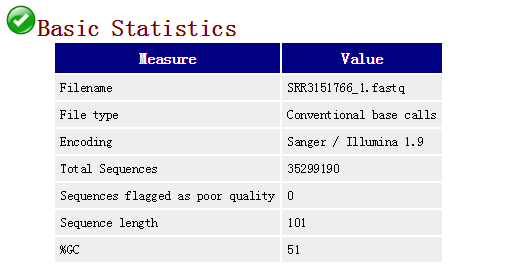
1. basic statistics基本结果图如下:
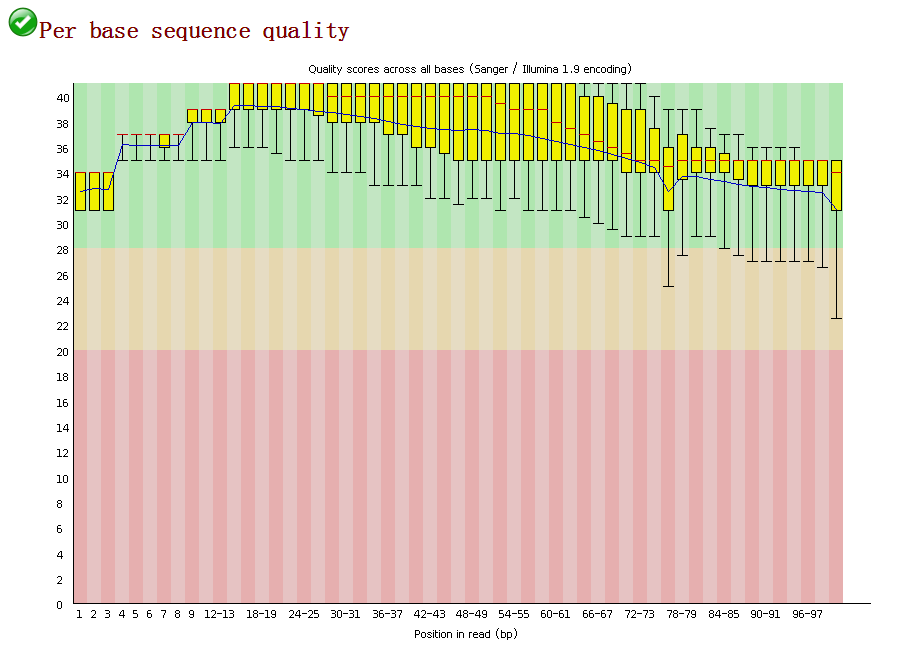
2. per base sequence quality quality就是fred值,-10*log10(p),p为测错的概率。所以一条reads某位置出错概率为0.01,其quality就是20。
横轴代表位置,纵轴quality.红色表示中位数,黄色是25%-75%区间,触须是10%-90%区间,蓝线是平均数。若任一位置的下四分位数低于10或者中位数低于25,报“WRAN”;若任一位置的下四分位数低于5或者中位数低于20,报“FALL”。
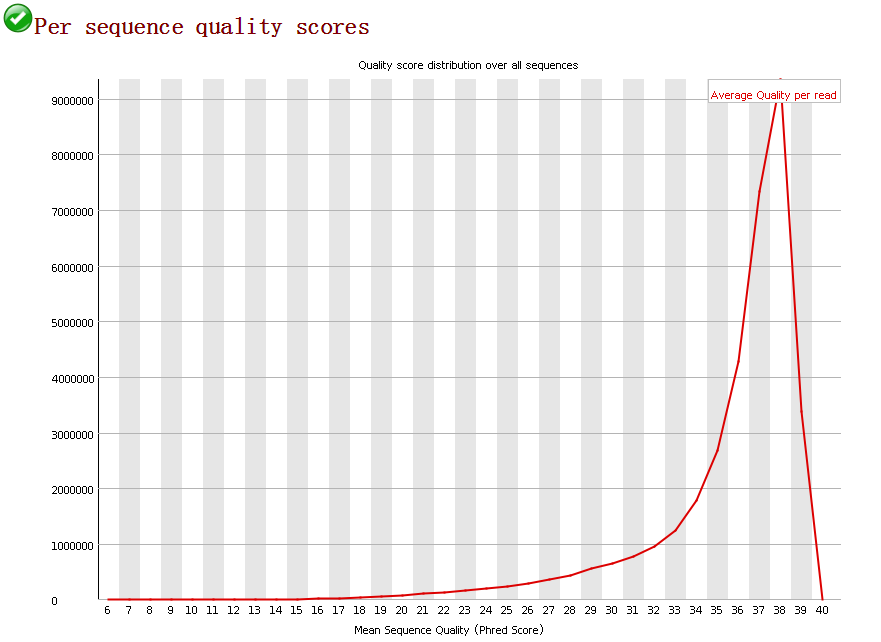
3.per sequence quality score 每条每条read的quality的均值分布。横轴为quality,纵轴是reads数。当峰值小于27(错误率0.2%)时报“WRAN”,当峰值小于20(错误率1%)是报“FALL”。
4.per base sequence content对所有reads的每个位置,统计ATGC四种碱基的分布。横轴为位置,纵轴为百分比。正常情况下四种碱基的出现频率应该是接近的,而且没有位置差异。因此好的样本中四条线应该是平行且接近的。当部分位置碱基的比例出现bias时,即四条线在某些位置纷乱交织,往往提示有overrepresented sequence 的污染,当所有的碱基比例一致地表现出bias时,即四条线平行但分开,往往代表文库有bias(建库过程或本身特点),或者是测序中的系统误差。当任一位置的A/T比例与G/C比例相差超过10%,报“WRAN”,当任一位置的A/T比例与G/C比例相差超过20%,报“FALL”。

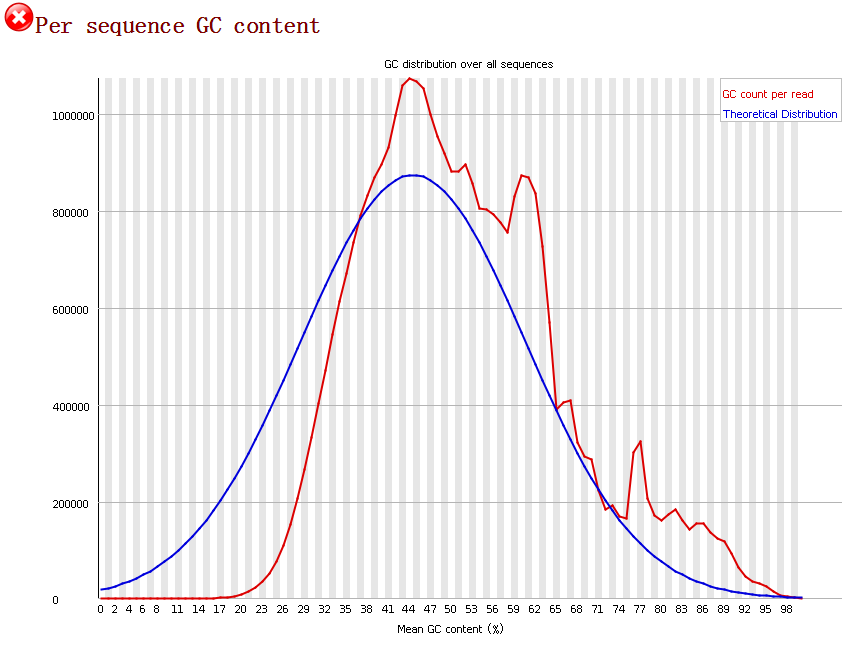
5.per sequence GC content统计reads的平均GC含量的分布。红线是实际情况,蓝线是理论分布(正态分布,均值不一定在50%,而是有平均GC含量推断的)。出现bias时推论同上。偏离理论分布的reads超过15%时,报“WRAN”, 偏离理论分布的reads超过30%时,报“FALL”。
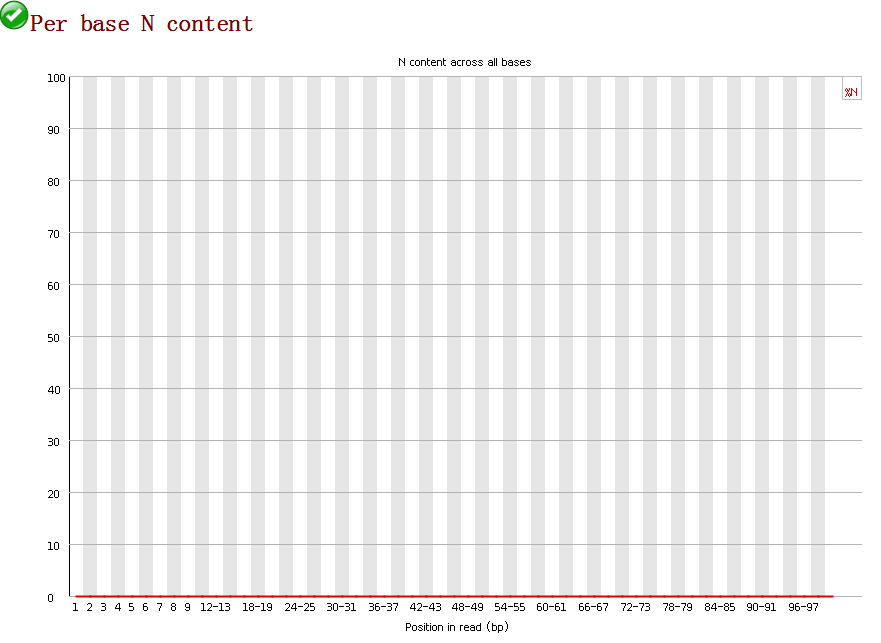
6.per base N content 当测序仪器不能辨别某条read的某个位置到底是什么碱基时,就会产生“N”.对所有reads的每个位置统计N比率。正常情况下N的比例很小,所以图上常常看到一条直线,但放大Y轴之后会发现还是有N的存在。当任一位置的N的比例超过5%,报“WRAN”, 当任一位置的N的比例超过20%,报“FALL”。
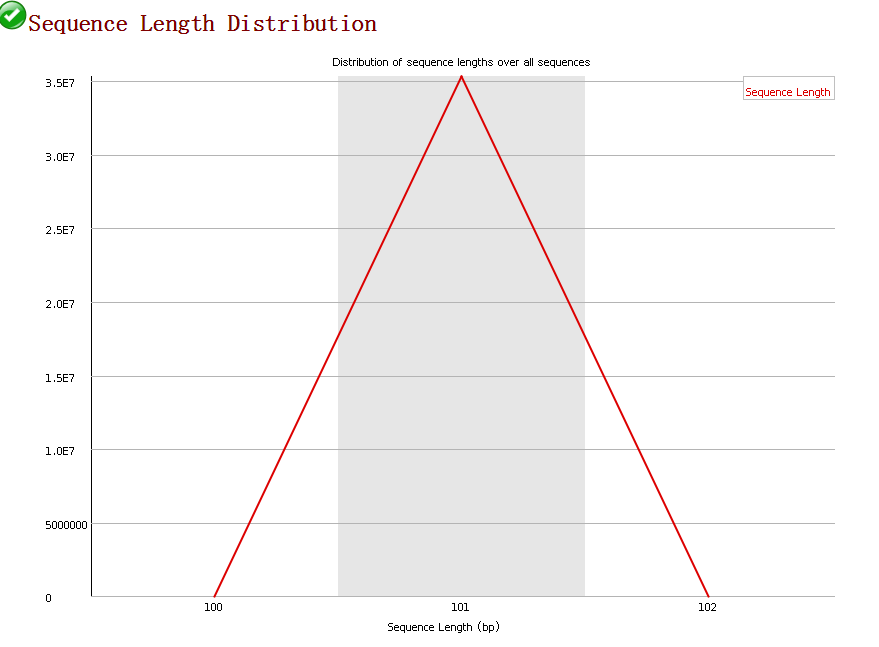
7.sequence length distribution reads的长度分布。当reads长度不一致时报“WRAN”,当有长度为0的read时报“FALL”.
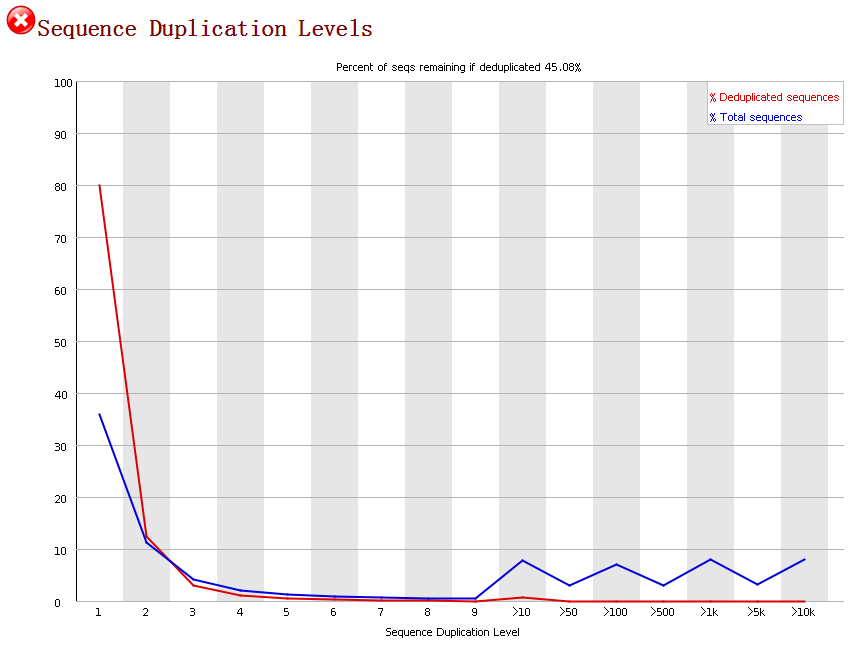
8.sequence duplication levels统计序列完全一样的reads的频率。测序深度越高,越容易产生一定程度的duplication,这是正常现象,但如果duplication的程度越高,就提示可能有bias的存在,横坐标是duplication的次数,纵坐标是duplicated reads 的数目,以unique reads 的总数作为100%。注,fastqc中用fastq数据的前200000条reads统计其在全部数据中的重复情况。当非unique reads占总数的比例大于20%时,报“WRAN”, 当非unique reads占总数的比例大于50%时,报“FALL”.
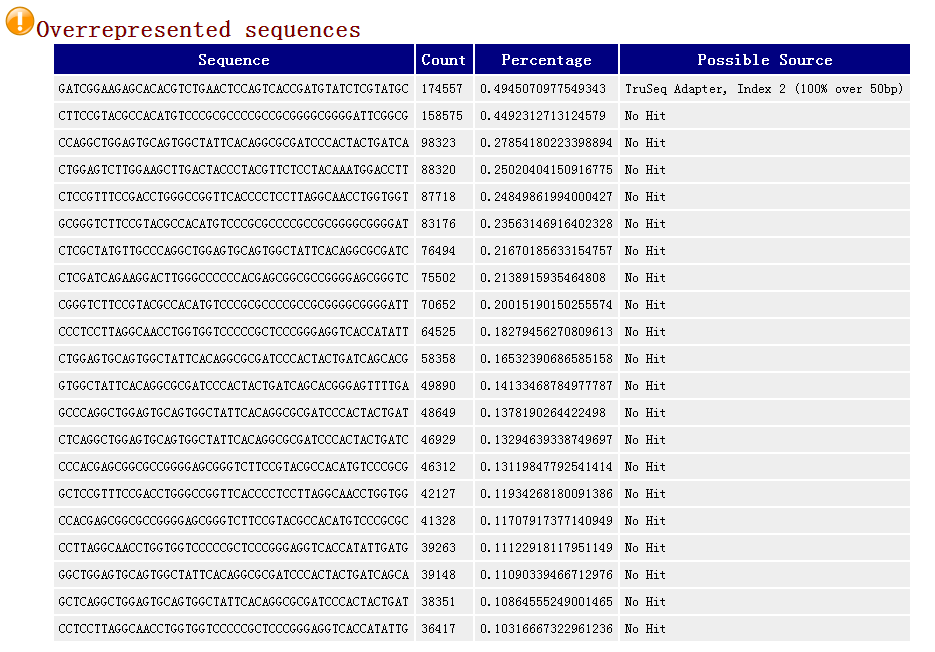
9. overrepresented sequence如果有某个序列大量出现,就叫做over-represented ,fastqc的标准是占全部reads的0.1%以上。和上面统计一样,取200000条数据统计,当发现超过总reads数0.1%的reads时报“WRAN”, 当发现超过总reads数1%的reads时报“FALL”.
10.adapter content:赎小编无能,没有找到相关介绍,欢迎各位留言给小编补习~在这里先谢过大家!

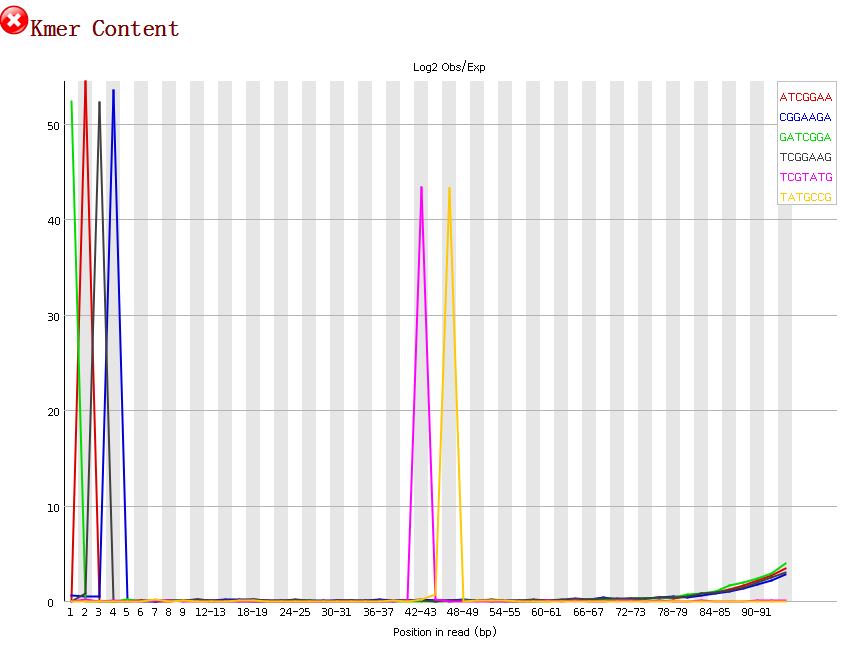
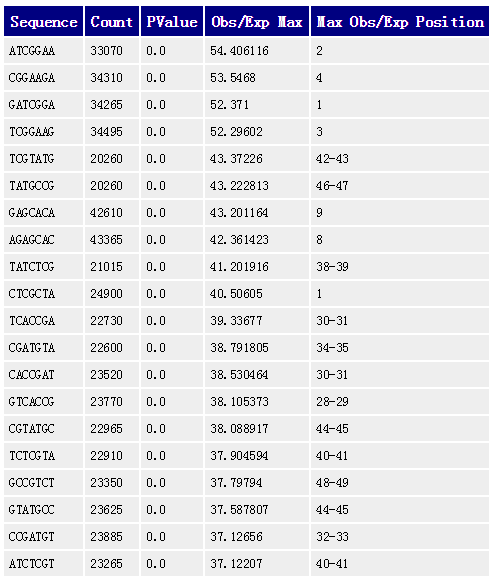
11.Kmer content 如果某k个bp的短序列在reads中大量出现,其频率高于统计期望的话,fastqc将其记为K-mer content。默认的k=5,可以用-k—kmer选项来调节,范围是2-10.fastqc除了列出所有overrepresented kmer,还会把前6个的per base distribution画出来。当有出现频率总体上3倍于期望或是在某位置上5倍于期望的kmer时,报“WRAN”, 当有出现频率总体上10倍于期望的kmer时,报“FALL”。
- 本文固定链接: https://www.maimengkong.com/kyjc/583.html
- 转载请注明: : 萌小白 2020年1月25日 于 卖萌控的博客 发表
- 百度已收录
