转眼间,十月已经到来,十月是一个充满希望的时节,是一个硕果累累的季节。在这么美好的时节里小锐有很多新技术、好消息与大家分享,接下来先向大家隆重介绍三代+二代宏基因组测序分析产品。

随着测序技术的不断更新,三代测序技术也越来越成熟,Nanopore测序读长更长,错误率更低,对生物信息相关领域的研究具有重大意义。近年来Nanopore测序的宏基因组高分文章已发表多篇,且已成功应用于微生物宏基因组研究领域。锐翌基因旨在为客户提供更优质的科研服务,特推出Nanopore三代+Illumina二代全新宏基因组测序分析服务产品。
基于Nanopore三代和Illumina二代的测序技术相结合,进行宏基因组研究,可以获得比纯二代更准确,更深入的数据挖掘结果。回顾二代宏基因组分析流程(如下图),大致可分为:序列质控,物种注释,序列组装,基因预测,基因集构建,功能数据库注释,菌株分析等。
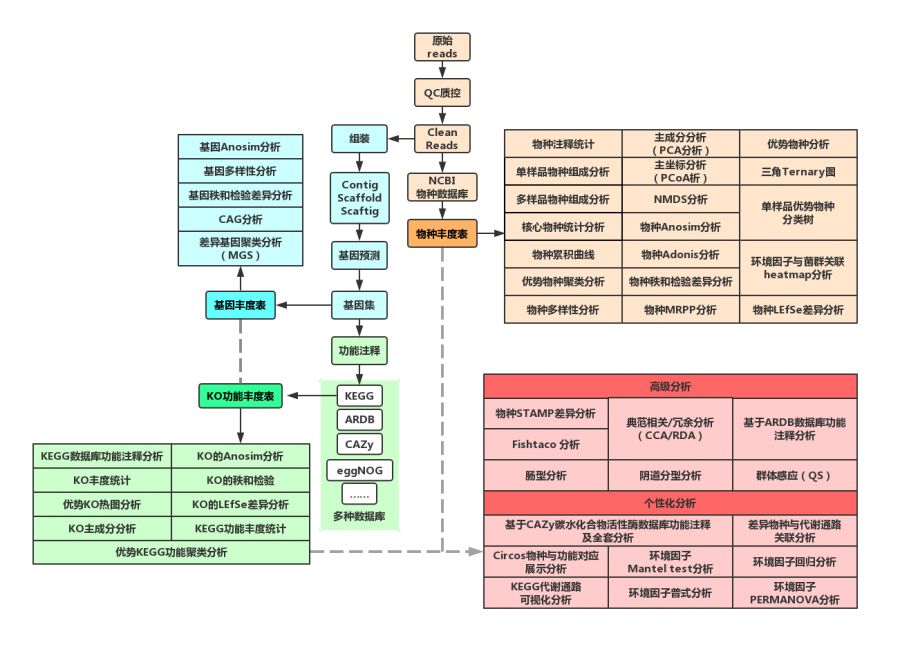
二代宏基因组分析流程图

在现有宏基因组测序分析产品基础上,结合三代测序技术,分析内容和结果都得到了极大的提升。
1
● 物种注释更准确、注释到的分类水平更低;
2
● 组装结果更理想,可组装出基因组近完成图,质粒完成图等,为深入探讨菌株、功能研究奠定坚实基础;
3
●增加了对菌株更加全面深入的数据挖掘,可深入剖析及探讨引起某种变化的具体机制。
1
产品优势

1
三代测序技术准确快速、超长读长、实时测序、随时终止,同时结合二代测序高质量碱基等优势,大大提高了宏基因组组装结果,比长读长更高的碱基准确度,比短读长更长的连续性;
2
长读长能提供丰富的信息,有助于denovo组装,获得稀有物种的高质量基因组近完成图和完整质粒序列;
3
较长的读长也有助于参考基因组mapping,获得更多的低丰度物种信息;
4
服务周期短,有竞争力的价格优势。
1
产品研究领域
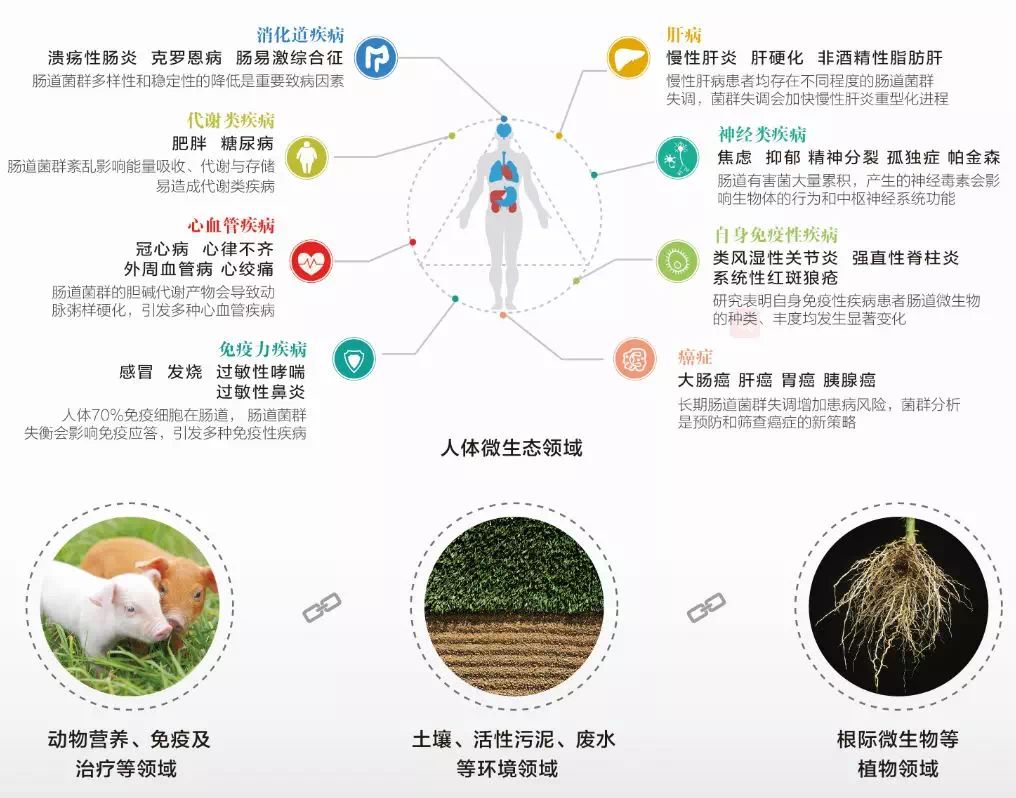
产品涉及人体各个部位微生物研究,同时广泛应用于土壤、水体、根际等多个领域。
1
技术流程

1
Nanopore测序+Illumina测序优势
1
二代测序碱基准确性高、测序通量大、成本低
2
三代测序长读长、直接测序、无需PCR扩增
3
二代、三代优势互补

Illumina测序

Nanopore测序
1
经典文献案例一
Nanopore metagenomics enables rapid clinical diagnosis of bacterial lower respiratory infection
Nature Biotechnology [IF:31.864]
DOI: https://doi.org/10.1038/s41587-019-0156-5
传统的细菌性下呼吸道感染(LRI)培养法诊断敏感性差,周期较长,不利于早期的靶向抗生素治疗,文章研究使用了三代测序技术,结合去除宿主DNA的试剂盒,可以快速诊断呼吸道感染。对病原检测的灵敏度为96.6%,特异性为41.7%,与特异性基因相结合,特异性和灵敏度都可增加至100%。
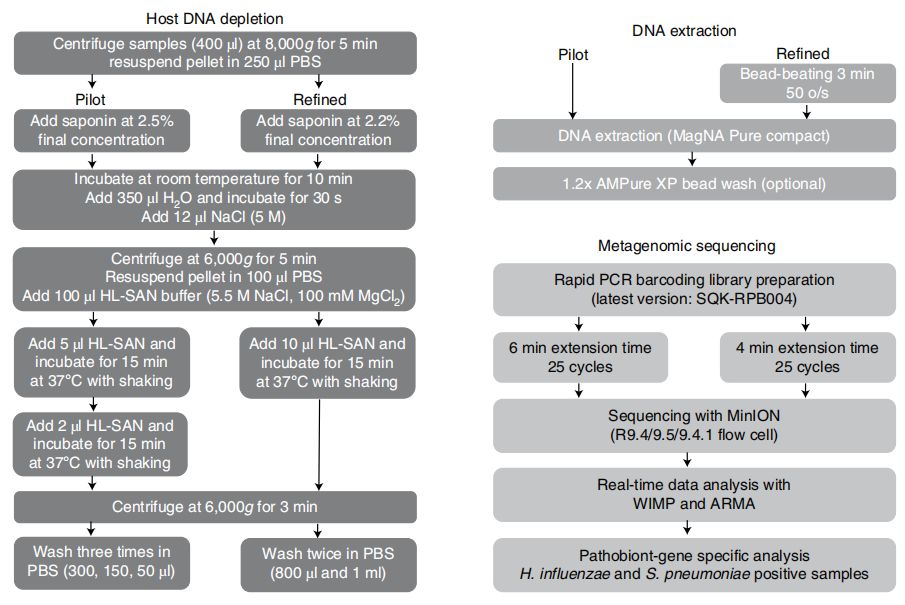
实时的纳米孔宏基因组测序6小时内准确鉴定病原菌
在研究测试的40个样品中有五个样品检测到了其他病原菌,相比于常规培养方法检测:M. catarrhalis(P8样中);Escherichia coli (P14样品中);H. influenzae(P22和P30 样品中);Klebsiella pneumoniae;M. catarrhalis(P29样品中)。
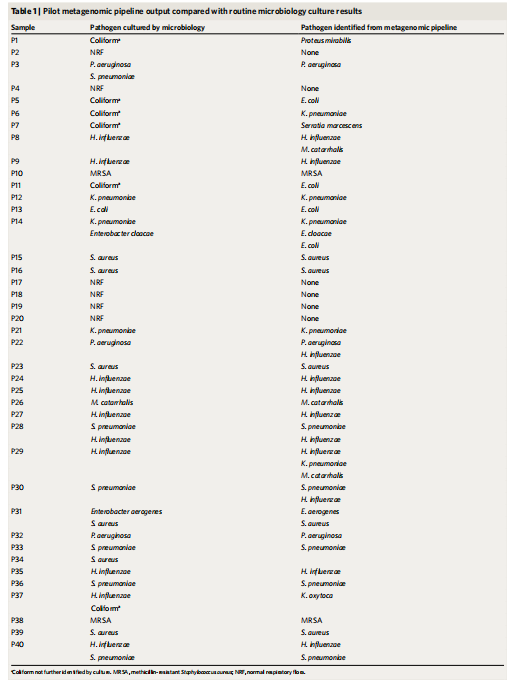
宏基因组测序与常规微生物培养结果比较表
1
经典文献案例二
Hybrid metagenomic assembly enables high-resolution analysis of resistance determinants and mobile elements in human microbiomes
Nature Biotechnology [IF:31.864]
DOI: https://doi.org/10.1038/s41587-019-0191-2
样本中微生物的耐药性基因一直是研究者关注重点,常见的耐药数据库有:ARDB、CARD、ARGs-OAP v1.0、ARGs-OAP v2.0、 ARG-ANNOT。为了更好的研究宏基因组的抗生素耐药基因,更好的对微生物质粒上携带的抗性元件,基因组上的抗性基因做研究。文章研发了OPERA-MS软件,用二、三代序列的进行混合组装,能组装出80个完整质粒或噬菌体序列,为肠道抗生素抗性精细研究提供依据。
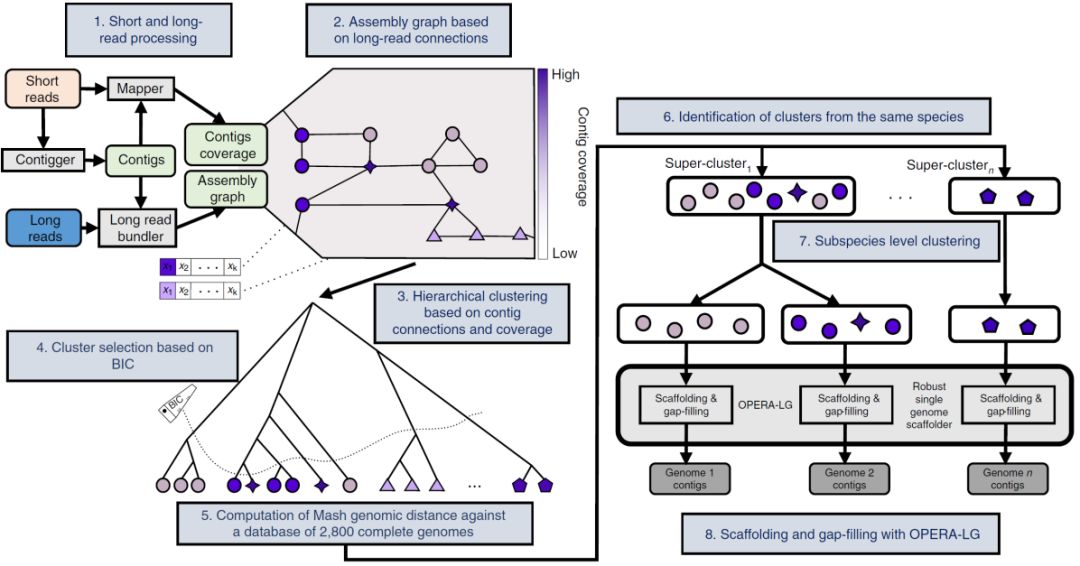
OPERA-MS工作流程图
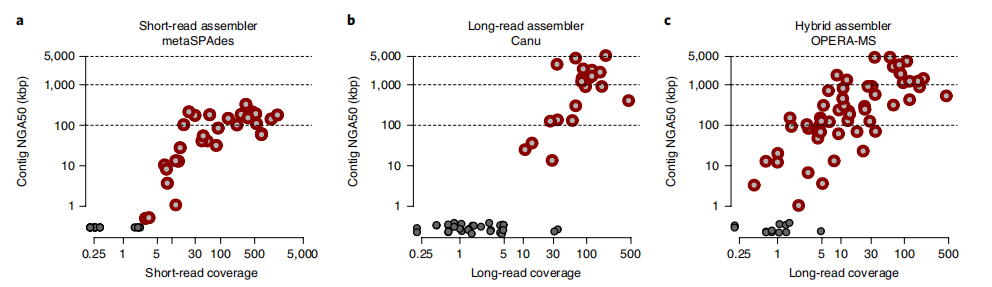
短读长代表性组装软件metaSPAdes(a),长读长组装软件Canu(b)和混合组装软件OPERA-MS(c),比较了三款软件的测序覆盖率增加与组装连续性统计图。研究结果表明混合组装在跨越覆盖方面有效改进了短读长和长读长的组装结果,可在低至9×长读长覆盖度下产生接近完整的基因组(NGA50>1 Mbp)。未组装的基因组显示为带有黑色边框的圆圈。
1
经典文献案例三
Compendium of 4,941 rumen metagenome assembled genomes for rumen microbiome biology and enzyme discovery
Nature biotechnology [IF:31.864]
DOI: 10.1038/s41587-019-0202-3
瘤胃微生物组在反刍动物消化植物物质中非常重要,但大多数微生物末被培养。文章使用283个牛瘤胃样本的二、三代测序数据(6.5T),组装得到4941个基因组(MAGs),同时鉴定了40多万个碳水化合物代谢相关的基因。
值得注意的是,由长读长数据组装而成的三个瘤胃细菌的全基因组,其中两个是以前未知的物种;使用文章的瘤胃物种基因组集合(4941个基因组),可以将测序序列的比对率从15%提高到50-70%。这些都助于更好地了解瘤胃微生物组的结构和功能。
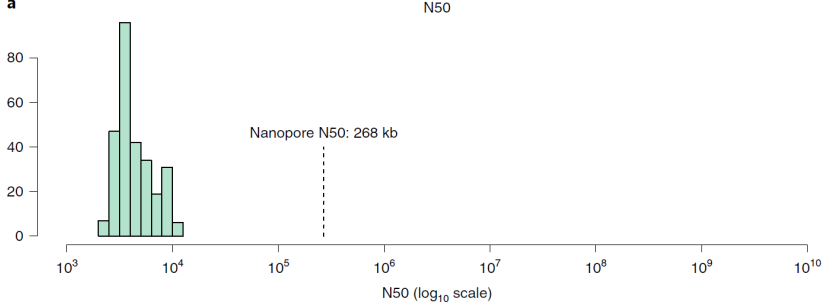
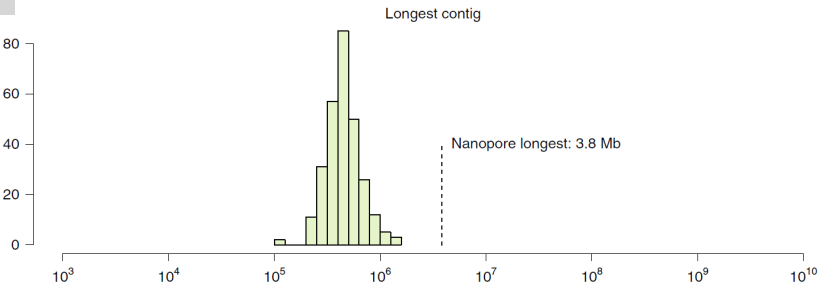
组装的统计结果分布直方图
图中显示了Nanopore组装远优于二代结果。Nanopore组装N50指标为268kb,比Illumina组装平均长度(4.7kb)长56倍以上;Nanopore组装产生最长contig是3.8 Mb,比Illumina平均长度(479 kb)长7倍,比ilumina最长contig的长1.74倍(1.38 Mb)。
参考文献
[1]Charalampous T, Kay GL, Richardson H, et al. Nanopore metagenomics enables rapid clinical diagnosis of bacterial lower respiratory infection. Nat Biotechnol 2019;37(7):783-92. doi:
10.1038/s41587-019-0156-5 [published Online First: 2019/06/27]
[2]Bertrand D, Shaw J, Kalathiyappan M, Ng AHQ, Kumar MS, Li C, Dvornicic M, Soldo JP, Koh JY, Tong C, Ng OT, Barkham T, Young B, Marimuthu K, Chng KR, Sikic M, Nagarajan N. Hybrid metagenomic assembly enables high-resolution analysis of resistance determinants and mobile elements in human microbiomes. Nat Biotechnol, 2019, 37(8): 937–944.
[3]Robert D. Stewart, et al. Compendium of 4,941 rumen metagenome assembled genomes for rumen microbiome biology and enzyme discovery.Nat Biotechnol. 2019,8;37(8):953-961
供稿:黄云
编辑:鲁淑妮
- 本文固定链接: https://maimengkong.com/zu/1986.html
- 转载请注明: : 萌小白 2025年7月26日 于 卖萌控的博客 发表
- 百度已收录
