基迪奥的GWAS系列课程已经上线3周了,上周收到一位学员(ID:可乐加冰啊)的学习心得。他学完GWAS系列课程后,用学到的技能完成了自己手里数据的分析。值得点赞的是,他把学习心得记录下来与大家分享,希望可以给学习GWAS的同学一个参考。以下是作者的原文(稍作修改):
随着测序技术的不断发展以及价格的不断降低,重测序越来越普及。下面给大家分享一下重测序数据的全基因组关联分析的分析流程。我将分几次给大家讲一下GWAS如何学习,表型处理,基因型处理,群体结构与亲缘关系计算,关联分析,结果筛选与展示。
首先,做分析需要各种软件。先给大家列举一下GWAS分析中所用的软件,vcftools(基因型文件过滤),bcftools(染色体文件合并等等),gcta(kinship计算),plink(vcf文件的格式转换以及过滤等),admixture(群体结构计算),tassel(用于GWAS分析),R(后续画图)。
那这些软件如何安装呢,当我还是个小白的时候(虽然我现在也白),我搜索admixture居然用的是360软件管家,emmm,现在想起来真是可怕! 为了避免大家多走弯路,今天以vcftools为例,给大家讲解如何安装GWAS所需要的软件。
首先 ,你需要掌握一些基本的Linux命令,比如cd pwdcp mkdir mv wget 你至少得会下载文件,建立目录吧。关于这方面的学习,建议看2本书,《Linux的鸟哥私房菜》和《Linux就该这么学》。这里不过多的给大家阐述Linux方面的知识。我只是想告诉大家,如果你想做重测序的GWAS分析,Linux是你必须掌握的,不要想着走捷径。
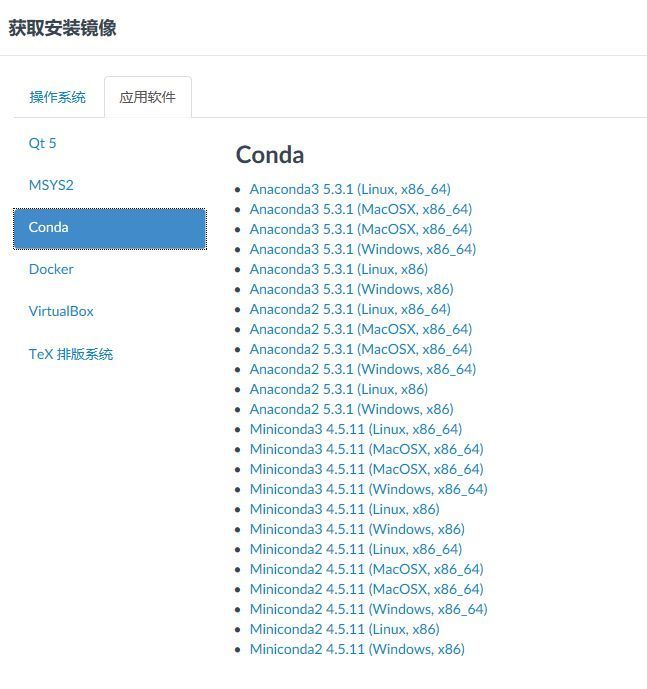
第二,给大家介绍一个服务器中的软件安装工具,conda,大家可以去清华镜像站去下载(https://mirrors.tuna.tsinghua.edu.cn/),如下图。

点击下载链接,选择Linux系统版本。

点击右边的应用软件选项卡,点击conda,右侧会出现很多下载链接,然后点右键,复制链接地址。这里由于我是在学校的服务器,所以我选择Minconda(Linux,x86)。
然后,wget链接地址,如下图:

下载之后启动conda:
Source ~/miniconda3/bin/activate

conda启动后在前面会有个(base)符号。
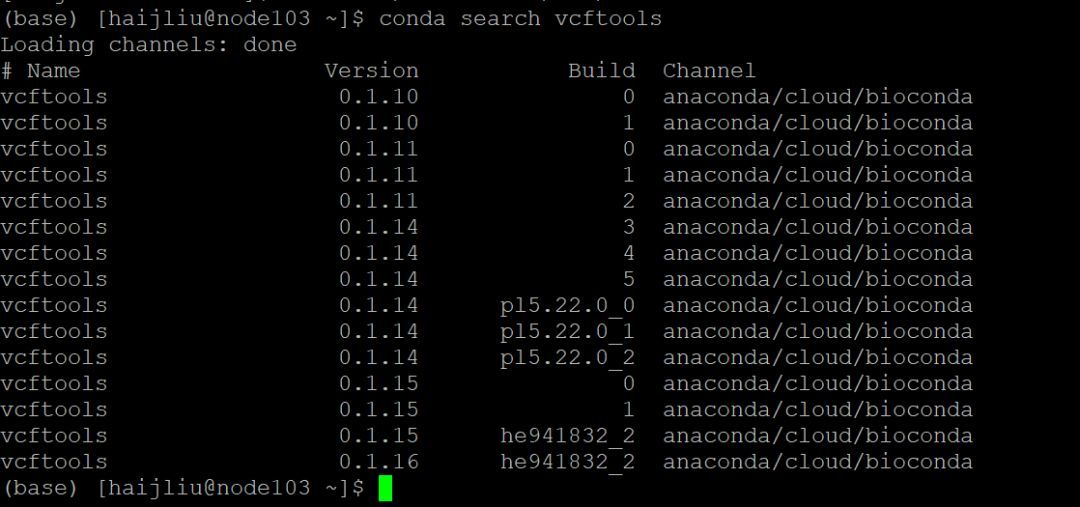
接下来就寻找软件,下图这些就是搜寻出来conda可以安装的vcftools版本。
Conda search vcftools
然后输入conda install vcftools就默认安装最新版本的vcftools了。

由于我服务器上已经安装好了vcftools这个软件了,我就不点确定了,你只需要按下enter这个键就可以啦。
安装之后,在服务器上输入vcftools,若出现版本信息,则这个软件安装成功,你就可以用vcftools进行vcf文件的过滤。
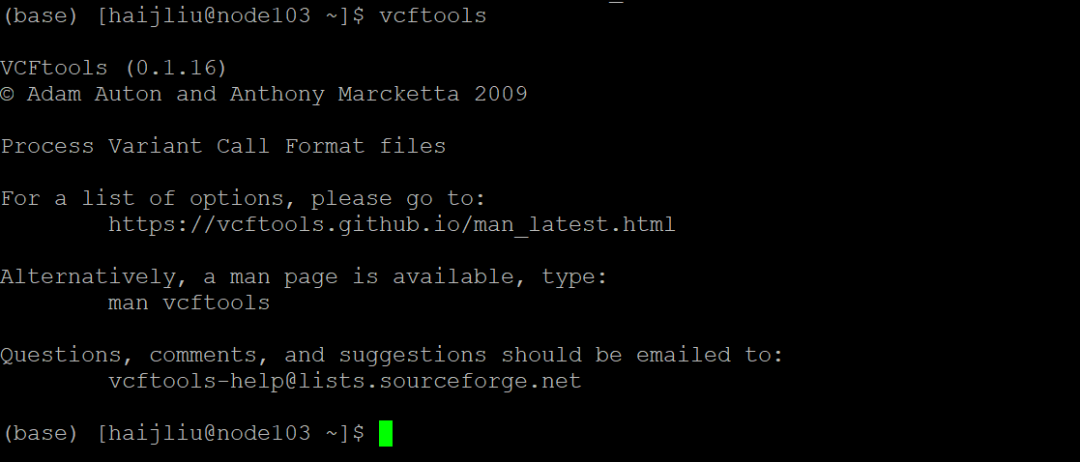
那安装完事之后,你只能在vcftools这个路径下运行vcftools这个软件,那如何在别的路径下运行vcftools,比如在你存放vcf文件的路径下运行vcftools?
不慌,我们把vcftools添加到环境变量中,我们先which vcftools看一下vcftools的可执行文件在哪个路径下。

我们可以发现vcftools在miniconda3/bin这个目录下,复制下这个目录
然后vim ~/.bashrc。
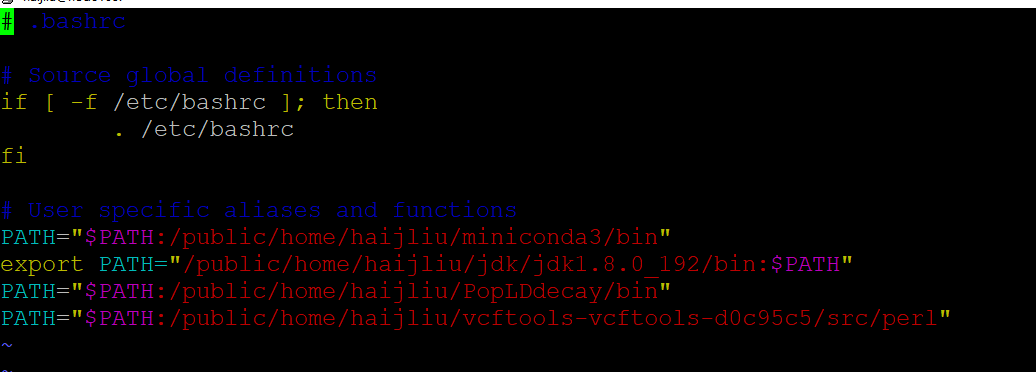
按住shift+g移动到最后一行,然后将vcftools的路径加入到环境变量中。
PATH="$PATH:/public/home/haijliu/miniconda3/bin"
好了,这样我们就可以在任何目录下都可以运行vcftools这个软件了。
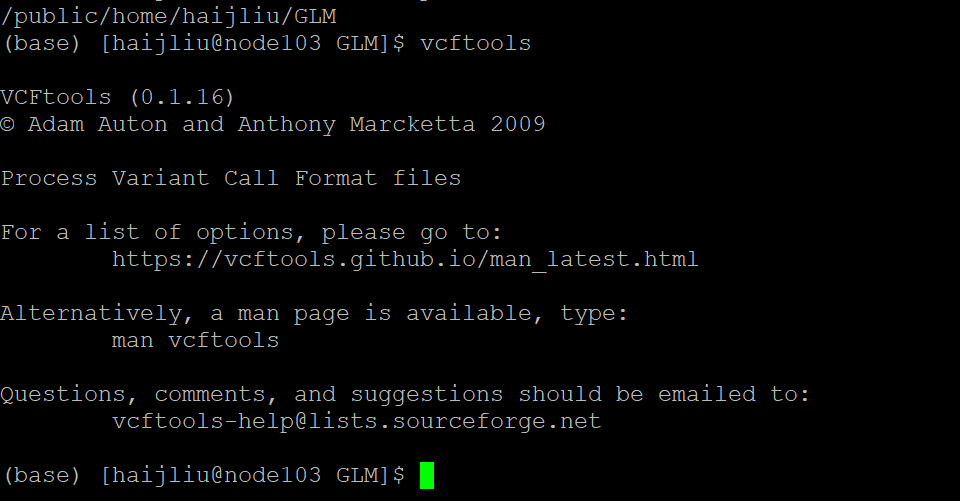
看,我在/public/home/haijliu/GLM这个路径下也可以运行啦~
本期的分享就到这里啦,下期给大家分享一下表型处理。更详细的GWAS学习课程,尽在基迪奥的Omicshare课堂,链接 :
http://www.omicshare.com/class/
转自:基迪奥
- 本文固定链接: https://maimengkong.com/zu/1424.html
- 转载请注明: : 萌小白 2023年4月11日 于 卖萌控的博客 发表
- 百度已收录
