把病理切片裂解制备成单细胞悬液,得到了“是什么”,却丢失了“在哪里”。如何兼得类型和位置信息呢?follow me 跟小编一起解开空间转录组技术的面纱吧。
在生命科学研究的道路上,历经了多组学联合覆盖“面“的,单细胞技术涉及“点“的研究历程,为了更加深入的对生命机制,疾病机理等研究,科学家不断发明新的技术,丰富新的维度,空间转录组技术也由此得到了很大的提升,操作上由繁至简,技术上由少至多。今天跟大家分享的便是10x Genomics Visium 系统。
初识
10x Genomics Visium
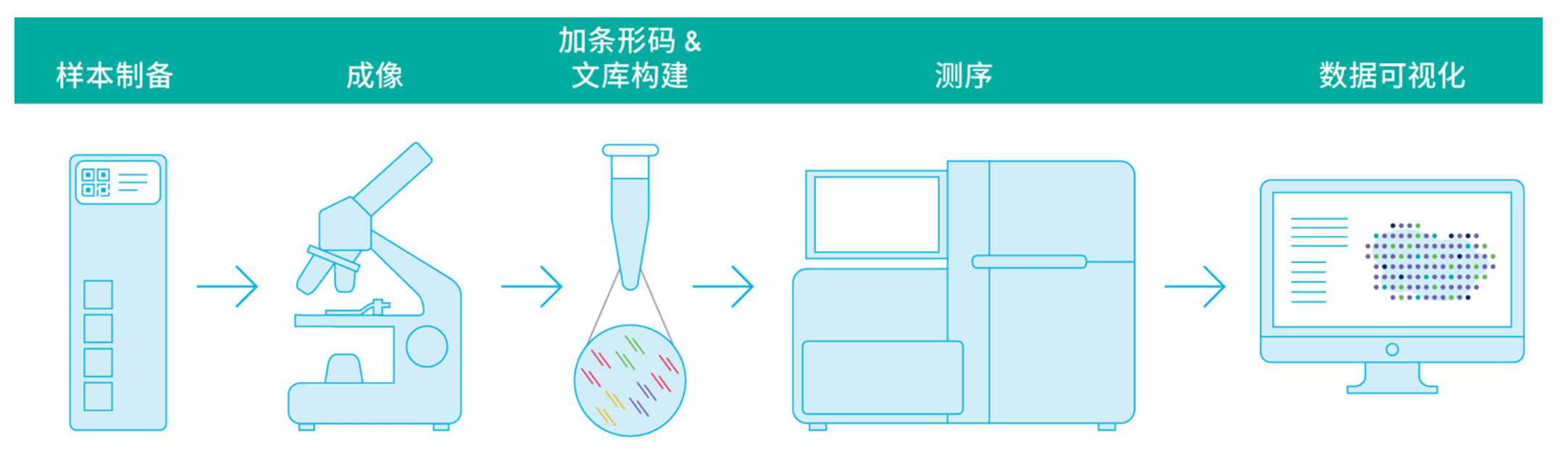
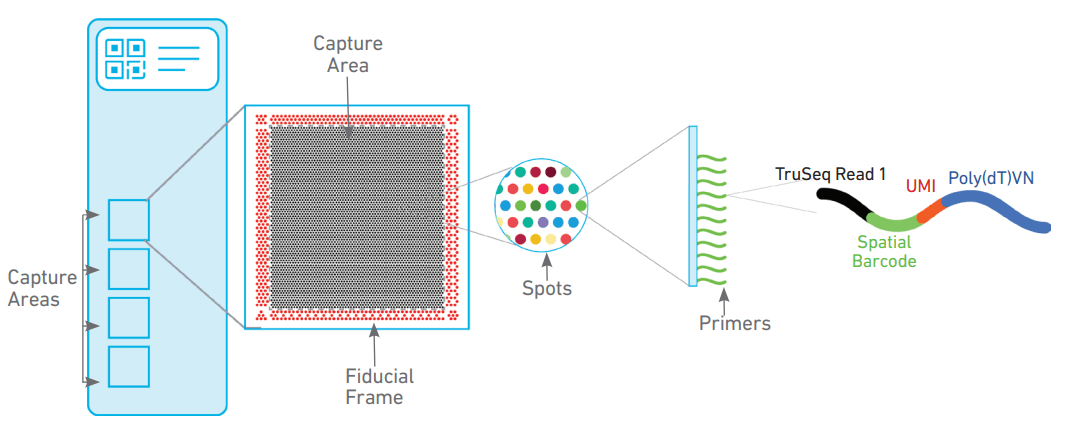
该系统可以划分为5个部分,样本制备,显微镜成像,文库构建,上机测序和数据分析。其中样本制备芯片是承载空间转录组信息捕获的核心技术,capture Area 为6.5 x 6.5 mm捕获点阵组成,每个点阵上包含有55μm直径,间距100μm的携带空间Barcodes和UMI,Poly(dT)的探针群组成。结构如下图所示:
01样本制备
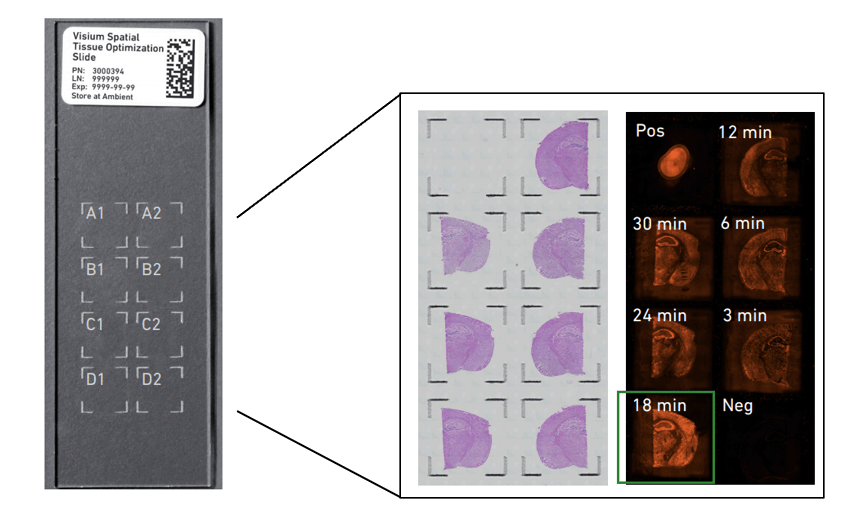
在样本制备之前,建议先进行组织优化,如下所示对待研究的组织进行透化时间摸索,一般根据研究的样本设置30分钟以内的时间梯度段,该示例为小鼠脑组织,透化时间根据显影选择18min。
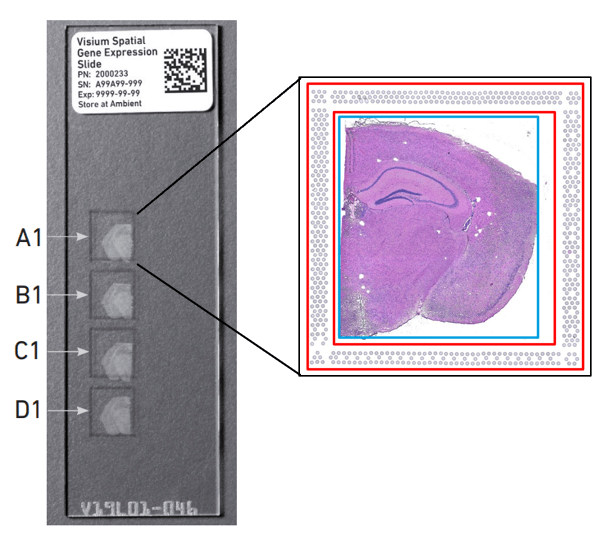
选择好透化的时间,就可以进行样本的制备了。注意组织切片不能覆盖芯片红色边框区域。
02文库构建
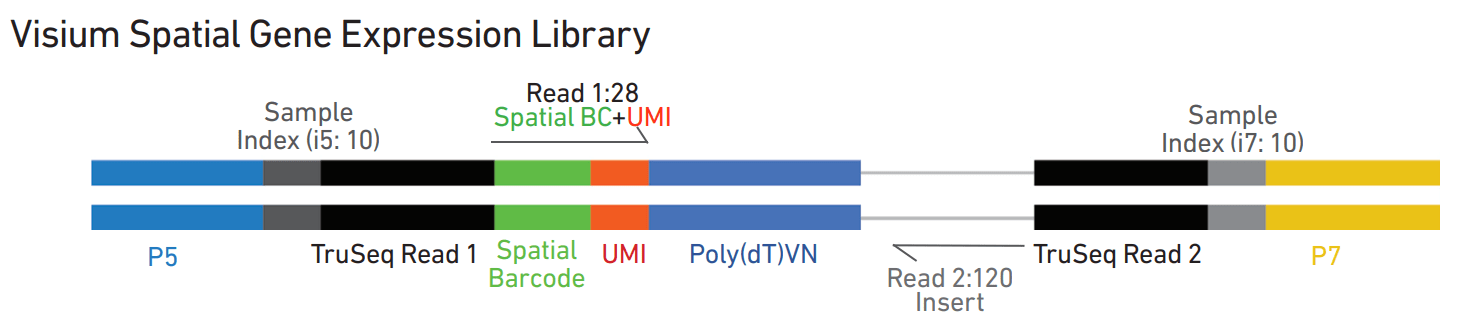
10x Genomics Visium 空间转录组文库结构如图所示:
03上机测序
10x Genomics Visium 系统对接大部分illumina测序平台,下机测序数据为fastq格式。由上述文库结构也可以得知,最后将获得包含空间barcode和UMI信息的28bp的Read1和捕获到的120bp的RNA目标序列Read2。
04数据分析
10x Genomics visium 提供了两款软件用以空间转录组数据的分析。一个是基于命令行进行图片和测序数据处理的软件SpaceRanger;一个是基于交互操作的可视化分析软件Loupe Browser。这两款软件都可以在其官网上获得,并且操作也比较简单。除此之外,也可以用seurat 单细胞分析的R软件包进行。
1. Spaceranger分析
整体分析流程如图所示:
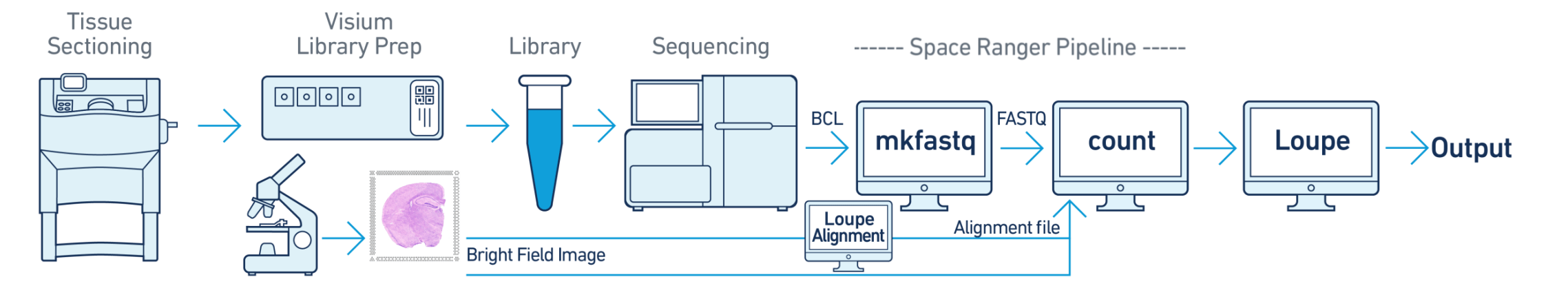
Space Ranger Pipeline 分步如下:
Step1:下载软件和参考数据库,自行在10x官网上下载;
Step2:解压软件和参考数据库文件;
tar -xzvf spaceranger-1.0.0.tar.gz
tar -xzvf refdata-cellranger-mm10-3.0.0.tar.gz
Step3:准备数据,执行命令行操作;
这里下载官网demo数据进行。
Space Ranger 包含以下功能,依次为fastq 文件转化,数据分析,文件格式转化,建库gtf文件转化,参考数据库构建,流程测试,上传日志和计算节点检查。mkfastq、mkref 和count 是常用的命令功能。
spaceranger mkfastq
spaceranger count
spaceranger mat2csv
spaceranger mkgtf
spaceranger mkref
spaceranger testrun
spaceranger upload
spaceranger sitecheck
spaceranger-1.0.0/bin/spaceranger count --id=分析的样本名称 --fastqs=待分析的fastq路径 --transcriptome=refdata-cellranger-mm10-3.0.0 --image=组织H&E图片(.jpg or .tiff格式 ) --unknown-slide(--slide=SLIDE 芯片的编号 --area=AREA组织所在的芯片区域) --sample=fastq文件前缀 --localcores=20 --localmem=128
Tips
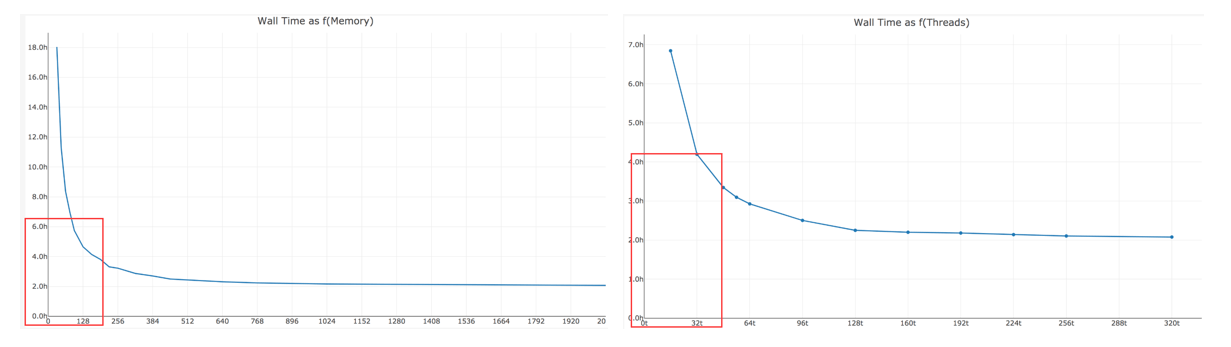
虽然空间芯片一共仅5000个Spots,但是程序需要位置信息的匹配等一系列操作,所以需要相当大的内存和线程。一般仅400M reads 数据,按照20个线程,128G内存,运行时间在4-6小时之间。
Step4:数据结果解读。

其中,
- analysis/文件夹包含了二次分析的结果文件,Spots 聚类信息;
- *.cloupe文件可以导入Loupe Browser进行可视化分析的;
- filtered_feature_bc_matrix 为空间Spots基因表达矩阵文件;
- Spatial/文件夹包含图像文件和图像位置信息文件;
- web summary.html 是空间转录组分析样本的基本信息文件,可以作为项目的质控文件(后期文章我们将会对空间转录组质控进行专一详细的介绍,敬请期待哦)。
2. Loupe Browser
Loupe Browser 可视化软件,操作个性化较强,也比较简单,这里就不做赘述了(后期文章我们将会对该软件进行专一详细的介绍,敬请期待哦)。这里需要了解的是它可以做组织H&E图片的矫正处理即可。
3. Seurat 分析
Seurat 是一款特别出色的单细胞分析R包,曾经推出了很多优秀的单细胞分析解决方案,在2019年年底推出了空间转录组分析的Seurat3.2版本。今天就和大家一起目睹下它的风采吧~
Step1:Seurat3.2安装;
在安装新版的seurat 之前,需要先安装R3.6版本
devtools::install_github("satijalab/seurat", ref = "spatial")
##也可以先行下载到本地进行安装install.packages("satijalab-seurat-v3.1.1-302-g1cb8a3d.tar.gz", repos = NULL, type = "source")
Step2:读取数据
##先导入需要的R包
library(Seurat)
library(ggplot2)
library(cowplot)
library(dplyr)
##官网直接导入的SeuratData,library(SeuratData)当然我们可以用前面Space Ranger运行的结果数据进行分析
brain<-Seurat::Load10X_Spatial("/path/out/") #读取数据的路径下应包含两个文件,表达矩阵文件和组织图片文件夹,即filtered_feature_bc_matrix.h5 和spatial/
Step3:数据展示
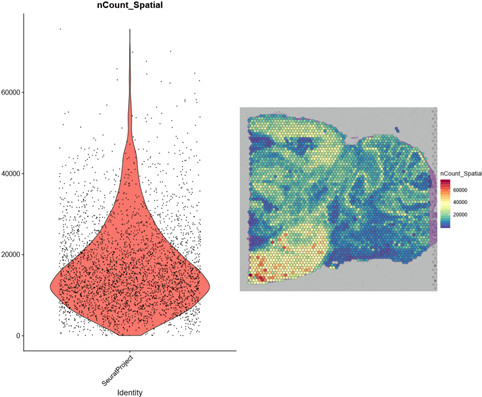
数据预处理,空间Spots表达水平
plot1 <- VlnPlot(brain, features = "nCount_Spatial", pt.size = 0.1) + NoLegend()
plot2 <- SpatialFeaturePlot(brain, features = "nCount_Spatial") + theme(legend.position = "right")
plot_grid(plot1, plot2)
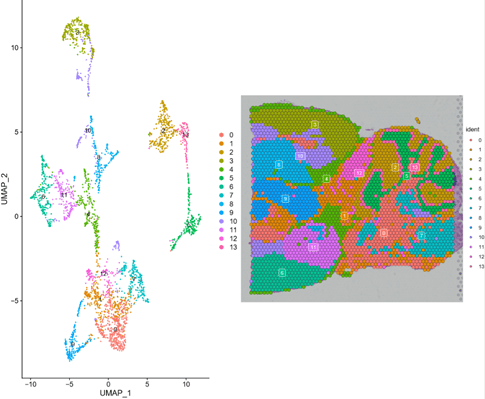
降维聚类分析
brain <- SCTransform(brain, assay = "Spatial", return.only.var.genes = FALSE, verbose = FALSE)##用SCT进行归一化
brain <- GroupCorrelation(brain, group.assay = "Spatial", assay = "SCT", slot = "scale.data", do.plot = FALSE)## 计算SCT noraml与表达水平的相关性
##降维,聚类
brain <- RunPCA(brain, assay = "SCT", verbose = FALSE)
brain <- FindNeighbors(brain, reduction = "pca", dims = 1:20)
brain <- FindClusters(brain, verbose = FALSE)
brain <- RunUMAP(brain, reduction = "pca", dims = 1:20
p1 <- DimPlot(brain, reduction = "umap", label = TRUE)
p2 <- SpatialDimPlot(brain, label = TRUE, label.size = 3)
plot_grid(p1, p2)
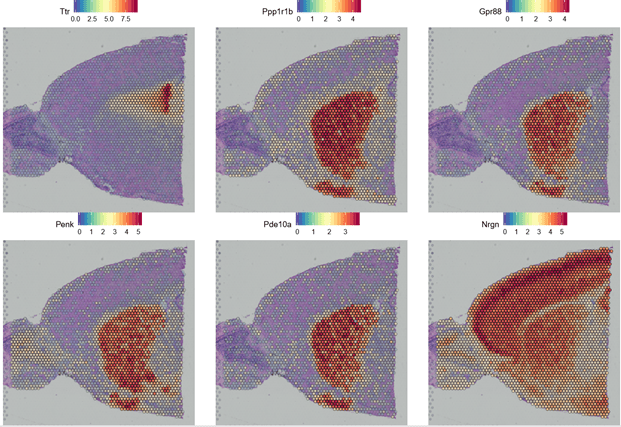
寻找marker基因
object.markers <- FindAllMarkers(object = brain, only.pos = FALSE, min.pct = 0.20, logfc.threshold = 0.25)
top <- object.markers %>% group_by(cluster) %>% top_n(1, avg_logFC) ##取每个cluster差异最显著的一个marker基因
VlnPlot(object = brain, features = top$gene, ncol = 2, pt.size = 0,idents=top$cluster)##对选取的marker基因进行小提琴图型展示
SpatialFeaturePlot(brain, features = top$gene, ncol = 3, pt.size.factor = 10)#marker基因高亮展示

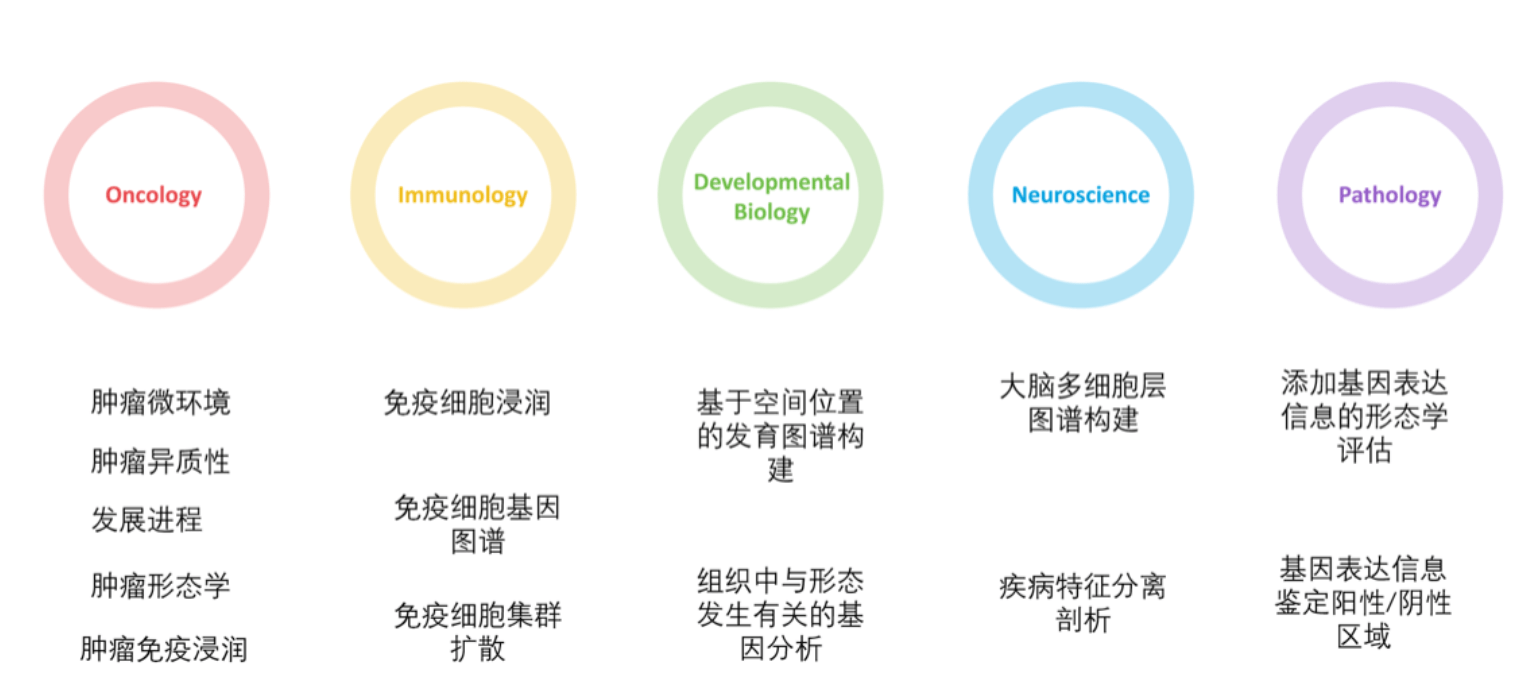
介绍到这里相信大家对空间转录组10x Genomics visium 分析流程和Seurat 包数据分析都有了一定的了解,那么这项技术主要应用在哪些方面呢?跟自己正研究或者待研究的课题有什么辅助指导意义呢?那就敬请关注我们下一讲吧,空间转录组应用研究思路。我们先预告一下应用领域,如下:
参考
https://support.10xgenomics.com/spatial-gene-expression
https://satijalab.org/seurat/vignettes.html- 本文固定链接: https://www.maimengkong.com/zu/1000.html
- 转载请注明: : 萌小白 2022年6月20日 于 卖萌控的博客 发表
- 百度已收录
