COG是Clusters of Orthologous Groups of proteins的缩写(http://www.ncbi.nlm.nih.gov/COG/)。COG是在对已完成基因组测序的物种的蛋白质序列进行相互比较的基础上构建的,COG数据库选取的物种包括各个主要的系统进化谱系。每个COG家族至少由来自3个系统进化谱系的物种的蛋白所组成,所以一个COG对应于一个古老的保守结构域。构成每个COG的蛋白被假定来自于同一个祖先蛋白。进行COG数据库比对可以对预测蛋白进行功能注释、归类以及蛋白进化分析。
eggNOG数据库(http://eggnog5.embl.de/#/app/home)是NCBI的COG数据库的扩展,它收集了更全面的物种和更大量的蛋白序列数据。它同样进行了同源基因聚类分析和对每个同源基因类的描述和功能分类。其中搜集了5090个生物(真核生物、代表性细菌和古菌)和2502个病毒的全基因组蛋白序列。将这些物种分成了379类(taxonomic levels)。每类的编号以NCBI的分类编号表示。包含4.4M个同源基因类(orthologous groups/OGs)。
1.对更全面的物种和更大量蛋白序列进行分类。相比于COG数据库纯人工且较为准确的分类,eggNOG数据库扩大物种和序列数据量,采用了非监督聚类方法进行计算。
2.对每个同源基因类进行了系统发育树构建、HMM模型构建、GO注释、KEGG Pathway注释、SMART/FPAM结构域注释、CAZyme注释等。
3.提供了本地化软件和网页工具进行eggNOG注释。
4.数据库内还包含KOG信息,其中原核生物的一般称为COG数据库;真核生物的一般称为KOG数据库。
eggNOG数据库首页
一
eggNOG数据库分类和功能概述
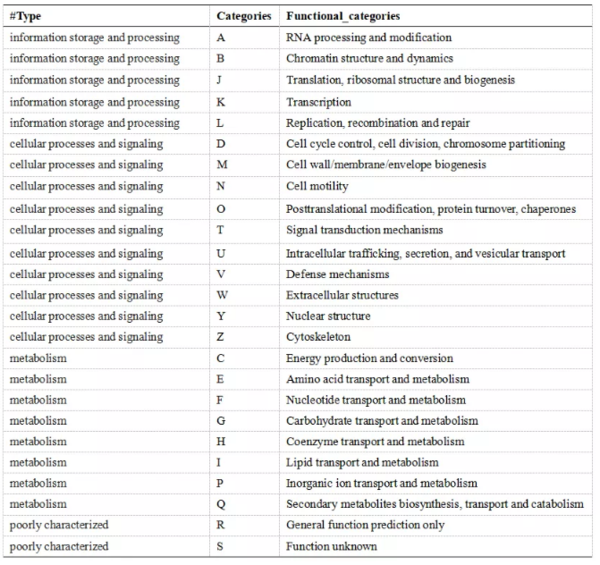
COG功能分成四大类,信息存储和处理(information storage and processing)、细胞过程和信号(cellular processes and signaling)、代谢(metabolism) 和缺失的功能描述(poorly characterized)。
每一类对应的信息如下:
二
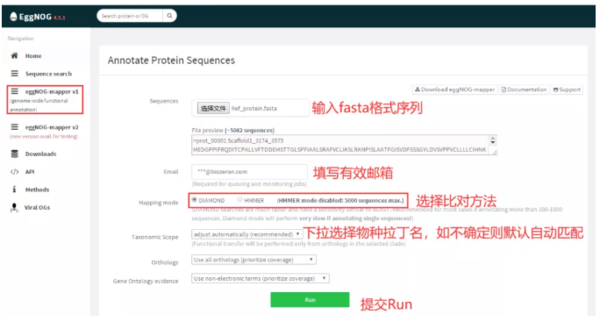
在线注释
/#/app/emapper)是COG在线注释工具,可使用预先计算的eggNOG簇和系统发育树,根据快速的直系同源比对进行功能注释。
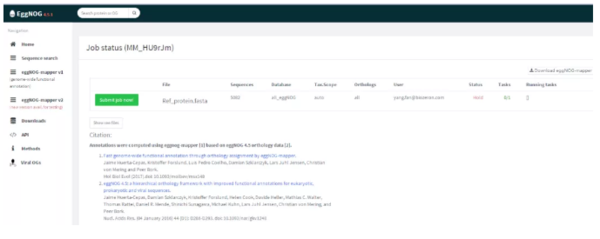
三
本地化配置
数据库
软件下载:https://github.com/eggnogdb/eggnog-mapper/archive/1.0.3.tar.gz
eggnog数据库(http://eggnogdb.embl.de/download/)
下载如下文件:
http://eggnogdb.embl.de/download/emapperdb-5.0.0/eggnog.db.gz
http://eggnogdb.embl.de/download/emapperdb-5.0.0/eggnog_proteins.dmnd.gz
软件
diamond:https://github.com/bbuchfink/diamond/
HMMER3:http://www.hmmer.org/download.html
sh Anaconda3-2019.10-Linux-x86_64.sh -b -p $HOME/anaconda3 修改环境变量
export PATH=$HOME/anaconda3/bin:$PATH 

conda install eggnog-mapper 

download_eggnog_data.py 

conda install -c bioconda diamond 

conda install -c bioconda hmmer 

emapper.py -i nuc.fa -o nuc -m diamond --cpu 64 --seed_ortholog_evalue 1e-5 --translate 核苷酸 emapper.py -i pep.fa -o pep -m diamond --cpu 64 --seed_ortholog_evalue 1e-5 --dmnd_db eggnog_proteins.dmnd
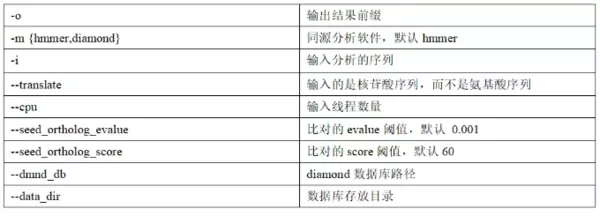
输出结果说明
pep.emapper.seed_orthologs比对结果列表:
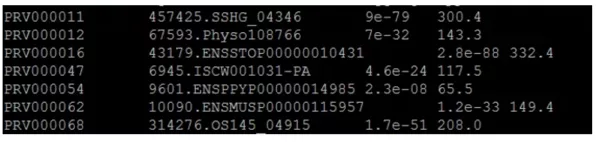
**列:输入基因id
第二列:eggNOG中的**蛋白质匹配
第三列:evalue
第四列:score值
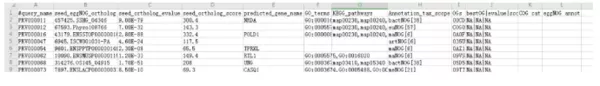
#表格说明:
seed_eggNOG_ortholog:eggNOG中的**蛋白质匹配
seed_ortholog_score:score值
GO_terms:GO功能信息
KEGG_pathways:KEGG功能信息
OGs:eggNOG直系同源群列表
eggNOG annot:功能描述
[1] Fast genome-wide functional annotation through orthology assignment by eggNOG-mapper.Mol Biol Evol,2017.(https://doi.org/10.1093/molbev/msx148)
[2] eggNOG 5.0: a hierarchical, functionally and phylogenetically annotated orthology resource based on 5090 organisms and 2502 viruses. Nucleic Acids Res, 2019.
(Doi: 10.1093/nar/gky1085)
- 本文固定链接: https://www.maimengkong.com/kyjc/917.html
- 转载请注明: : 萌小白 2022年5月12日 于 卖萌控的博客 发表
- 百度已收录
