01 启动子的基本类型
启动子是基因的重要组成部分,其主要功能为调控基因表达(转录)的起始时间和表达程度。但启动子本身并不控制基因活动,而是通过与转录因子的结合来控制基因活动。根据启动子对转录水平的调控程度,将其分为弱启动子和强启动子;此外,参照转录模式,其又可被分为:
(1)组成型启动子(Constitutive promoter),能够调控基因表达,使其基本恒定在一定程度上,从而使基因在不同部位或组织中的表达水平不存在明显差异;
(2)组织特异性启动子(Tissue specific promoter),其调控作用使得基因往往只在某些特定器官或组织中表达,且表现出发育调节的特性;
(3)诱导型启动子(Inducible promoter),即在某些特定的物理或化学信号刺激后,基因转录水平在该类启动子的调控下大幅度地提升。
02 启动子的特点
(1)通常含一些特定的元件,如TATA box;
(2)具有方向性,调控启动下游的基因表达;
(3)长度的不确定性,一般认为ATG上游2500bp的序列是该基因的启动子序列;
(4)一个基因通常含有多个启动子,启动不同转录本的表达;
(5)某些启动子具有表达的时空性和组织特异性。
03 启动子的研究思路
启动子功能及结构的研究,可以从以下几个方面着手:
(1)启动子结构研究,包括核心启动子区域、正调控区域、负调控区域以及增强子的确定;
(2)组织特异性启动子筛选及确定;
(3)转录因子与顺式作用元件相互作用研究;
(4)启动子甲基化作用研究;
(5)新顺式作用元件发现及功能鉴定。
04 研究路线
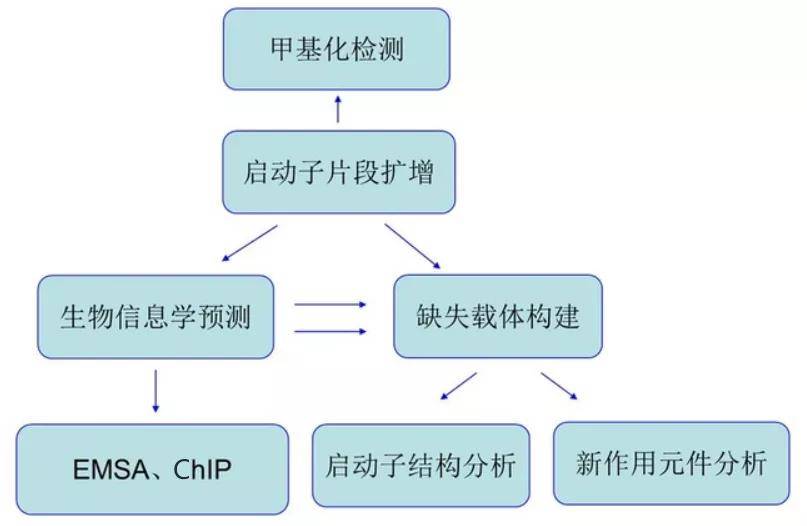
05 启动子研究的相关技术方法
5.1 启动子克隆
启动子克隆,一般分为两种类型:一种是已知启动子序列,可以直接采用基因克隆的方法进行实验;另一种是未知启动子序列,只知道转录组或部分编码序列,这个时候就可以采用特殊的的扩增方法——染色体步移进行未知序列的扩增。
染色体步移(Genome Walking)可以克隆已知序列的侧翼未知序列,基于PCR技术的染色体步移技术先后有十几种方法,目前最常使用的是TAIL-PCR(Thermal Asymmetric Interlaced PCR),即交错式热不对称PCR技术。利用特异性套嵌引物,结合简并引物钓取侧翼序列,其基本原理是利用目标序列附近的已知序列设计3个嵌套的特异引物(SP1,SP2,SP3),用它们分别和1个具有低Tm值的短的随机简并引物(AD)相结合,根据引物的长短和特异性的差异设计不对称的温度循环,通过分级反应来扩增特异产物。
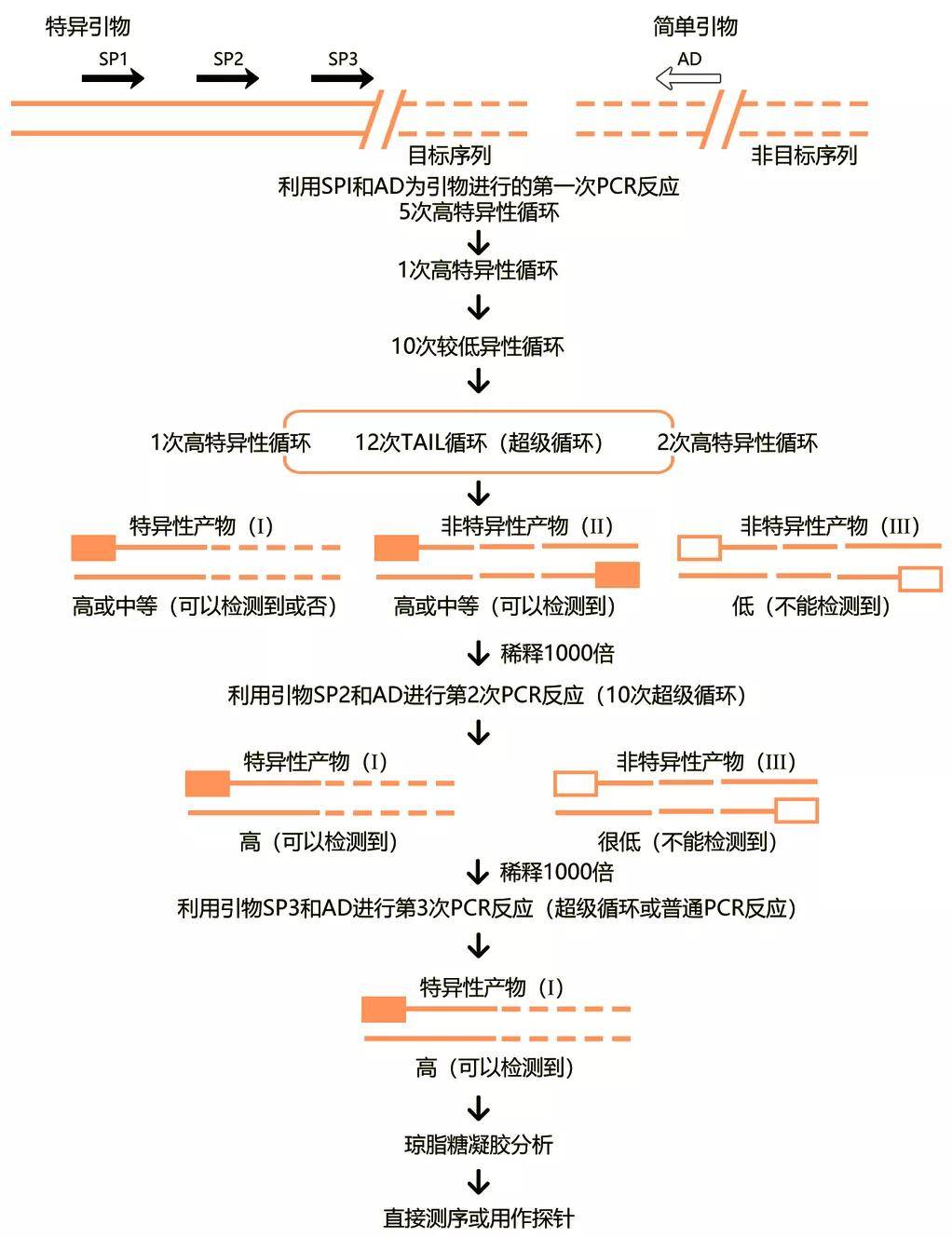
图1 TAIL-PCR原理示意图。
5.2 启动子活性分析
启动子的表达模式及强度只能通过报告基因来检测,目前在植物启动子的研究中常用的报告基因有β-葡萄糖苷酸酶基因(GUS)、绿色荧光蛋白基因(GFP)及其衍生出来的其他荧光蛋白、荧光素酶基因(LUC)等,其中最常用的是GUS报告基因。使用“启动子-报告基因-终止子”的载体构建方式,该方式适用的转化系统有原生质体转化系统、基因枪轰击或农杆菌介导的瞬时表达系统以及转基因植物的稳定表达系统等。将启动子与GUS报告基因相连进行转化,根据植物组织染色的结果可判断启动子表达的时空性和表达强度。
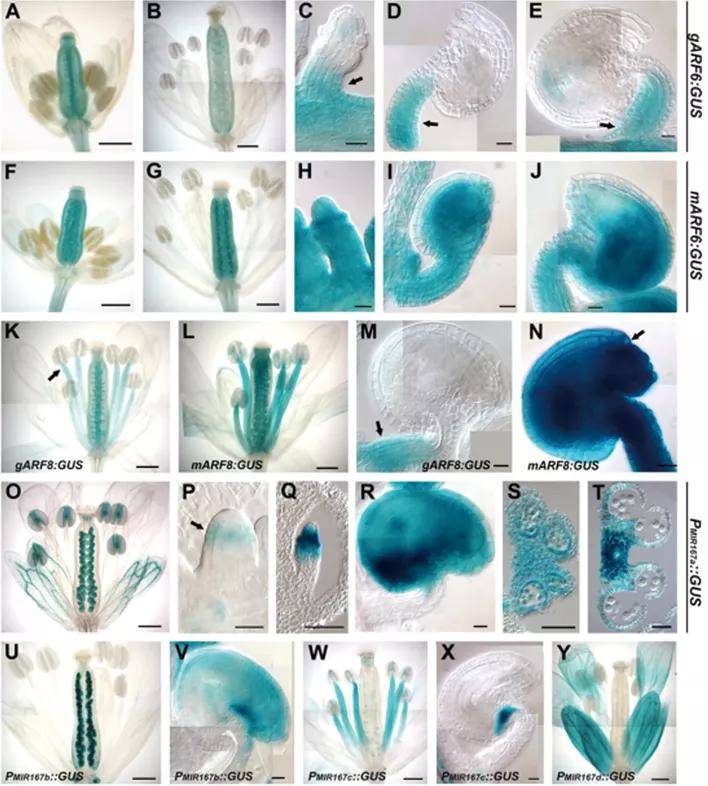
图2 GUS作为报告基因分析启动子在不同组织的表达模式(Qin et al., 2011)。
5.3 启动子核心元件分析
对于启动子,我们可以通过5’非翻译区一系列的缺失突变,构建启动子分析载体(如pCAMBIA1391,pBWA(v)H-gus载体),不断缩小范围,找到某一段对于该基因的启动子活性是必须的或者是最重要的序列。当找到这个比较核心的启动子序列后,可以通过一些在线的软件去预测其顺式作用元件的位点,每个在线软件都有自己独特的算法分析,得到的结果并不都是可靠的,每个软件都有自己的优势,需要多综合一些软件预测的结果,并通过分析预测出来的转录因子是不是和该基因有功能上的联系等做进一步的选择,继续做下面的验证实验。常见的启动子预测软件有:
(1)Promoter 2.0 Prediction Server http://www.cbs.dtu.dk/services/Promoter/
(2)Softberry预测TSSW、TSSP、TSSG、FPROM都是Softberry提供的启动子预测工具,http://www.softberry.com/berry.phtml?topic=tssp&group=programs&subgroup=promoter
(3)Plant CARE http://bioinformatics.psb.ugent.be/webtools/plantcare/html/
5.4 基因编辑技术
基因编辑技术主要是利用序列特异性核酸酶在特定基因位点产生DNA双链断裂,借助编辑受体自身的DNA修复系统在非同源末端连接(Non-homologous end joining, NHEJ)过程中产生随机的Indels(Small insertions and deletions)或在同源重组修复过程中插入或替换相应的基因片段,最终实现基因组序列的突变。
植物的启动子功能分析中,可以基于基因编辑技术,对启动子核心区域进行大片段删除,使启动子功能缺失,更好的研究启动子的核心功能。另外,还可以借助点编辑进行关键性元件的点突变,研究启动子元件的重要意义。

图3 gRNA指导的CRISPR/Cas9基因剪切系统。
5.5 EMSA
凝胶迁移实验(Electrophoretic Mobility Shift Assay, EMSA)是一种检测蛋白质和DNA序列相互结合的技术,可用于定性和定量分析,是转录因子研究的经典方法。反式作用因子一般指的就是转录因子,顺式作用元件一般是指转录因子和启动子结合的那部分序列,一般是10-20bp左右。点突变证实该顺式作用元件对于启动子活性是有影响的,接下来就要用EMSA实验验证反式作用因子和该顺式作用元件在体外是有直接结合的,反式作用因子可以用细胞核抽提物做,也可以用体外表达纯化的蛋白做或该反式作用因子的抗体做Super shift实验,利用这些实验可以证实顺式作用元件与反式作用因子的特异性结合。
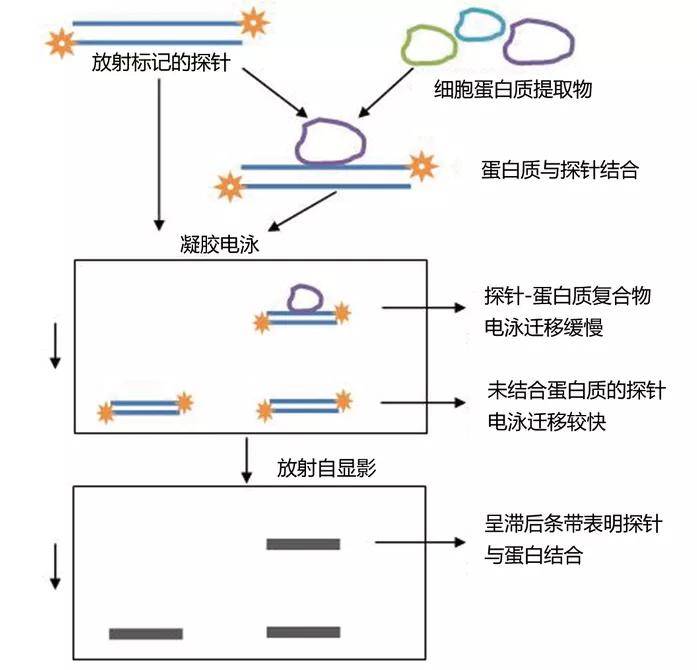
图4 EMSA基本原理示意图。
5.6 ChIP实验
EMSA的实验结果并不能说明反式作用因子和顺式作用元件有结合,毕竟体外的结合并不能代表生理条件下的结合。接下来就要用染色质免疫共沉淀(Chromatin immunoprecipitation assay,ChIP)技术来验证顺式作用元件和反式作用因子是否有结合。
ChIP是在生理条件下将细胞内的蛋白质与DNA进行交联并在活细胞状态下固定蛋白质DNA复合物,在超声波作用下将其随机切断成一定长度的染色质片段,然后沉淀该复合物,特异性富集目的蛋白结合的DNA片段,经过DNA纯化以及其鉴定,从而获得蛋白质和DNA相互作用的信息。
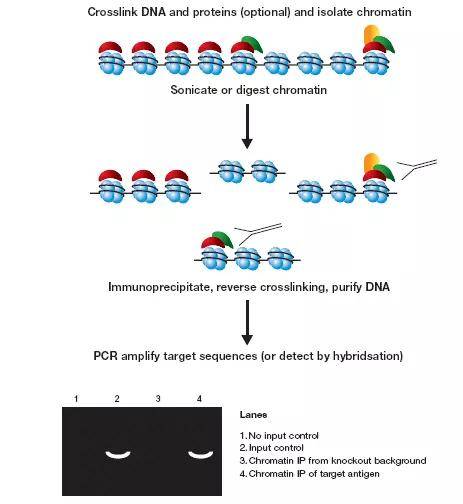
图5 ChIP原理图。
5.7 DNase I足迹法
DNase I足迹法是一种确定DNA结合蛋白在DNA分子上的结合位点的方法。先将含有特定顺式元件的双链DNA片段进行单链末端标记,然后加入适当浓度的DNase I,对DNA蛋白质复合物进行酶切,形成不同长度的DNA片段,最好保证相邻的DNA片段只相差一个核苷酸,片段经变性后用PAGE电泳分离,放射自显影,便可形成只有一个核苷酸差异的DNA梯形带。DNase I由于空间位阻不能直接绑定相邻的DNA结合蛋白,因此,足迹特征比较明显,一般比自身位点大8~10bp个碱基对。

图6 DNase I足迹法原理图(Song et al., 2015)。
5.8 双荧光报告系统
利用荧光素酶与底物结合发生化学发光反应的特性,把感兴趣的基因转录调控元件克隆在萤火虫荧光素酶基因的上/下游,构建成荧光素酶报告质粒,然后转染细胞,经适当刺激或处理后裂解细胞,测定荧光素酶活性。通过荧光素酶活性高低判断刺激前后或不同刺激对感兴趣的调控元件的影响。双荧光报告系统在启动子分析中有如下应用:
(1)启动子结构分析:将待测启动子区域序列进行分段截短,或对特定位点进行突变,再分别插入LUC/GUS报告基因上游,检测其启动子的活性。
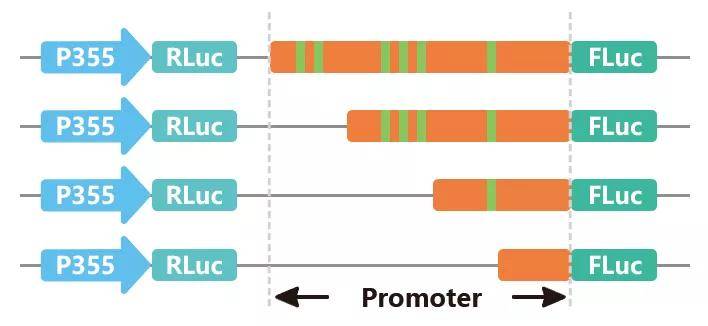
图7 启动子结构分析。
(2)启动子SNP分析:一些基因的启动子区域存在单核苷酸多态性,将待测启动子插入FLuc报告基因上游,检测萤光素酶活性,从而判断这些启动子的相对活性。
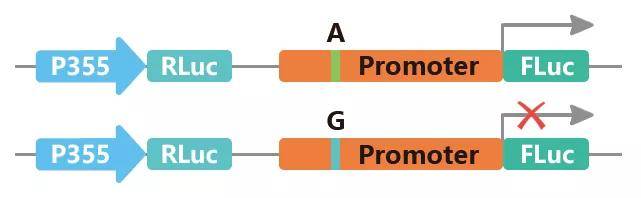
图8 启动子SNP分析。
(3)转录因子与启动子的互作分析:将该启动子序列插入LUC报告基因上游,同时在细胞中共转表达转录因子,分析转录因子是否调控萤光素酶的活性,从而判断转录因子对启动子的调控作用。
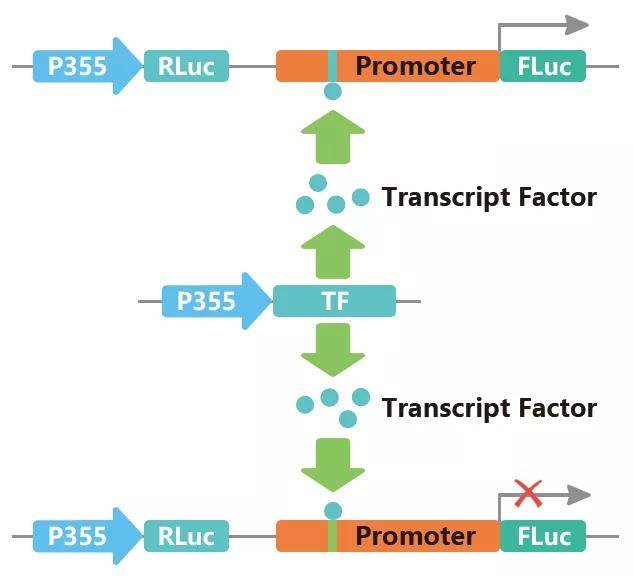
图9 启动子与转录因子的互作分析。
5.9 启动子区域甲基化
DNA甲基化是一个生物过程,可在DNA分子中引入甲基化基团,其不会改变DNA序列本身,而只是改变DNA片段的活性。据报道,在几乎所有已分析过的生物中,启动子区域的甲基化水平均与基因表达负相关,且具有转录活性基因的CpG密集启动子从未被甲基化,但是,转录沉默基因并不一定带有甲基化的启动子。值得注意的是,尽管CpG岛的DNA甲基化与转录抑制作用明确相关,但对CG缺乏的启动子中DNA甲基化的功能仍不清楚。研究显示,DNA甲基化可能以两种方式影响基因转录:其一为DNA本身的甲基化可能在物理上阻碍转录因子与基因的结合;其二为甲基化的DNA可能被称为甲基CpG结合域(methyl-CpG-binding domain,MBD)的蛋白结合,促使MBD蛋白将其他蛋白募集到位点,例如组蛋白脱乙酰基酶和其他可以修饰组蛋白的染色质重塑因子,从而形成致密的、无活性的异染色质。

图10 BSP甲基化检测结果(Date from labreal. net)。
5.10 植物转基因技术
植物转基因技术是指把从动物、植物、微生物中分离到的目的基因或者人工设计合成的核酸片段,通过多种方法转移到植物的基因组中,使之稳定遗传。植物启动子研究过程中往往需要进行稳定的遗传转化,实验中需要将构建的启动子分析载体,借助于农杆菌或者基因枪,稳定转入植物之中,获得转基因苗,然后对其进行进一步的实验分析,(PS:我司目前可提供的常规转化物种有:水稻、烟草、油菜、番茄、拟南芥、棉花、大豆、玉米、杨树、马铃薯、苜蓿和黄瓜等)。
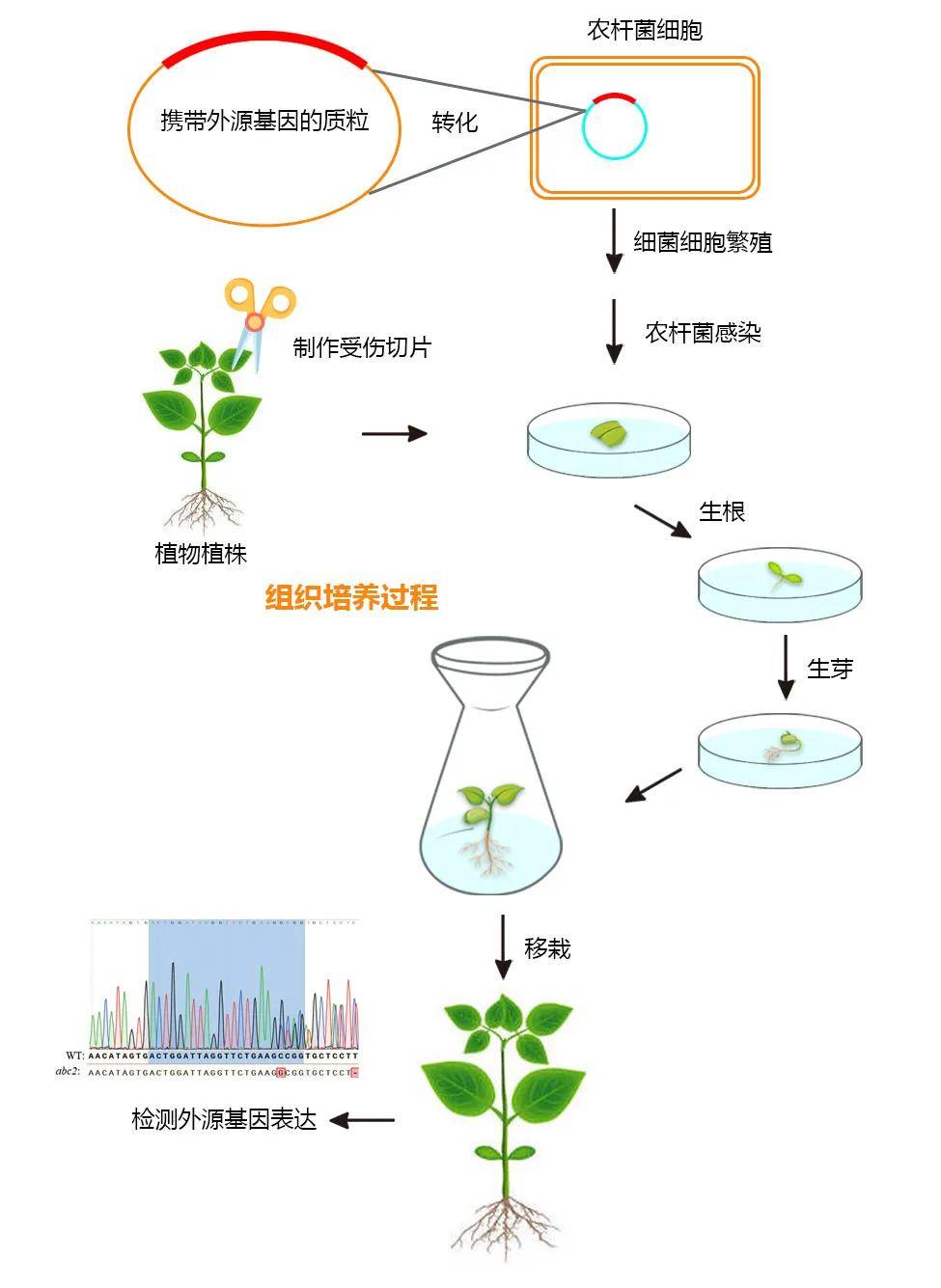
图11 农杆菌介导的植物遗传转化流程。
REFERENCES
Qin BX, Tang D, Huang J, et al. Rice OsGL1-1 is involved in leaf cuticular wax and cuticle membrane. Mol Plant, 2011, 4(6): 985-95.
Song C, Zhang S, Huang H. Choosing a suitable method for the identification of replication origins in microbial genomes. Front Microbiol, 2015, 6: 1049.- 本文固定链接: https://www.maimengkong.com/kyjc/1435.html
- 转载请注明: : 萌小白 2023年4月15日 于 卖萌控的博客 发表
- 百度已收录

网友评论(1)